
Chapitre 02. Statistiques descriptives et visualisation des données univariables.
Tout le code avec R.
F. Bertrand et M. Maumy
2025-09-22
Source:vignettes/CodeChap02.Rmd
CodeChap02.Rmd

|

|
Sources des données
Statistique Canada
Données libres d’utilisation, commerciale ou non, sur Statistique Canada. https://www.statcan.gc.ca/fra/reference/droit-auteur
répartition en classes d’âge de la population du Canada en 2020. Statistique Canada. Tableau 17-10-0005-01 Estimations de la population au 1er juillet, par âge et sexe DOI : https://doi.org/10.25318/1710000501-fra
population des dix provinces et trois territoires du Canada au quatrième trimestre 2020. Source : Statistique Canada. Tableau 17-10-0009-01. Estimations de la population, trimestrielles. DOI : https://doi.org/10.25318/1710000901-fra
Statistique de l’OCDE.
- Taux d’emploi en % de la classe d’age
Taux d’emploi par groupe d’âge (indicateur). OCDE (2021). doi: 10.1787/b01db125-fr (Consulté le 11 février 2021)
Le taux d’emploi d’une classe d’âge se mesure en fonction du nombre des actifs occupés d’un âge donné rapporté à l’effectif total de cette classe d’âge. Les actifs occupés sont les personnes de 15 ans et plus qui, durant la semaine de référence, déclarent avoir effectué un travail rémunéré pendant une heure au moins ou avoir occupé un emploi dont elles étaient absentes. Les taux d’emploi sont présentés pour quatre classes d’âge : les personnes âgées de 15 à 64 ans (personnes en âge de travailler); les personnes âgées de 15 à 24 ans sont celles qui font leur entrée sur le marché du travail à l’issue de leur scolarité, les personnes âgées de 25 à 54 ans sont celles qui sont au plus fort de leur activité professionnelle, et les personnes âgées de 55 à 64 ans sont celles qui ont dépassé le pic de leur carrière professionnelle et approchent de l’âge de la retraite. Cet indicateur est désaisonnalisé et est mesuré en pourcentage de l’effectif total de la classe d’âge.
- Par secteur dans les pays de l’OCDE en 2020-Q3.
Emploi par activité (indicateur). OCDE (2021). doi: 10.1787/6b2fff89-fr (Consulté le 11 février 2021)
- Emploi par niveau d’études en % des 25-64 ans
Emploi par niveau d’études (indicateur). OCDE (2021) doi: 10.1787/6e3d44f3-fr (Consulté le 11 février 2021)
Cet indicateur fournit les taux d’emploi selon le niveau d’études : premier cycle du second degré, deuxième cycle du second degré, supérieur. Le taux d’emploi est le pourcentage d’actifs occupés dans la population en âge de travailler. Les actifs occupés sont les personnes qui travaillent au moins une heure par semaine en tant que salarié ou à titre lucratif, ou qui ont un emploi mais sont temporairement absentes de leur travail pour maladie, congé ou conflit social. Cet indicateur donne le pourcentage des actifs occupés âgés de 25 à 64 ans dans la population des individus âgés de 25 à 64 ans.
- Part du revenu national total équivalent en Euro en 2019. Répartitition du revenu par quantiles - enquêtes EU-SILC et PCM (ILC_DI01).
INSEE
- Ménages par taille du ménage,
MEN4 - Ménages par taille du ménage, sexe et âge de la personne de référence en 2017. France métropolitaine. Insee.
Données Covid officielles
Répartition par région française du nombre de personnes hospitalisées et atteintes du Covid 19 le 21 février 2021.
Répartition par région française du nombre de personne en réanimation et atteintes du Covid 19 le 21 février 2021.
if(!("sageR" %in% installed.packages())){install.packages("sageR")}
library(sageR)Effectifs
Nombre de personnes dans un foyer Tableau 2.5
data(Personnes_Foyer)
xi <- Personnes_Foyer$xi
ni <- Personnes_Foyer$niDispersion
sqrt(weighted.mean((xi-weighted.mean(xi,ni))^2,ni))
#> [1] 1.246994
sqrt(weighted.mean((xi-weighted.mean(xi,ni))^2,ni))/weighted.mean(xi,ni)*100
#> [1] 56.89321
rrr=rep(xi, ni)
mean(rrr)
#> [1] 2.191815
quantile(rrr)
#> 0% 25% 50% 75% 100%
#> 1 1 2 3 6
median(abs(rrr-median(rrr)))
#> [1] 1
mad(rrr,constant = 1)
#> [1] 1Population classes d’age Canada
data(AgevsProter_Canada_full)
Age <- margin.table(as.matrix(AgevsProter_Canada_full),1)
Age
#> 0 à 4 ans 5 à 9 ans 10 à 14 ans 15 à 19 ans 20 à 24 ans
#> 1921944 2044603 2072100 2100865 2482802
#> 25 à 29 ans 30 à 34 ans 35 à 39 ans 40 à 44 ans 45 à 49 ans
#> 2645240 2661723 2630680 2464247 2390116
#> 50 à 54 ans 55 à 59 ans 60 à 64 ans 65 à 69 ans 70 à 74 ans
#> 2449915 2744896 2560241 2167275 1786622
#> 75 à 79 ans 80 à 84 ans 85 à 89 ans 90 à 94 ans 95 à 99 ans
#> 1218303 811370 517710 248593 74476
#> 100 ans et plus
#> 11517
cumsum(Age)
#> 0 à 4 ans 5 à 9 ans 10 à 14 ans 15 à 19 ans 20 à 24 ans
#> 1921944 3966547 6038647 8139512 10622314
#> 25 à 29 ans 30 à 34 ans 35 à 39 ans 40 à 44 ans 45 à 49 ans
#> 13267554 15929277 18559957 21024204 23414320
#> 50 à 54 ans 55 à 59 ans 60 à 64 ans 65 à 69 ans 70 à 74 ans
#> 25864235 28609131 31169372 33336647 35123269
#> 75 à 79 ans 80 à 84 ans 85 à 89 ans 90 à 94 ans 95 à 99 ans
#> 36341572 37152942 37670652 37919245 37993721
#> 100 ans et plus
#> 38005238
Age/sum(Age)
#> 0 à 4 ans 5 à 9 ans 10 à 14 ans 15 à 19 ans 20 à 24 ans
#> 0.0505705029 0.0537979265 0.0545214320 0.0552783014 0.0653278898
#> 25 à 29 ans 30 à 34 ans 35 à 39 ans 40 à 44 ans 45 à 49 ans
#> 0.0696019849 0.0700356882 0.0692188798 0.0648396676 0.0628891207
#> 50 à 54 ans 55 à 59 ans 60 à 64 ans 65 à 69 ans 70 à 74 ans
#> 0.0644625617 0.0722241497 0.0673654774 0.0570256921 0.0470098885
#> 75 à 79 ans 80 à 84 ans 85 à 89 ans 90 à 94 ans 95 à 99 ans
#> 0.0320561866 0.0213488993 0.0136220697 0.0065410194 0.0019596246
#> 100 ans et plus
#> 0.0003030372
cumsum(Age)/sum(Age)
#> 0 à 4 ans 5 à 9 ans 10 à 14 ans 15 à 19 ans 20 à 24 ans
#> 0.0505705 0.1043684 0.1588899 0.2141682 0.2794961
#> 25 à 29 ans 30 à 34 ans 35 à 39 ans 40 à 44 ans 45 à 49 ans
#> 0.3490980 0.4191337 0.4883526 0.5531923 0.6160814
#> 50 à 54 ans 55 à 59 ans 60 à 64 ans 65 à 69 ans 70 à 74 ans
#> 0.6805440 0.7527681 0.8201336 0.8771593 0.9241692
#> 75 à 79 ans 80 à 84 ans 85 à 89 ans 90 à 94 ans 95 à 99 ans
#> 0.9562253 0.9775742 0.9911963 0.9977373 0.9996970
#> 100 ans et plus
#> 1.0000000
njall <- Age
nj <- njall[-length(njall)]
centres <- c(5*(0:19)+2.5)
cj <- 5*(0:20)
x <- actuar::grouped.data(Group = cj, Frequency = as.numeric(nj))
class(x)
#> [1] "grouped.data" "data.frame"Position
mean(x)
#> Frequency
#> 41.42872
quantile(x)
#> 0% 25% 50% 75% 100%
#> 0.00000 22.73666 40.88648 59.79263 100.00000
sum(nj*centres)/sum(nj)
#> [1] 41.42872
print(cumsum(nj/sum(nj)),digits=3)
#> 0 à 4 ans 5 à 9 ans 10 à 14 ans 15 à 19 ans 20 à 24 ans 25 à 29 ans
#> 0.0506 0.1044 0.1589 0.2142 0.2796 0.3492
#> 30 à 34 ans 35 à 39 ans 40 à 44 ans 45 à 49 ans 50 à 54 ans 55 à 59 ans
#> 0.4193 0.4885 0.5534 0.6163 0.6808 0.7530
#> 60 à 64 ans 65 à 69 ans 70 à 74 ans 75 à 79 ans 80 à 84 ans 85 à 89 ans
#> 0.8204 0.8774 0.9244 0.9565 0.9779 0.9915
#> 90 à 94 ans 95 à 99 ans
#> 0.9980 1.0000
centres
#> [1] 2.5 7.5 12.5 17.5 22.5 27.5 32.5 37.5 42.5 47.5 52.5 57.5 62.5 67.5 72.5
#> [16] 77.5 82.5 87.5 92.5 97.5
40+(45-40)*(.5-0.4885)/(0.5534-0.4885)
#> [1] 40.88598
40.88598
#> [1] 40.88598
20+(25-20)*(0.2500-0.2142)/(0.2796-0.2142)
#> [1] 22.737
22.74
#> [1] 22.74
55 + (60-55)*(0.75-0.6808)/(0.7530-0.6808)
#> [1] 59.79224
59.79224
#> [1] 59.79224Dispersion
59.79-22.74
#> [1] 37.05
#Centres
(x[,1][-1]+x[,1][-length(x[,1])])/2
#> [1] 2.5 7.5 12.5 17.5 22.5 27.5 32.5 37.5 42.5 47.5 52.5 57.5 62.5 67.5 72.5
#> [16] 77.5 82.5 87.5 92.5 97.5
#sd
sqrt(weighted.mean(((x[,1][-1]+x[,1][-length(x[,1])])/2-mean(x))^2,x[,2])-1/12*weighted.mean(diff(x[,1])^2,x[,2]))
#> [1] 23.02927
#CV%
sqrt(weighted.mean(((x[,1][-1]+x[,1][-length(x[,1])])/2-mean(x))^2,x[,2])-1/12*weighted.mean(diff(x[,1])^2,x[,2]))/mean(x)*100
#> Frequency
#> 55.58769Concentration
Lorentz
if(!("ggplot2" %in% installed.packages())){install.packages("ggplot2")}
library(ggplot2)
if(!("gglorenz" %in% installed.packages())){install.packages("gglorenz")}
library(gglorenz)Jeu de données d’exemple du package gglorenz
ggplot(billionaires, aes(TNW)) +
stat_lorenz()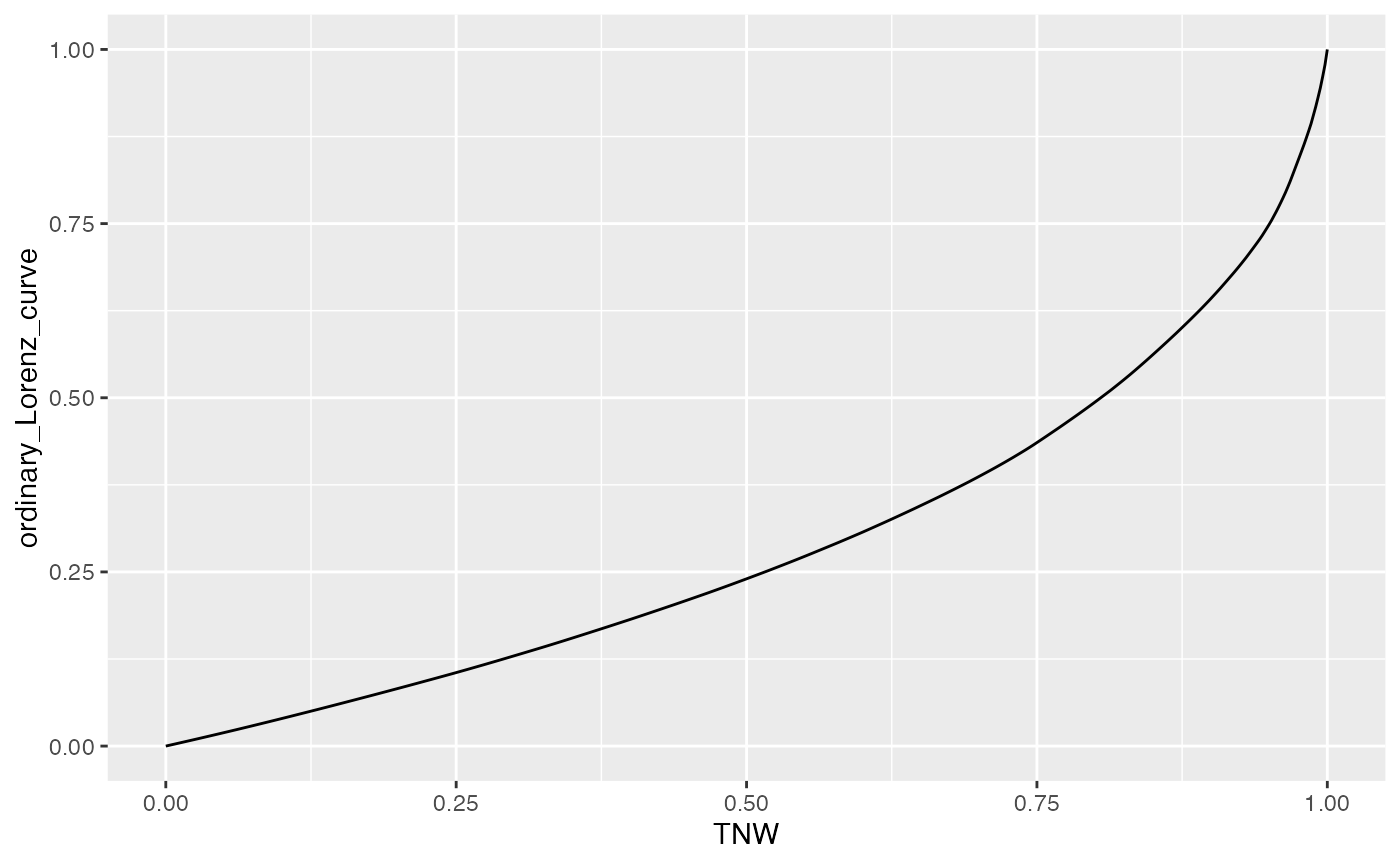
ggplot(billionaires, aes(TNW)) +
stat_lorenz(desc = TRUE) +
coord_fixed() +
geom_abline(linetype = "dashed") +
theme_minimal()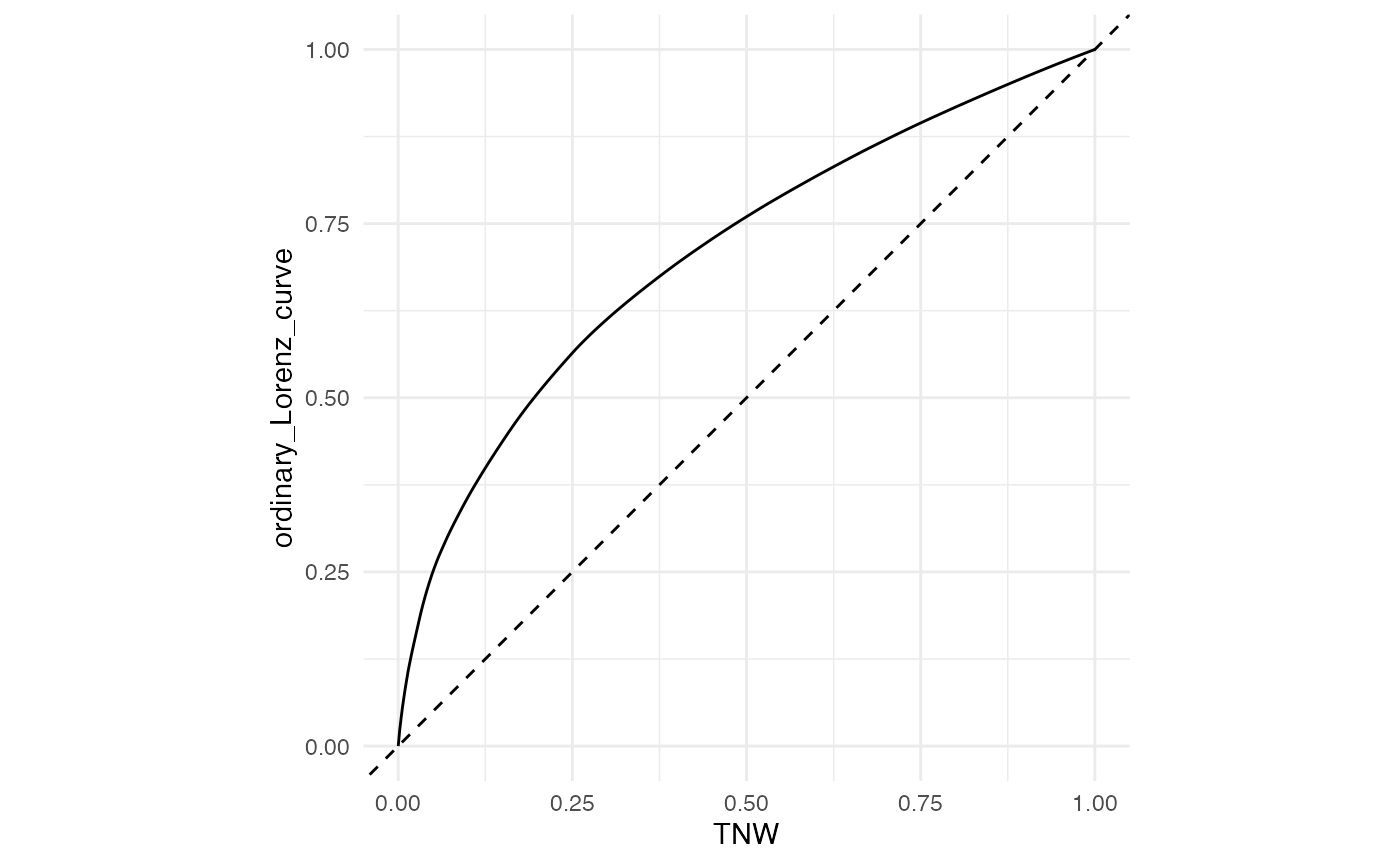
Répartition de la richesse dans les pays de l’OCDE
data(Richesse)
if(!("ggplot2" %in% installed.packages())){install.packages("ggplot2")}
library(ggplot2)
ggplot(Richesse, aes(Belgique)) +
stat_lorenz() +
coord_fixed() +
geom_abline(linetype = "dashed") +
theme_minimal() +
ylab("Courbe de Lorenz ordinaire") +
annotate_ineq(Richesse$Belgique,
measure_ineq = "Gini")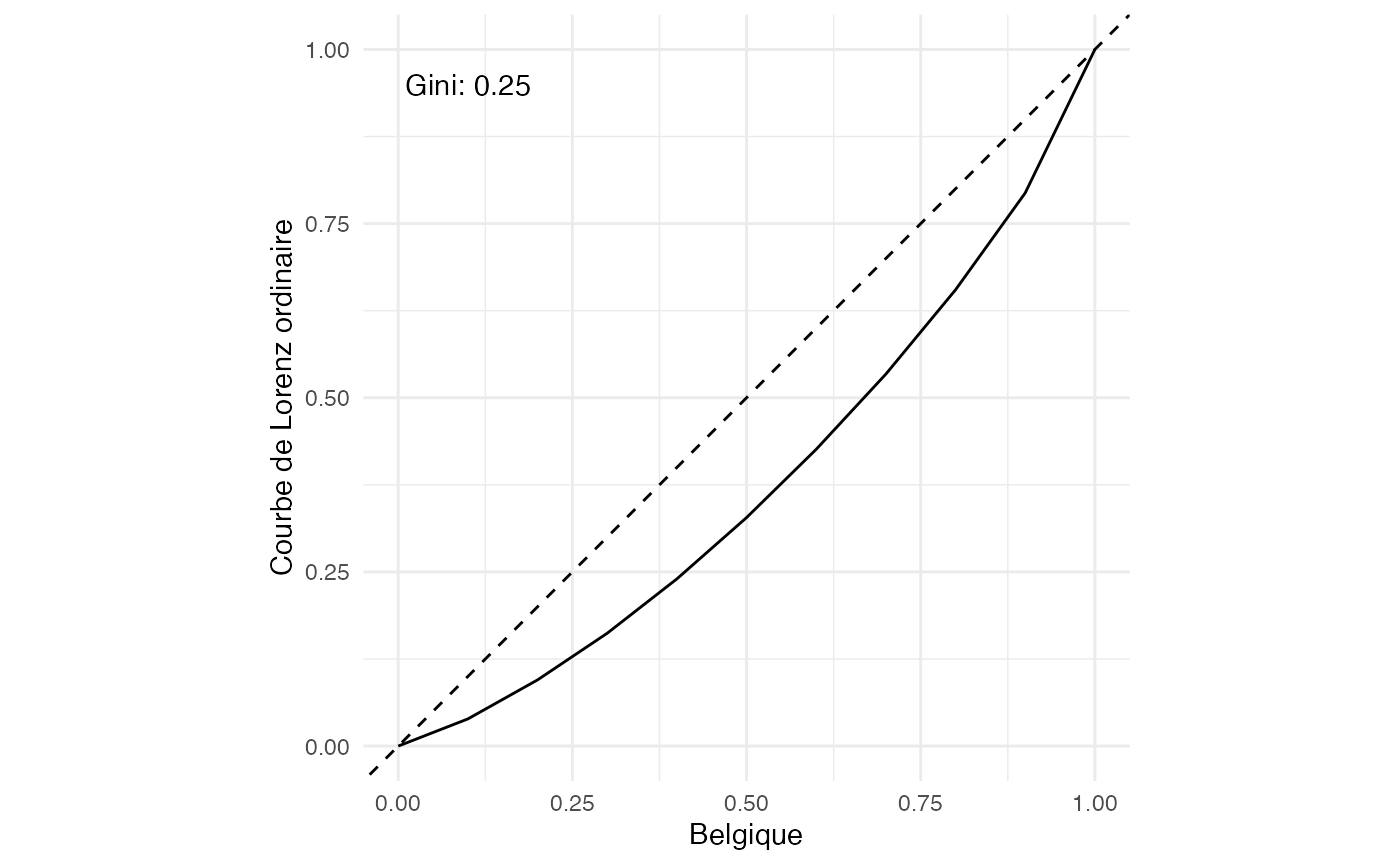
Chemin = "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
ggsave(filename = paste(Chemin,"Lorenz_Belgique.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Lorenz curve", paper = "special", colormodel = colmodel)
ggplot(Richesse, aes(France)) +
stat_lorenz() +
coord_fixed() +
geom_abline(linetype = "dashed") +
theme_minimal() +
ylab("Courbe de Lorenz ordinaire") +
annotate_ineq(Richesse$France,
measure_ineq = "Gini")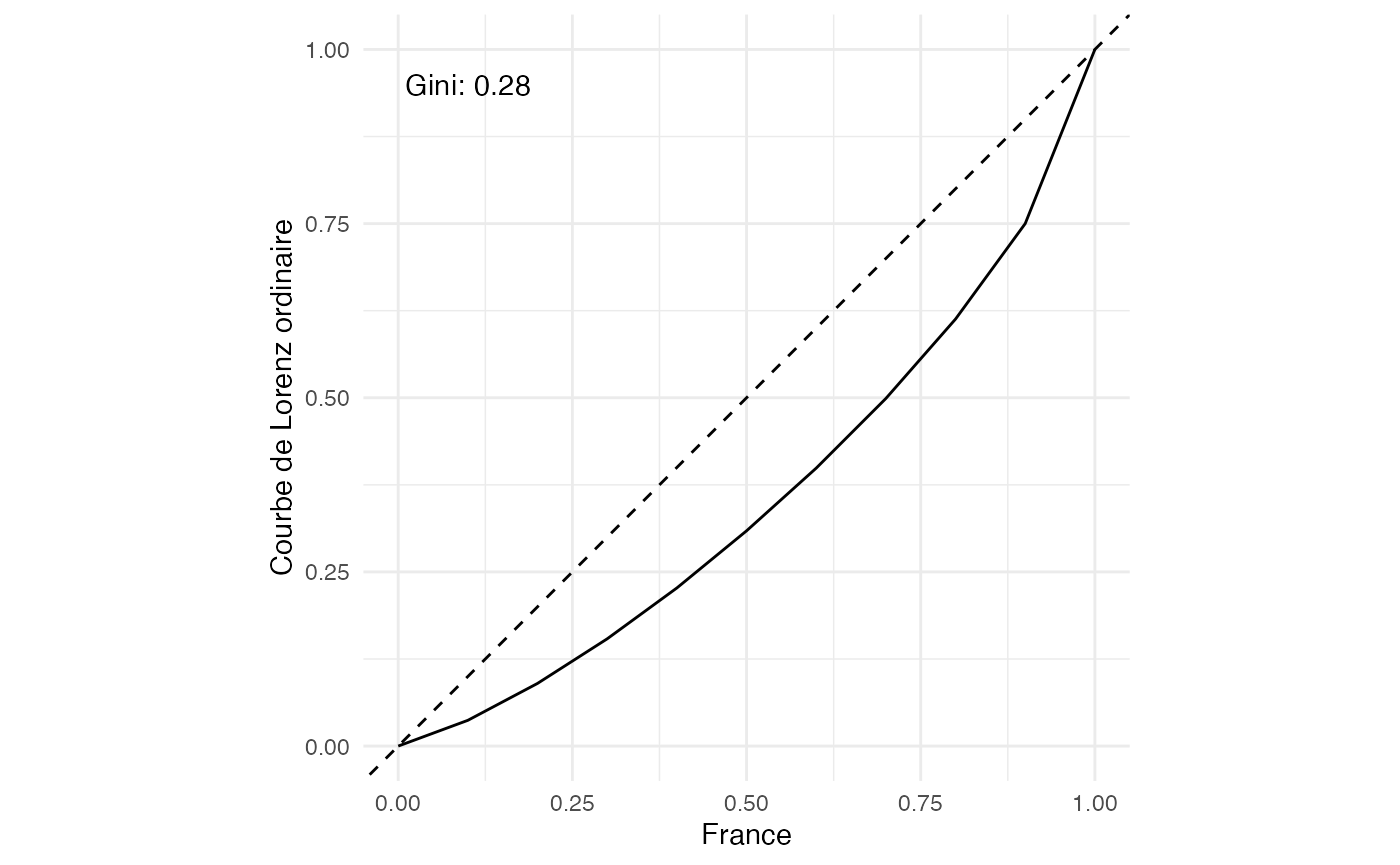
Représentations graphiques univariés
Histogramme
data3 <- rep(c(2,3.5,4.75,5.75,7,9,10.5,11.75),c(4,8,10,14,20,12,9,3))
table(data3)
#> data3
#> 2 3.5 4.75 5.75 7 9 10.5 11.75
#> 4 8 10 14 20 12 9 3
data4 <-rnorm(25,8,3)
layout(1:2)
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data4,breaks=c(min(c(data4,1)),3,4,5.5,6,8,10,11,max(12.5,data4)),main="",freq=F,xlab="",ylab="",lwd=2)
histofre <- hist(data4,main="",breaks=8,freq=F,xlab="",ylab="",lwd=2,xlim=c(min(data4),max(data4)))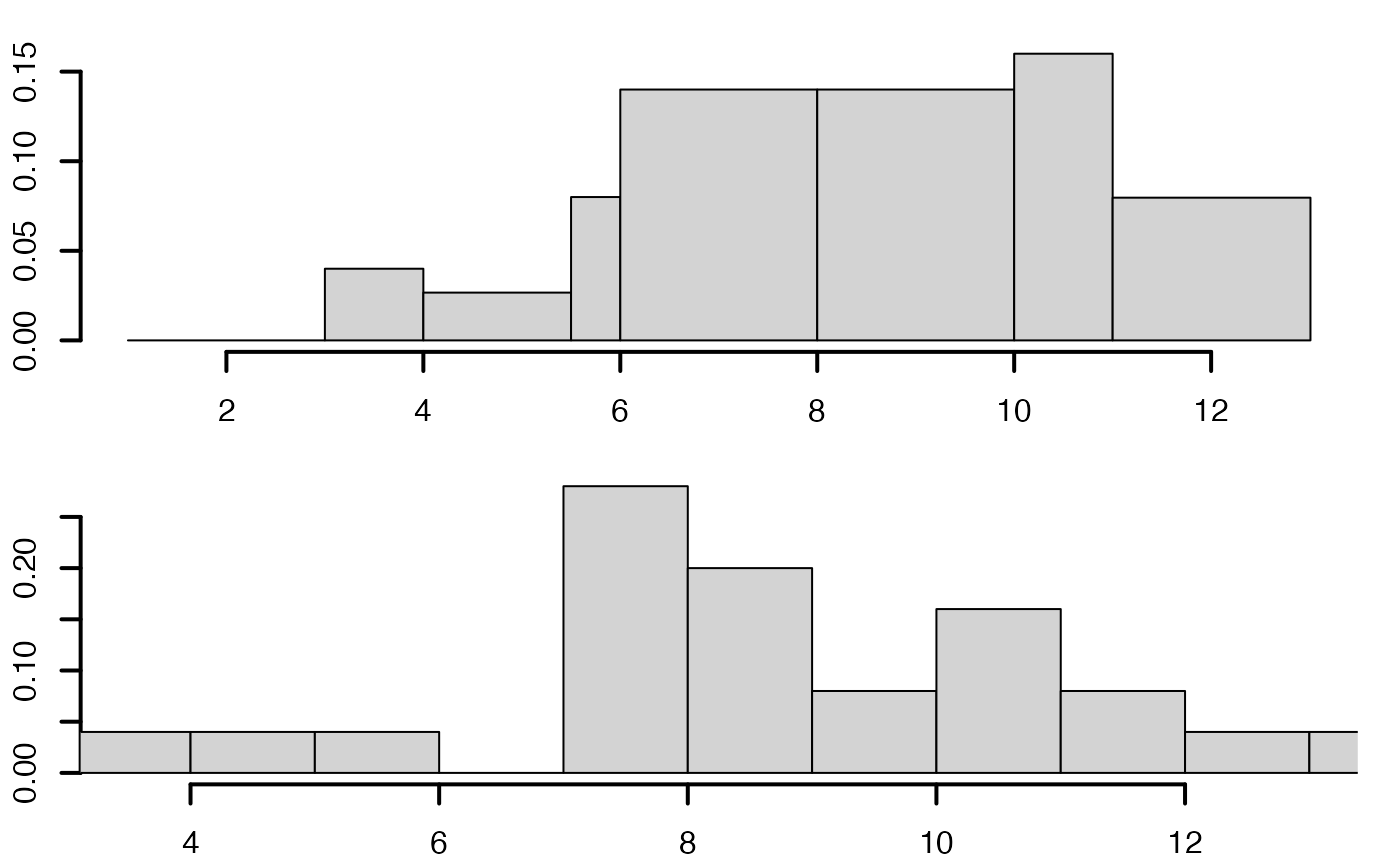
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"exemple_histo.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "europeSalaries boxplot", paper = "special")
layout(1:2)
oldpar <- par()
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data4,main="",breaks=8,freq=F,xlab="",ylab="",lwd=2,xlim=c(min(data4),max(data4)))
histofre <- hist(data4,breaks=c(min(c(data4,1)),3,4,5.5,6,8,10,11,max(12.5,data4)),main="",freq=F,xlab="",ylab="",lwd=2)
#suppressWarnings(par(oldpar))
dev.off()
#> agg_png
#> 2Avec le package MASS.
layout(1:2)
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
truehist(data4,breaks=c(min(c(data4,1)),3,4,5.5,6,8,10,11,max(12.5,data4)),main="",freq=F,xlab="",ylab="",lwd=2)
#> Warning in plot.window(...): "freq" is not a graphical parameter
#> Warning in plot.xy(xy, type, ...): "freq" is not a graphical parameter
#> Warning in axis(side = side, at = at, labels = labels, ...): "freq" is not a
#> graphical parameter
#> Warning in axis(side = side, at = at, labels = labels, ...): "freq" is not a
#> graphical parameter
#> Warning in box(...): "freq" is not a graphical parameter
#> Warning in title(...): "freq" is not a graphical parameter
truehist(data4,main="",nbins=8,h=diff(range(data4))/7,xlab="",ylab="",lwd=2,xlim=c(min(data4),max(data4)))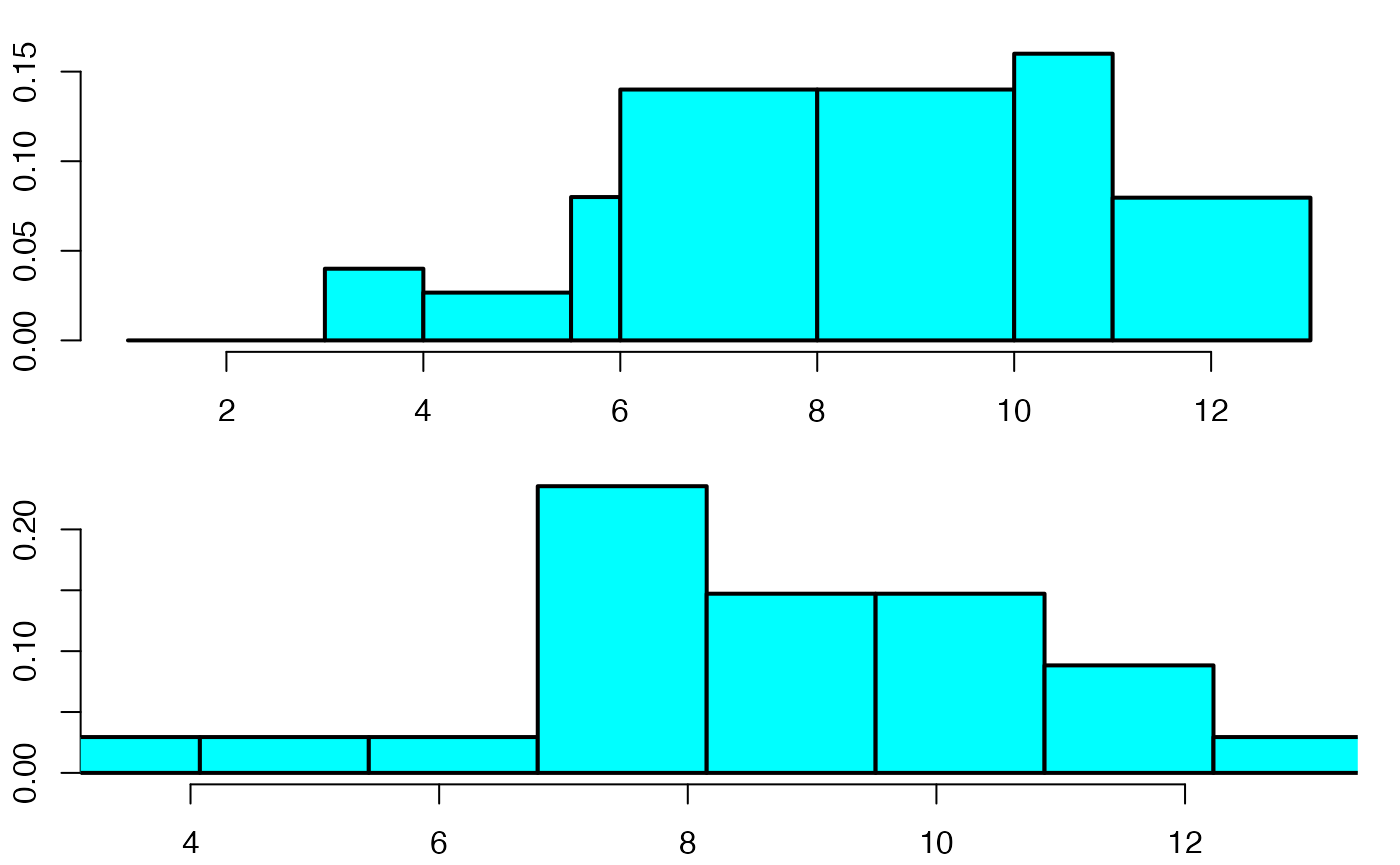
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"exemple_histo_MASS.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "europeSalaries boxplot", paper = "special")
layout(1:2)
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
truehist(data4,breaks=c(min(c(data4,1)),3,4,5.5,6,8,10,11,max(12.5,data4)),main="",freq=F,xlab="",ylab="",lwd=2)
#> Warning in plot.window(...): "freq" is not a graphical parameter
#> Warning in plot.xy(xy, type, ...): "freq" is not a graphical parameter
#> Warning in axis(side = side, at = at, labels = labels, ...): "freq" is not a
#> graphical parameter
#> Warning in axis(side = side, at = at, labels = labels, ...): "freq" is not a
#> graphical parameter
#> Warning in box(...): "freq" is not a graphical parameter
#> Warning in title(...): "freq" is not a graphical parameter
truehist(data4,main="",nbins=8,h=diff(range(data4))/7,xlab="",ylab="",lwd=2,xlim=c(min(data4),max(data4)))
#suppressWarnings(par(oldpar))
dev.off()
#> agg_png
#> 2Par secteur de l’OCDE en 2020-Q3
data(Total_Secteur)
Total_Secteur
#> Secteur NameX Effectif
#> 1 AGR Agriculture 21143585
#> 2 CONSTR Construction 35197834
#> 3 INDUSwithoutCONSTR Industrie 69941779
#> 4 MFG Fabrication 64298386
#> 5 SERV Services 368931820
NameX <- Total_Secteur$NameX
Effectif <- Total_Secteur$Effectif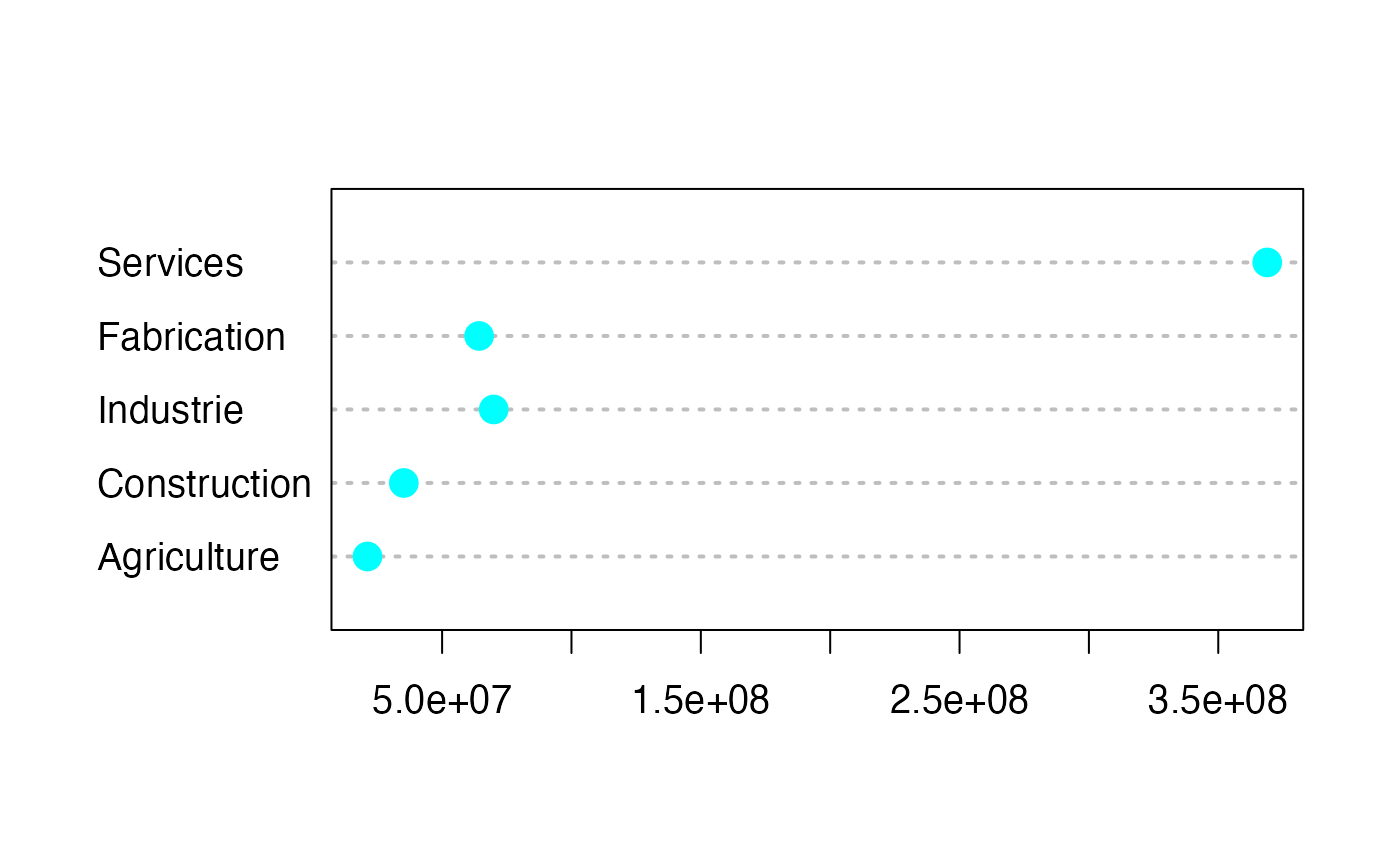
dotchart3(Effectif,labels=NameX,pch=19,col="#00FFFF")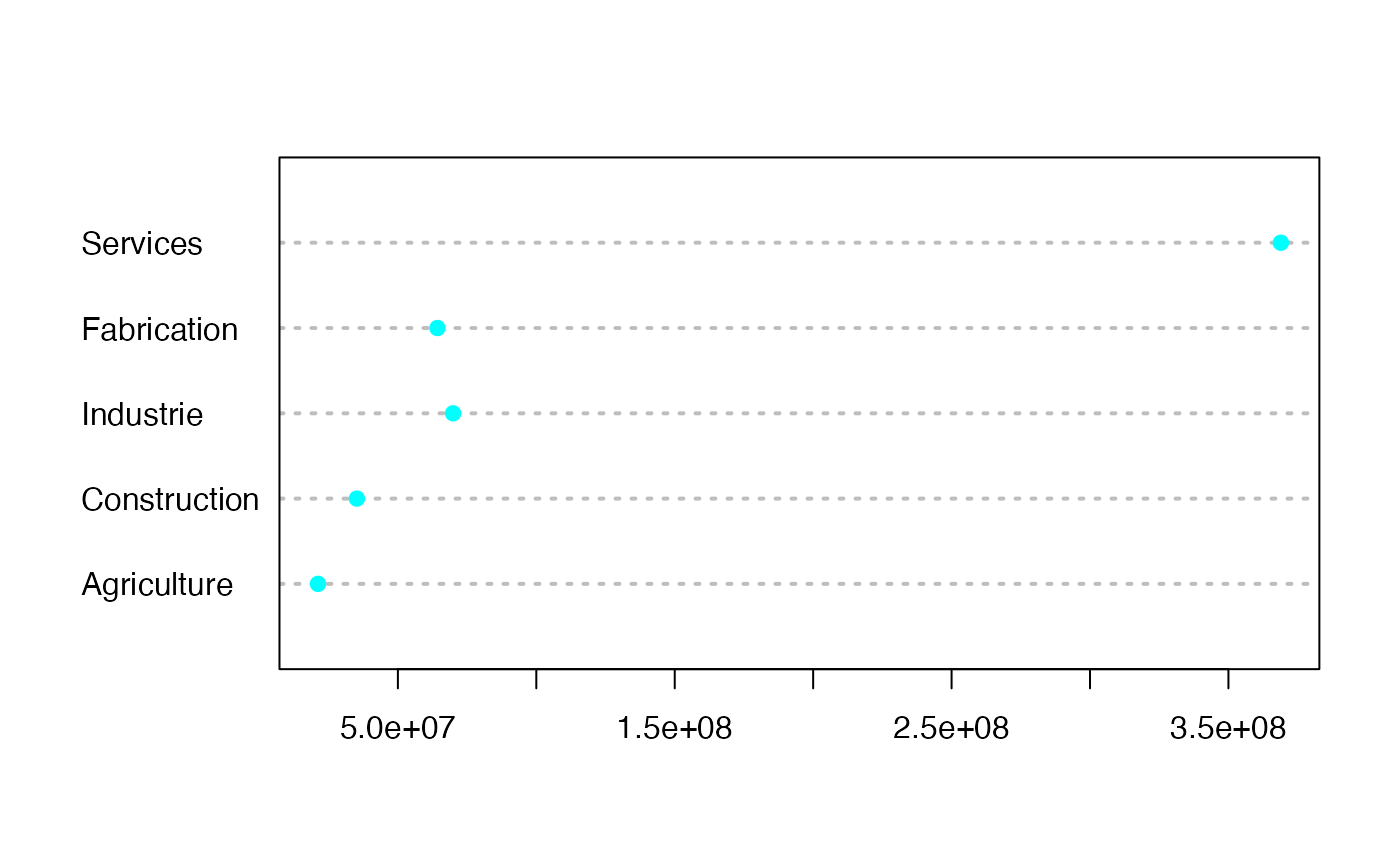
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/"
colmodel="cmyk"
pdf(file = paste(Chemin,"pointshoriz.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(Effectif,labels=NameX,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"pointshoriz_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(Effectif/sum(Effectif),labels=NameX,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dev.off()
#> agg_png
#> 2
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX,col=c("black","#00FFFF","black","#00FFFF"),density = c(rep(10,4),15),border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)#,density = c(4*(4:1))
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
#pie(Effectif,labels=NameX,col=c("#00000050","#00FFFF80","#00000050","#00FFFF80"),border=c("#00000050","#00FFFF80","#00000050","#00FFFF80"),cex=1.2)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX,col=c("black","#00FFFF","black","#00FFFF"),border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)
pdf(file = paste(Chemin,"diagcircu.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX,col=c("black","#00FFFF","black","#00FFFF","#FF00FF"),border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"diagcircubis.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX,col=c("black","#00FFFF","black","#00FFFF"),density = c(rep(10,4),15),border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)#,density = c(4*(4:1))
dev.off()
#> agg_png
#> 2
NameXbar <- NameX
par(mar = c(8.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,cex.names=1.2)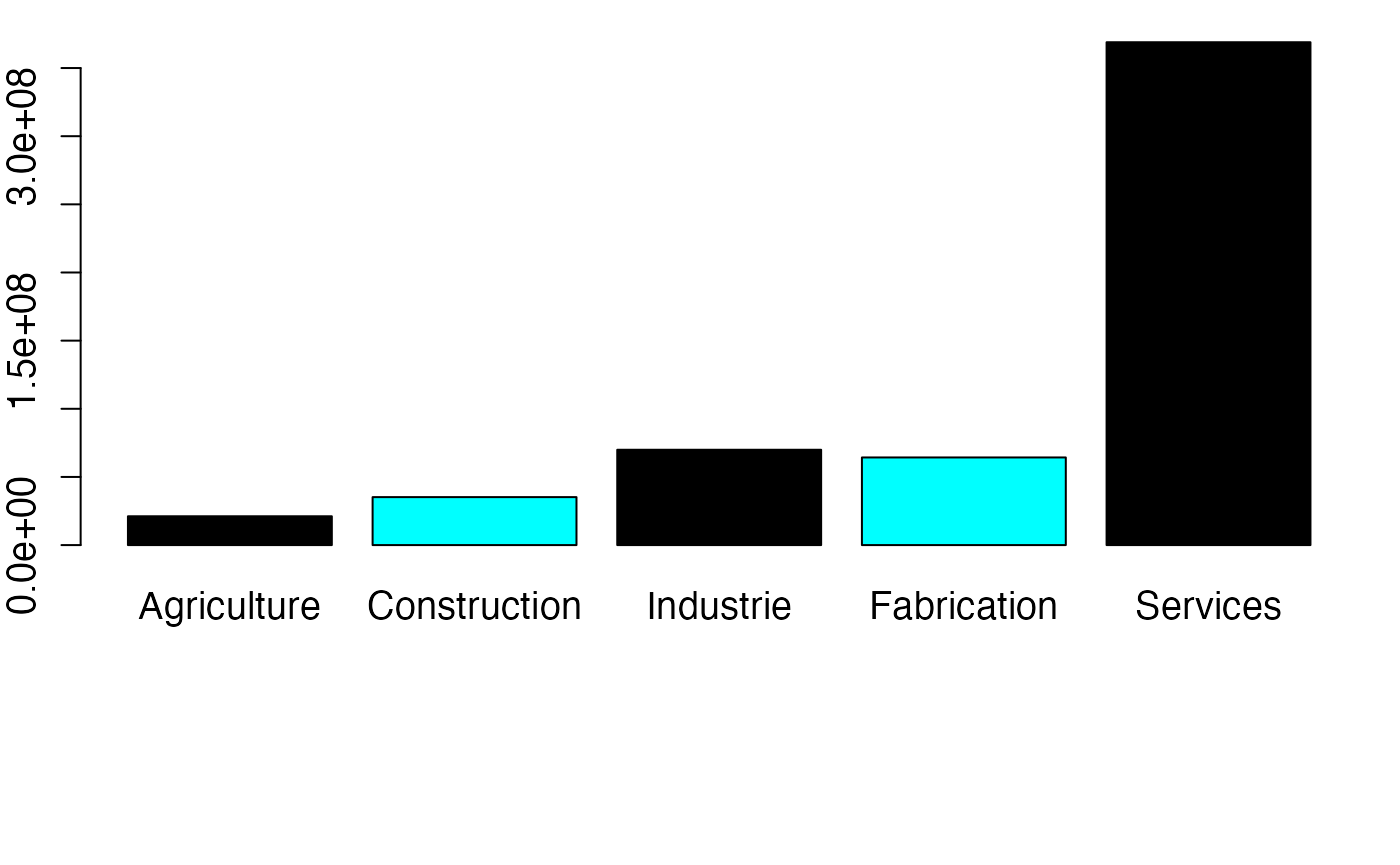
pdf(file = paste(Chemin,"barverti.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(8.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),las=3)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barvertibis.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(8.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barverti_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(8.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),las=3)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barvertibis_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(8.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)
dev.off()
#> agg_png
#> 2
NameXbar <- NameX
par(mar = c(2, 8.1, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,cex.names=1.2,horiz=TRUE,las=1)
pdf(file = paste(Chemin,"barhoriz.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 8.1, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),horiz=TRUE,las=1)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barhorizbis.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 8.1, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,horiz=TRUE,las=1)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barhoriz_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 8.1, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),horiz=TRUE,las=1)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barhorizbis_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 8.1, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,horiz=TRUE,las=1)
dev.off()
#> agg_png
#> 2
#suppressWarnings(par(oldpar))Par pays de l’OCDE en 2020-Q3
data(Total_Pays)
Total_Pays
#> NameX Effectif
#> 1 AUT 5167648
#> 2 BEL 5721200
#> 3 CAN 21122321
#> 4 CHE 5568900
#> 5 CHL 8732089
#> 6 COL 22791085
#> 7 CZE 7015674
#> 8 DNK 3343269
#> 9 ESP 22701902
#> 10 EST 200241
#> 11 FIN 1154495
#> 12 FRA 31775373
#> 13 GBR 37353540
#> 14 GRC 4382010
#> 15 HUN 5787638
#> 16 IRL 2692289
#> 17 ISL 40222
#> 18 ISR 4366751
#> 19 ITA 28409214
#> 20 JPN 81977909
#> 21 KOR 33262656
#> 22 LTU 1647481
#> 23 LUX 316889
#> 24 LVA 253516
#> 25 NLD 10143660
#> 26 NOR 3114348
#> 27 NZL 3242630
#> 28 POL 21006175
#> 29 PRT 5902090
#> 30 SVK 3385346
#> 31 SVN 1275598
#> 32 SWE 5872257
#> 33 TUR 33604952
#> 34 USA 171209618
Effectif<-Total_Pays$Effectif
NameX<-Total_Pays$NameX
NameX2<-Total_Pays$NameX
dotchart3(Effectif,labels=NameX2,pch=19,col="#00FFFF")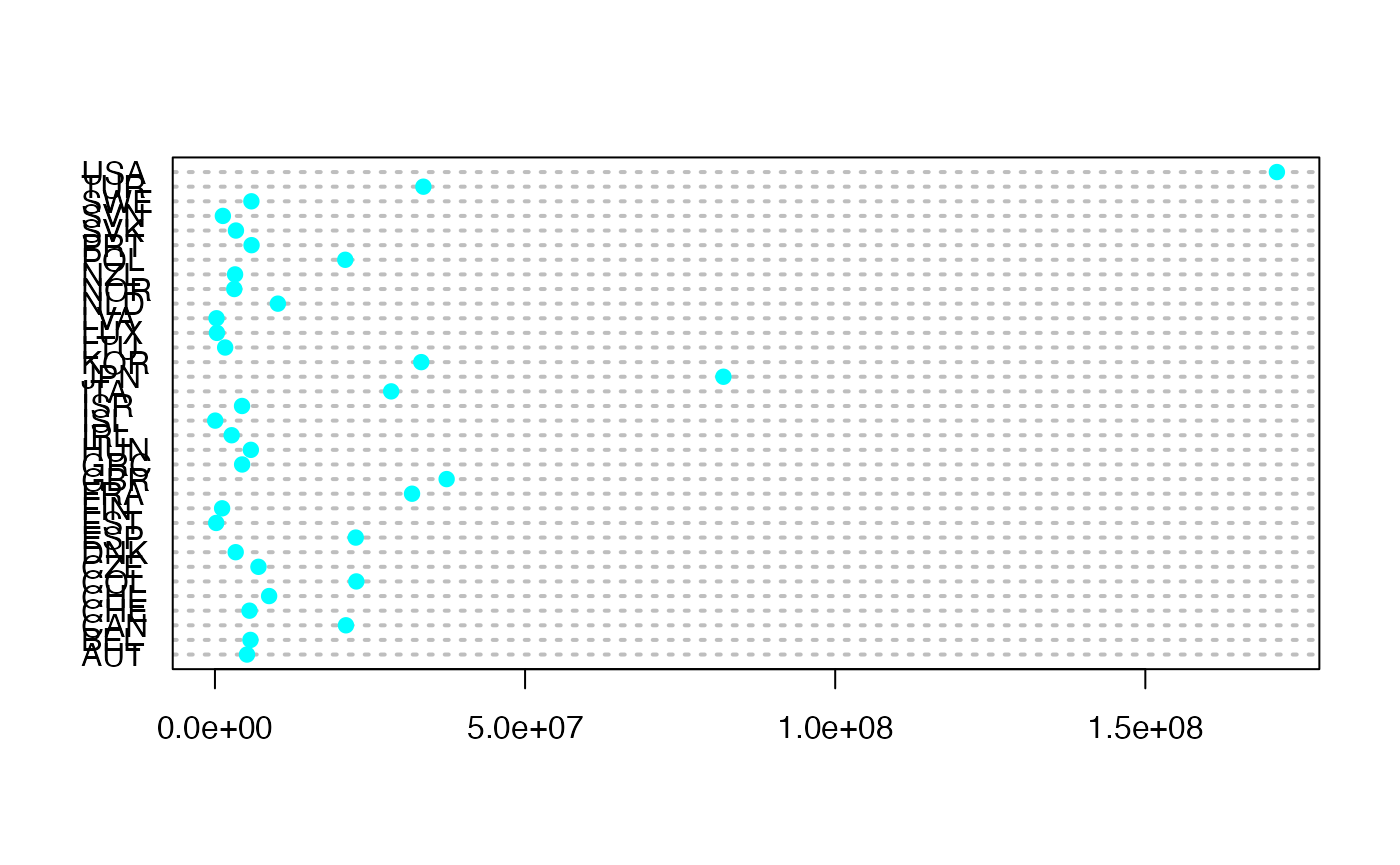
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/"
colmodel="cmyk"
pdf(file = paste(Chemin,"pointshoriz_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(Effectif,labels=NameX2,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"pointshoriz_fre_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(Effectif/sum(Effectif),labels=NameX2,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dev.off()
#> agg_png
#> 2
if(!("dplyr" %in% installed.packages())){install.packages("dplyr")}
library(dplyr)
#>
#> Attaching package: 'dplyr'
#> The following object is masked from 'package:MASS':
#>
#> select
#> The following objects are masked from 'package:stats':
#>
#> filter, lag
#> The following objects are masked from 'package:base':
#>
#> intersect, setdiff, setequal, union
df <- data.frame(NameX2=rev(NameX2),
Effectif=rev(Effectif))
df <- df %>%
mutate(text_y = cumsum(Effectif) - Effectif/2)
df
#> NameX2 Effectif text_y
#> 1 USA 171209618 85604809
#> 2 TUR 33604952 188012094
#> 3 SWE 5872257 207750698
#> 4 SVN 1275598 211324626
#> 5 SVK 3385346 213655098
#> 6 PRT 5902090 218298816
#> 7 POL 21006175 231752948
#> 8 NZL 3242630 243877351
#> 9 NOR 3114348 247055840
#> 10 NLD 10143660 253684844
#> 11 LVA 253516 258883432
#> 12 LUX 316889 259168634
#> 13 LTU 1647481 260150820
#> 14 KOR 33262656 277605888
#> 15 JPN 81977909 335226170
#> 16 ITA 28409214 390419732
#> 17 ISR 4366751 406807714
#> 18 ISL 40222 409011201
#> 19 IRL 2692289 410377456
#> 20 HUN 5787638 414617420
#> 21 GRC 4382010 419702244
#> 22 GBR 37353540 440570019
#> 23 FRA 31775373 475134476
#> 24 FIN 1154495 491599410
#> 25 EST 200241 492276778
#> 26 ESP 22701902 503727849
#> 27 DNK 3343269 516750434
#> 28 CZE 7015674 521929906
#> 29 COL 22791085 536833286
#> 30 CHL 8732089 552594872
#> 31 CHE 5568900 559745367
#> 32 CAN 21122321 573090978
#> 33 BEL 5721200 586512738
#> 34 AUT 5167648 591957162
if(!("ggrepel" %in% installed.packages())){install.packages("ggrepel")}
library(ggrepel)
theme_set(theme_void())
p1 <- ggplot(data = df, aes(x = "", y = Effectif, fill = NameX2)) +
geom_bar(stat = "identity")+
geom_label_repel(aes(label = NameX2, y = text_y), nudge_x = 1.6)+
coord_polar(theta = "y") + theme(legend.position='none')+
theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank())
p1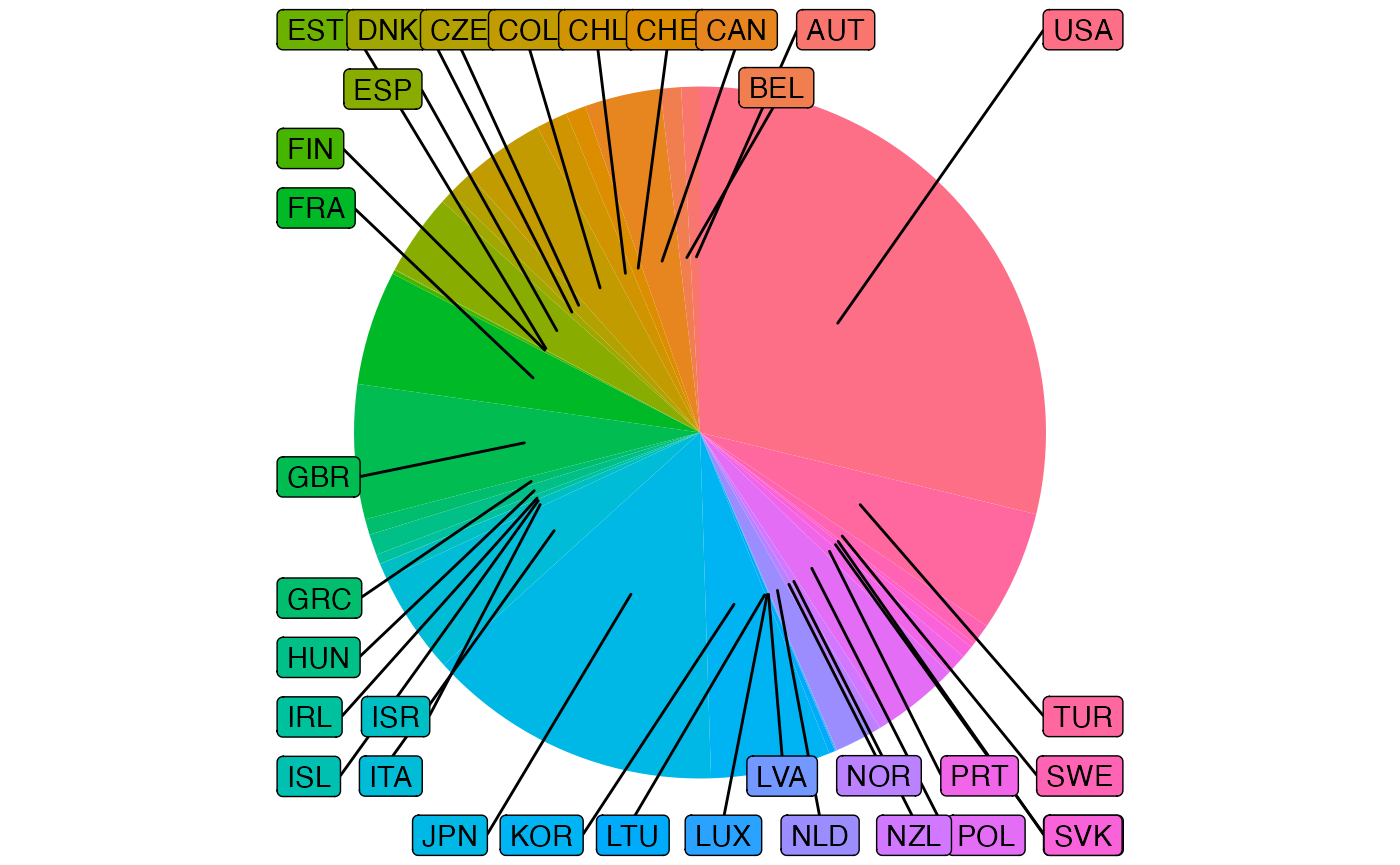
p1 + scale_fill_manual(values=rep(c(grey.colors(4)[c(2,4)]),17))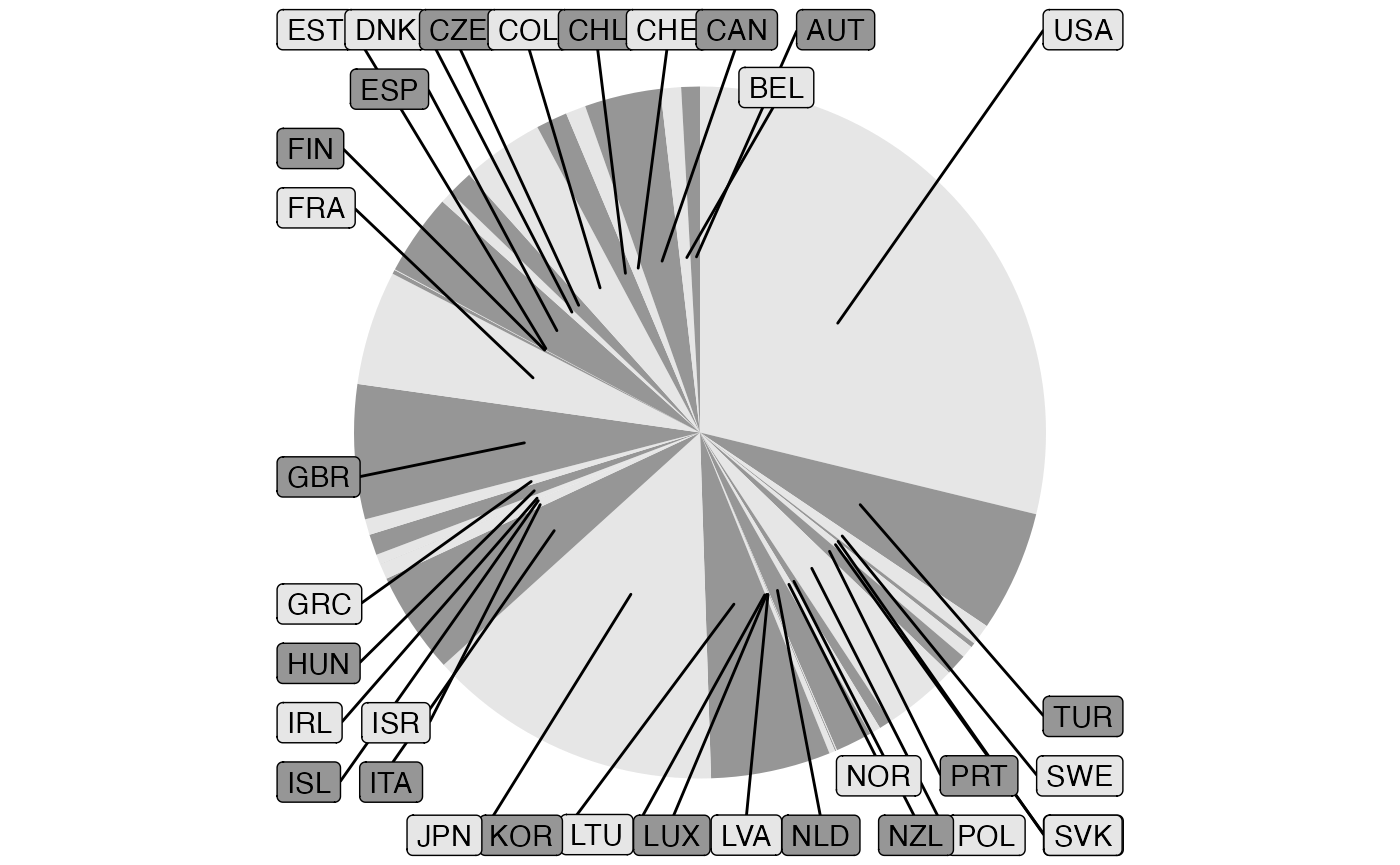
ggsave(filename = paste(Chemin,"diagcircu_app2_ggplot2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX2,col=c("black","#00FFFF","black","#00FFFF"),border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)
pdf(file = paste(Chemin,"diagcircu_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX2,col=c("black","#00FFFF","black","#00FFFF"),border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)
dev.off()
#> agg_png
#> 2
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX2,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)#,density = c(4*(4:1))
pdf(file = paste(Chemin,"diagcircubis_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(Effectif,labels=NameX2,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1.2)#,density = c(4*(4:1))
dev.off()
#> agg_png
#> 2
par(mar = c(6.3, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
NameXbar <- NameX2
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,cex.names=1.2,las=3)
pdf(file = paste(Chemin,"barverti_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(6.3, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),las=3)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barvertibis_app2.pdf",sep=""),
width = 8.1, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(6.3, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barverti_fre_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(6.3, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),las=3)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barvertibis_fre_app2.pdf",sep=""),
width = 8.1, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(6.3, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)
dev.off()
#> agg_png
#> 2
par(mar = c(2, 7, 1, 1) + 0.1, mgp = c(2, 1, 0))
NameXbar <- NameX2
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,cex.names=1.2,las=1,horiz=TRUE)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"barhoriz_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 7, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),las=1,horiz=TRUE)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barhorizbis_app2.pdf",sep=""),
width = 8.1, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 7, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif,names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=1,horiz=TRUE)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barhoriz_fre_app2.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 7, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),las=1,horiz=TRUE)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"barhorizbis_fre_app2.pdf",sep=""),
width = 8.1, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 7, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(Effectif/sum(Effectif),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=1,horiz=TRUE)
dev.off()
#> agg_png
#> 2Nombre de personnes par famille
data(Personnes_Foyer)
ni <- Personnes_Foyer$ni
xi <- Personnes_Foyer$xi
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,xaxt="n")
axis(1, at = 1:6, labels = c(1:5,6))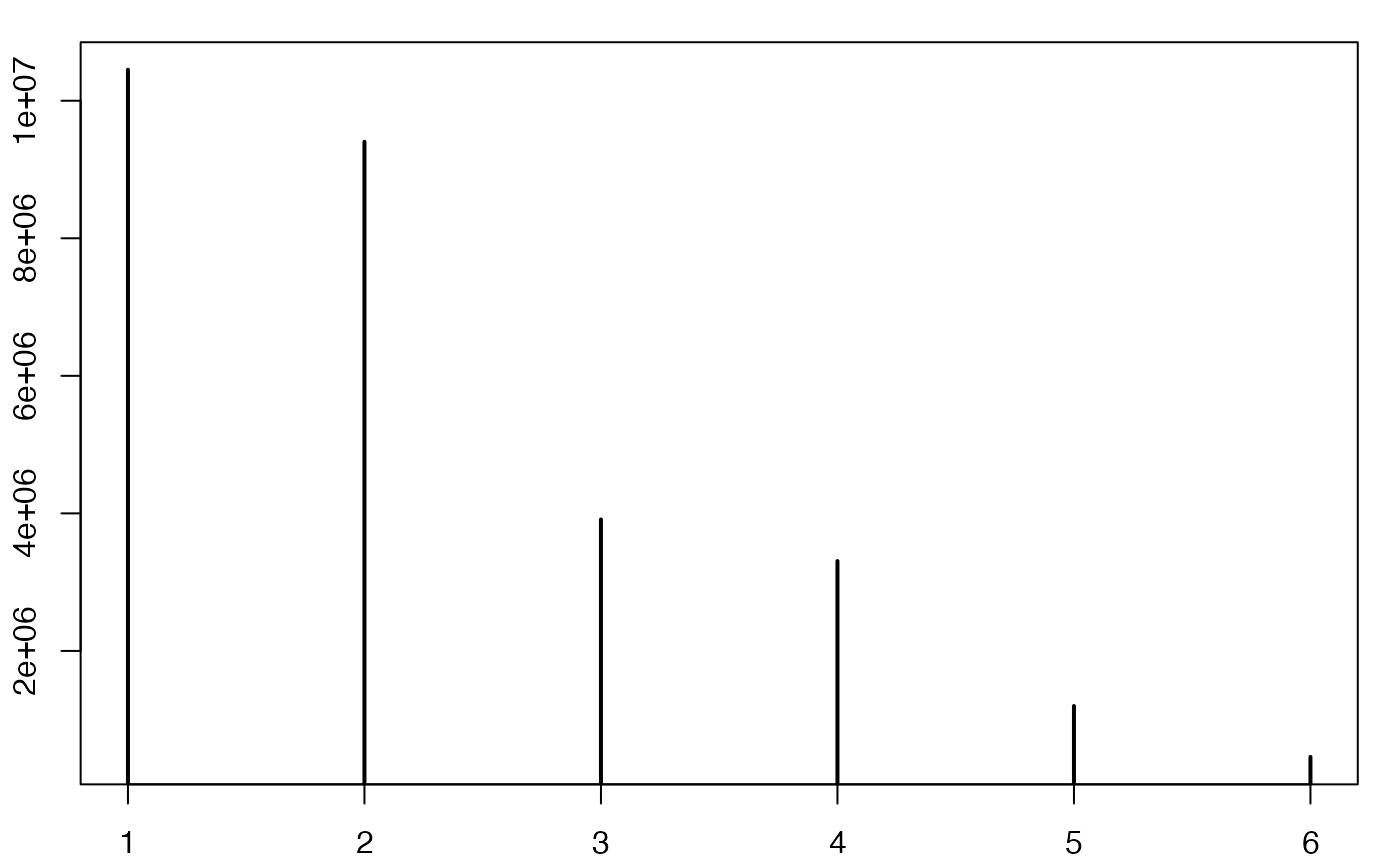
#suppressWarnings(par(oldpar))
if(!("ggplot2" %in% installed.packages())){install.packages("ggplot2")}
library(ggplot2)Diagramme en bâtons des effectifs
p1 <- ggplot(Personnes_Foyer) +
geom_col(aes(y = ni, x = xi),width = .05)+
xlab( expression(paste(x[i])))+
ylab( expression(paste(n[i])))+
scale_x_discrete(name =expression(paste(x[i])),
limits=c("1","2","3","4","5","6"))
p1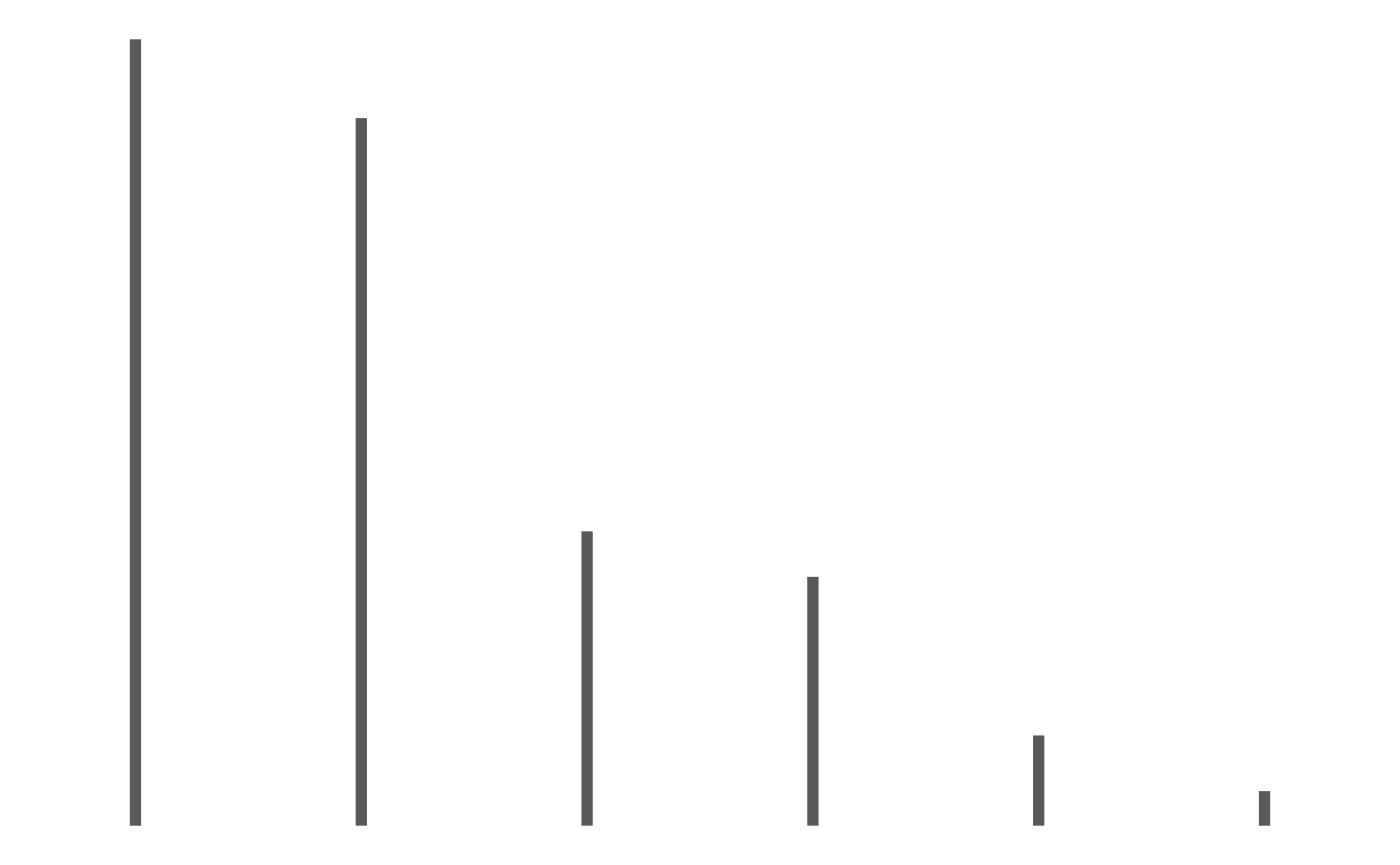
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Sources/"
colmodel="cmyk"
ggsave(paste(Chemin,"batoneffnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs",
paper = "special", colormodel = colmodel)
oldpar <- par()
p1 <- ggplot(Personnes_Foyer) +
geom_col(aes(y = ni/sum(ni), x = xi),width = .05)+
xlab( expression(paste(x[i])))+
ylab( expression(paste(n[i])))+
scale_x_discrete(name =expression(paste(x[i])),
limits=c("1","2","3","4","5","6"))
p1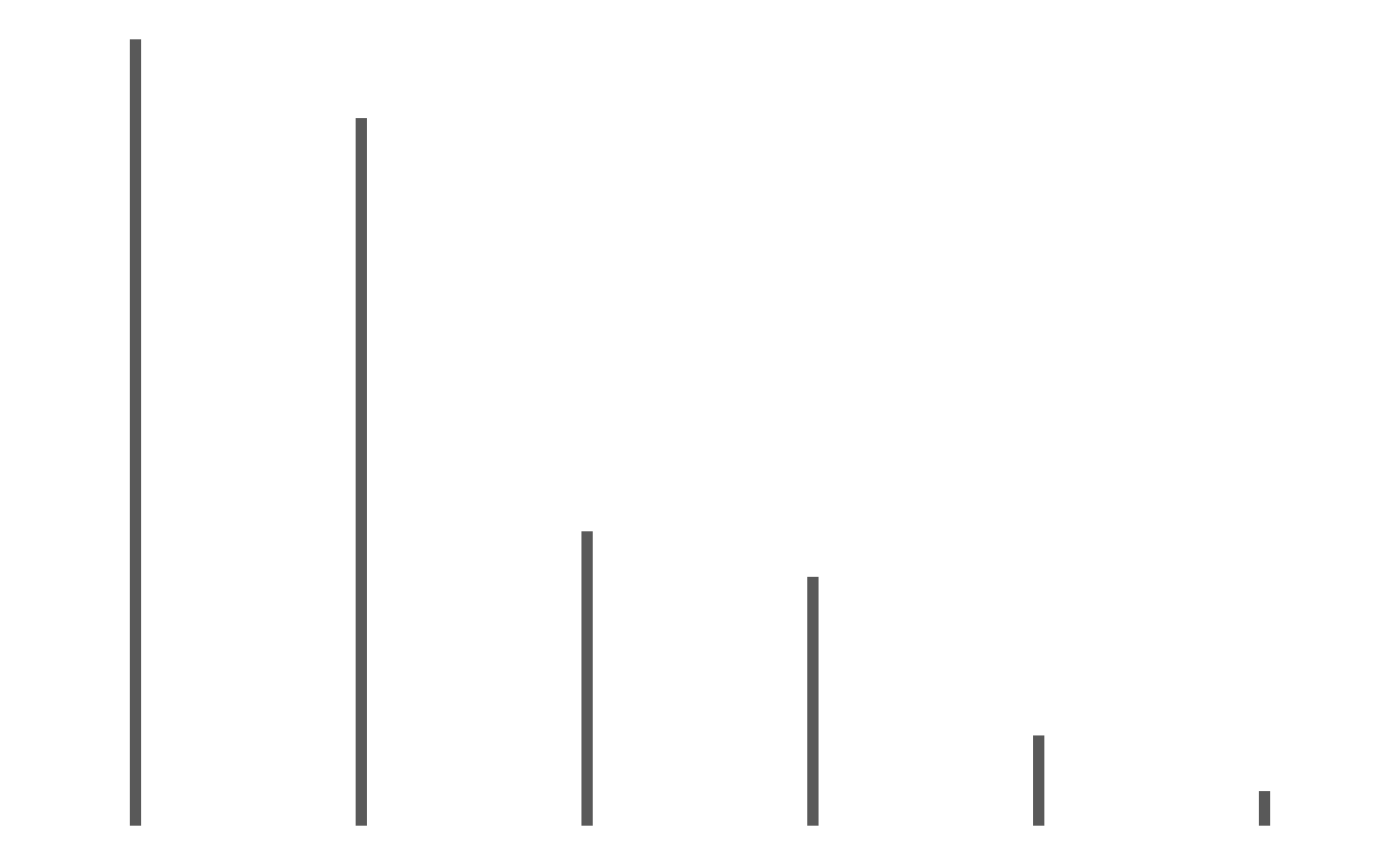
#suppressWarnings(par(oldpar))
ggsave(paste(Chemin,"batonfrenew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs",
paper = "special", colormodel = colmodel)
oldpar <- par()
ggplot(Personnes_Foyer, aes(x = xi)) +
geom_ribbon(aes(ymin=0,ymax=ni,group=""),fill=I("gray80"))+
geom_line(aes(y=ni,group=""), size=1, color=I("gray40")) +
geom_pointrange(aes(ymin=0,ymax=ni,y=ni,group=""), size=1)+
xlab( expression(paste(x[i])))+
ylab( expression(paste(n[i])))+
scale_x_discrete(name =expression(paste(x[i])),
limits=c("1","2","3","4","5","6"))
#> Warning: Using `size` aesthetic for lines was deprecated in ggplot2 3.4.0.
#> ℹ Please use `linewidth` instead.
#> This warning is displayed once every 8 hours.
#> Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
#> generated.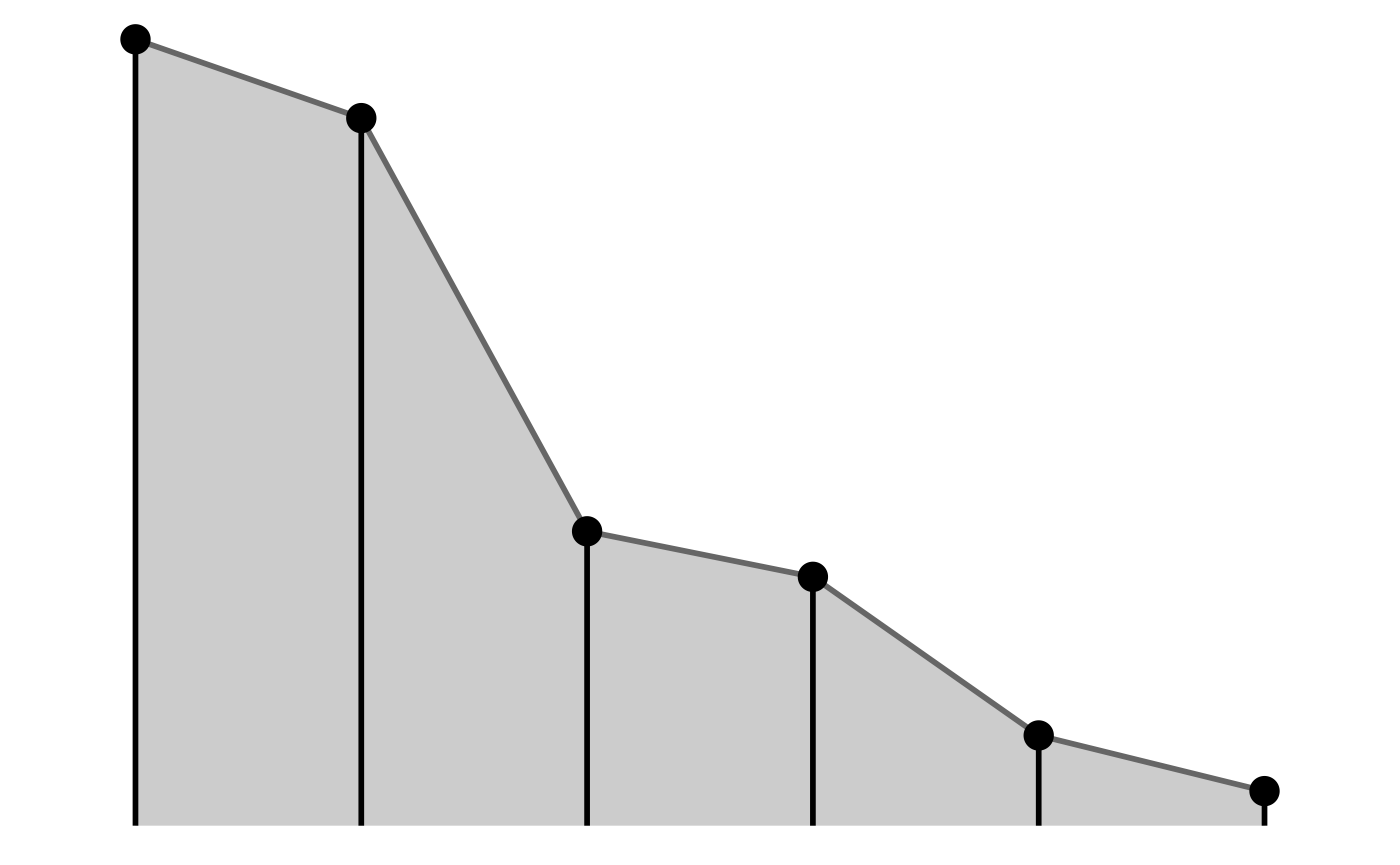
#suppressWarnings(par(oldpar))
ggsave(paste(Chemin,"polyeffnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs",
paper = "special", colormodel = colmodel)
oldpar <- par()
ggplot(Personnes_Foyer, aes(x = xi)) +
geom_ribbon(aes(ymin=0,ymax=ni/sum(ni),group=""),fill=I("gray80"))+
geom_line(aes(y=ni/sum(ni),group=""), size=1, color=I("gray40")) +
geom_pointrange(aes(ymin=0,ymax=ni/sum(ni),y=ni/sum(ni),group=""), size=1)+
xlab( expression(paste(x[i])))+
ylab( expression(paste(n[i])))+
scale_x_discrete(name =expression(paste(x[i])),
limits=c("1","2","3","4","5","6"))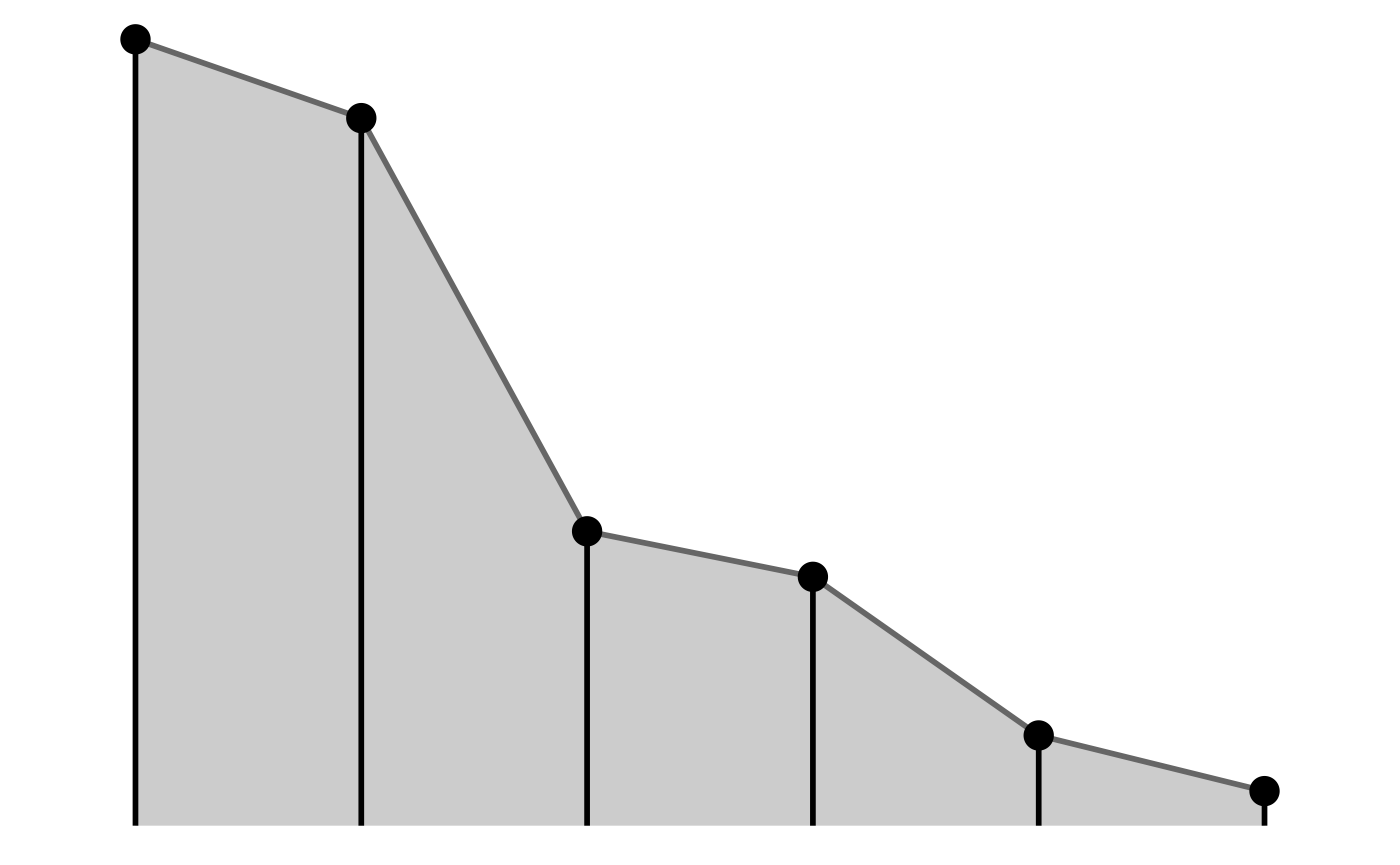
#suppressWarnings(par(oldpar))
ggsave(paste(Chemin,"polyfrenew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs",
paper = "special", colormodel = colmodel)
nisum <- rep(ni[1],length(ni))
for (iii in 2:length(ni))
{
nisum[iii] <- nisum[iii-1]+ni[iii]
}
data3<-data.frame(xi=xi,nisum=nisum)
oldpar <- par()
ggplot(data3, aes(x = xi)) +
geom_ribbon(aes(ymin=0,ymax=nisum,group=""),fill=I("gray80"))+
geom_line(aes(y=nisum,group=""), size=1, color=I("gray40")) +
geom_pointrange(aes(ymin=0,ymax=nisum,y=nisum,group=""), size=1)+
xlab( expression(paste(x[i])))+
ylab( expression(paste(n[i])))+
scale_x_discrete(name =expression(paste(x[i])),
limits=c("1","2","3","4","5","6"))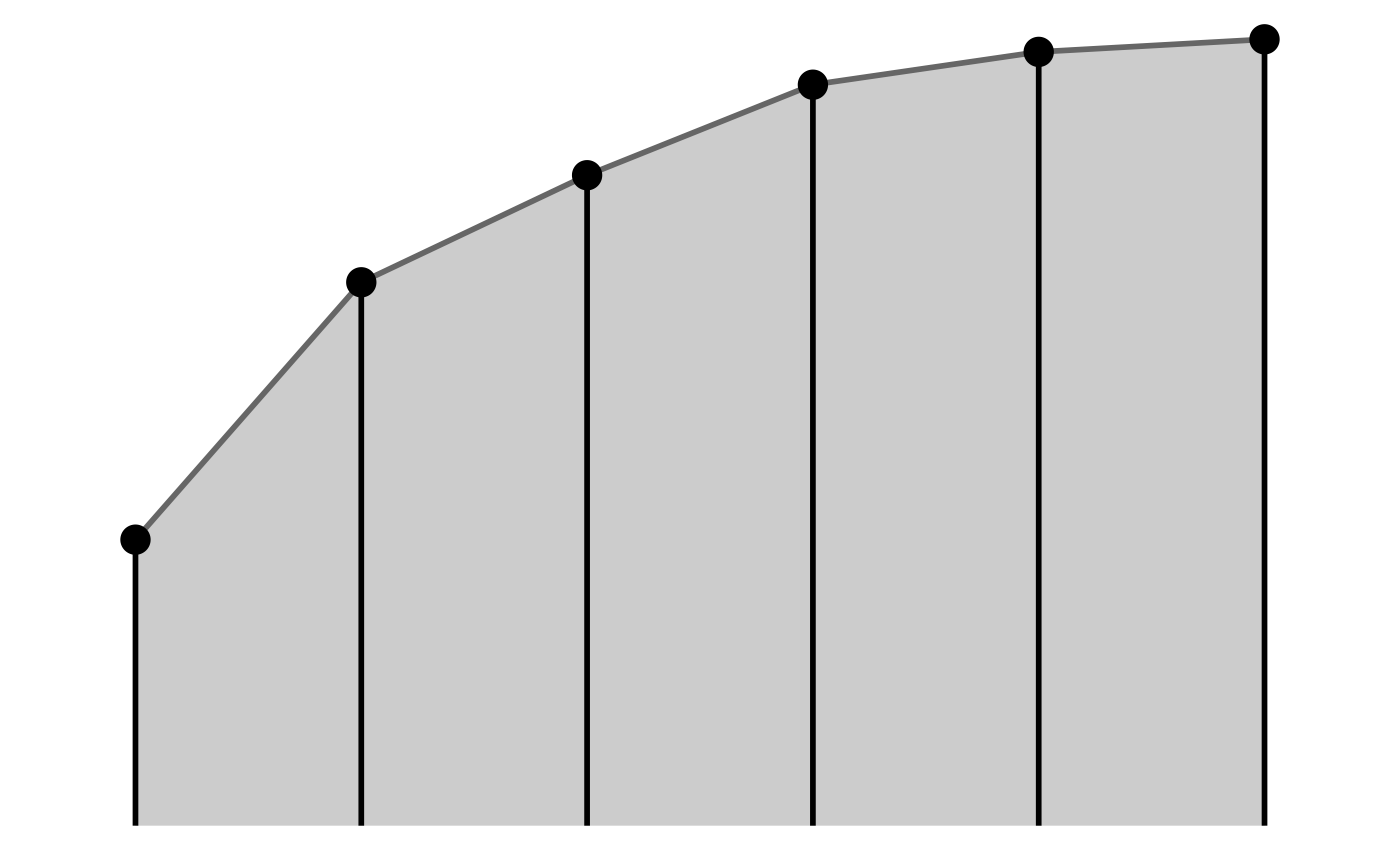
#suppressWarnings(par(oldpar))
ggsave(paste(Chemin,"polyeffcumnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs",
paper = "special", colormodel = colmodel)
ggsave(paste(Chemin,"polyeffcumnew.svg",sep=""),device="svg",
width = 8, height = 7)
data4<-data.frame(xi=xi,nifre=nisum/sum(ni))
oldpar <- par()
ggplot(data4, aes(x = xi)) +
geom_ribbon(aes(ymin=0,ymax=nifre,group=""),fill=I("gray80"))+
geom_line(aes(y=nifre,group=""), size=1, color=I("gray40")) +
geom_pointrange(aes(ymin=0,ymax=nifre,y=nifre,group=""), size=1)+
xlab( expression(paste(x[i])))+
ylab( expression(paste(n[i])))+
scale_x_discrete(name =expression(paste(x[i])),
limits=c("1","2","3","4","5","6"))
#> Warning: Using `size` aesthetic for lines was deprecated in ggplot2 3.4.0.
#> ℹ Please use `linewidth` instead.
#> This warning is displayed once every 8 hours.
#> Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
#> generated.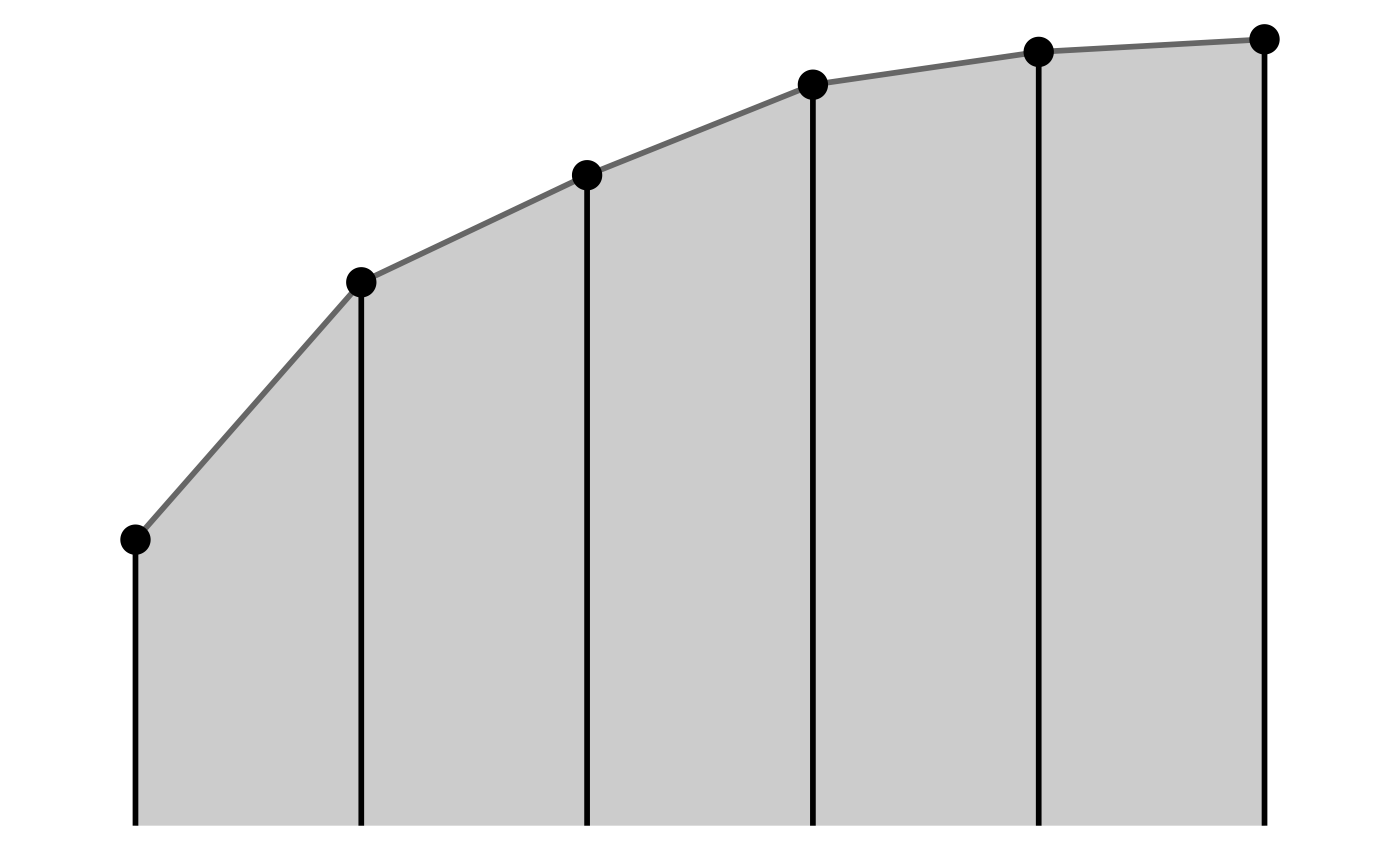
#suppressWarnings(par(oldpar))
ggsave(paste(Chemin,"frecumnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs",
paper = "special", colormodel = colmodel)
p1 <- ggplot(data3) +
geom_col(aes(y = nisum, x = xi),width = .05)+
xlab( expression(paste(x[i])))+
ylab( expression(paste(n[i])))+
scale_x_discrete(name =expression(paste(x[i])),
limits=c("1","2","3","4","5","6"))
p1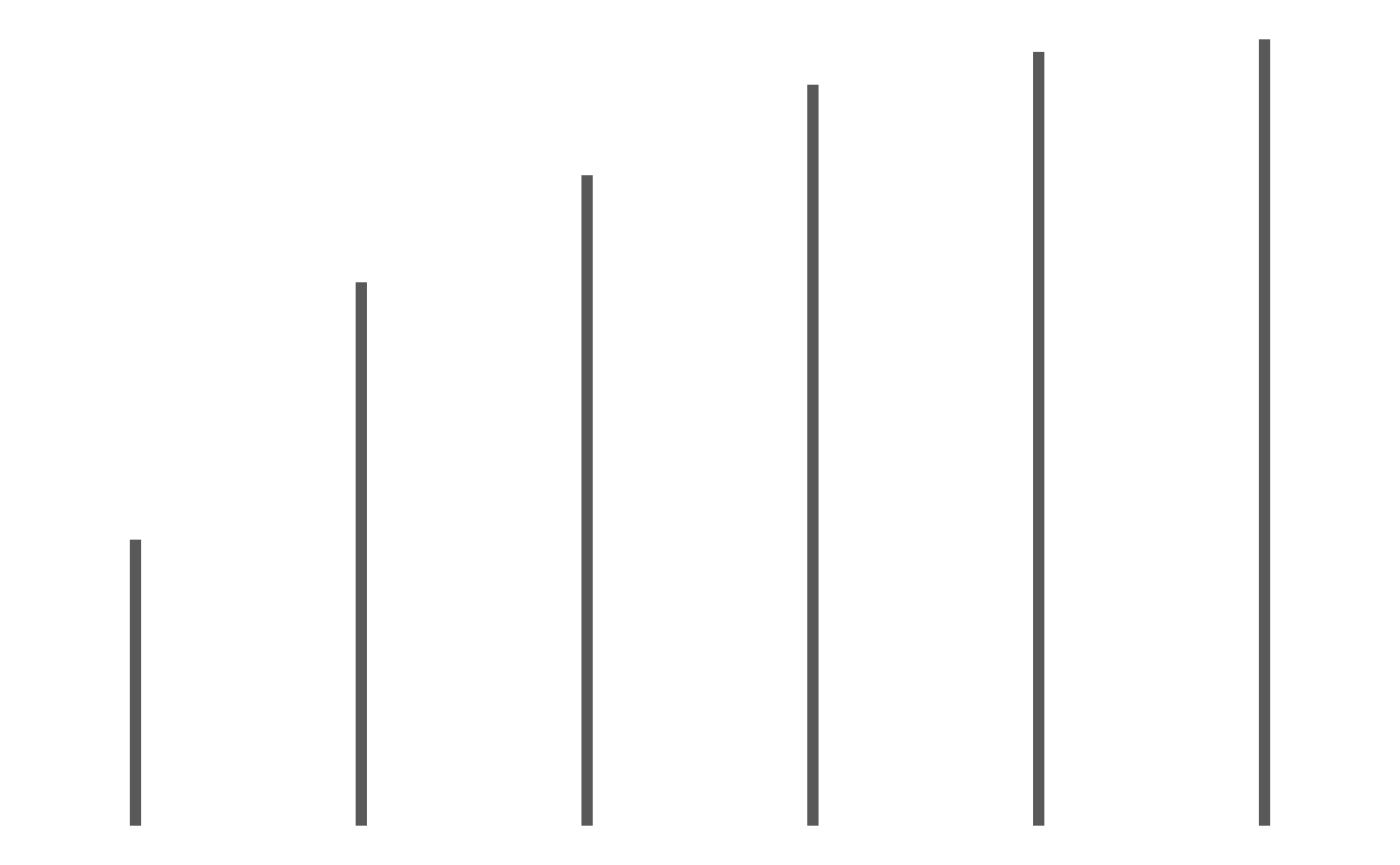
Population du Canada
data(AgevsProter_Canada_full)
valeursall <- margin.table(as.matrix(AgevsProter_Canada_full[,-1]),1)
valeurs <- valeursall[-length(valeursall)]
valeurs
#> 0 à 4 ans 5 à 9 ans 10 à 14 ans 15 à 19 ans 20 à 24 ans 25 à 29 ans
#> 1901542 2021227 2045914 2073871 2453892 2617164
#> 30 à 34 ans 35 à 39 ans 40 à 44 ans 45 à 49 ans 50 à 54 ans 55 à 59 ans
#> 2632781 2600529 2432821 2354362 2409701 2701791
#> 60 à 64 ans 65 à 69 ans 70 à 74 ans 75 à 79 ans 80 à 84 ans 85 à 89 ans
#> 2517902 2127557 1753443 1197845 798753 511048
#> 90 à 94 ans 95 à 99 ans
#> 245878 73725
centres <- c(5*(0:19)+2.5)
limites <- 5*(0:20)
oldpar <- par()
data3 <- rep(centres,valeurs)
table(data3)
#> data3
#> 2.5 7.5 12.5 17.5 22.5 27.5 32.5 37.5 42.5 47.5
#> 1901542 2021227 2045914 2073871 2453892 2617164 2632781 2600529 2432821 2354362
#> 52.5 57.5 62.5 67.5 72.5 77.5 82.5 87.5 92.5 97.5
#> 2409701 2701791 2517902 2127557 1753443 1197845 798753 511048 245878 73725
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=limites,main="",freq=T,xlab="",ylab="",lwd=2,density=10)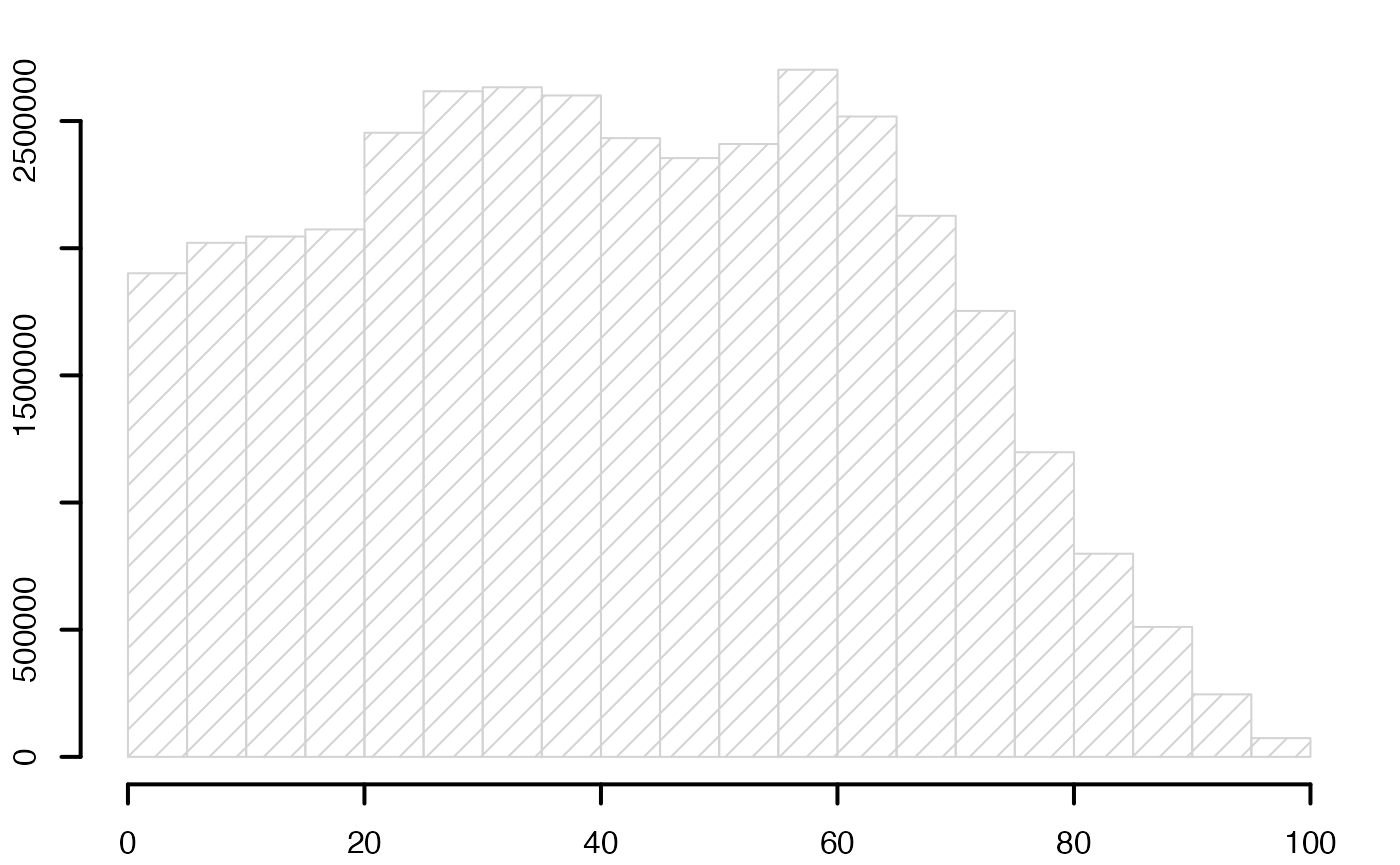
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Sources/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histoeffnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=limites,main="",freq=T,xlab="",ylab="",lwd=2,density=10)
dev.off()
#> agg_png
#> 2
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,density=10)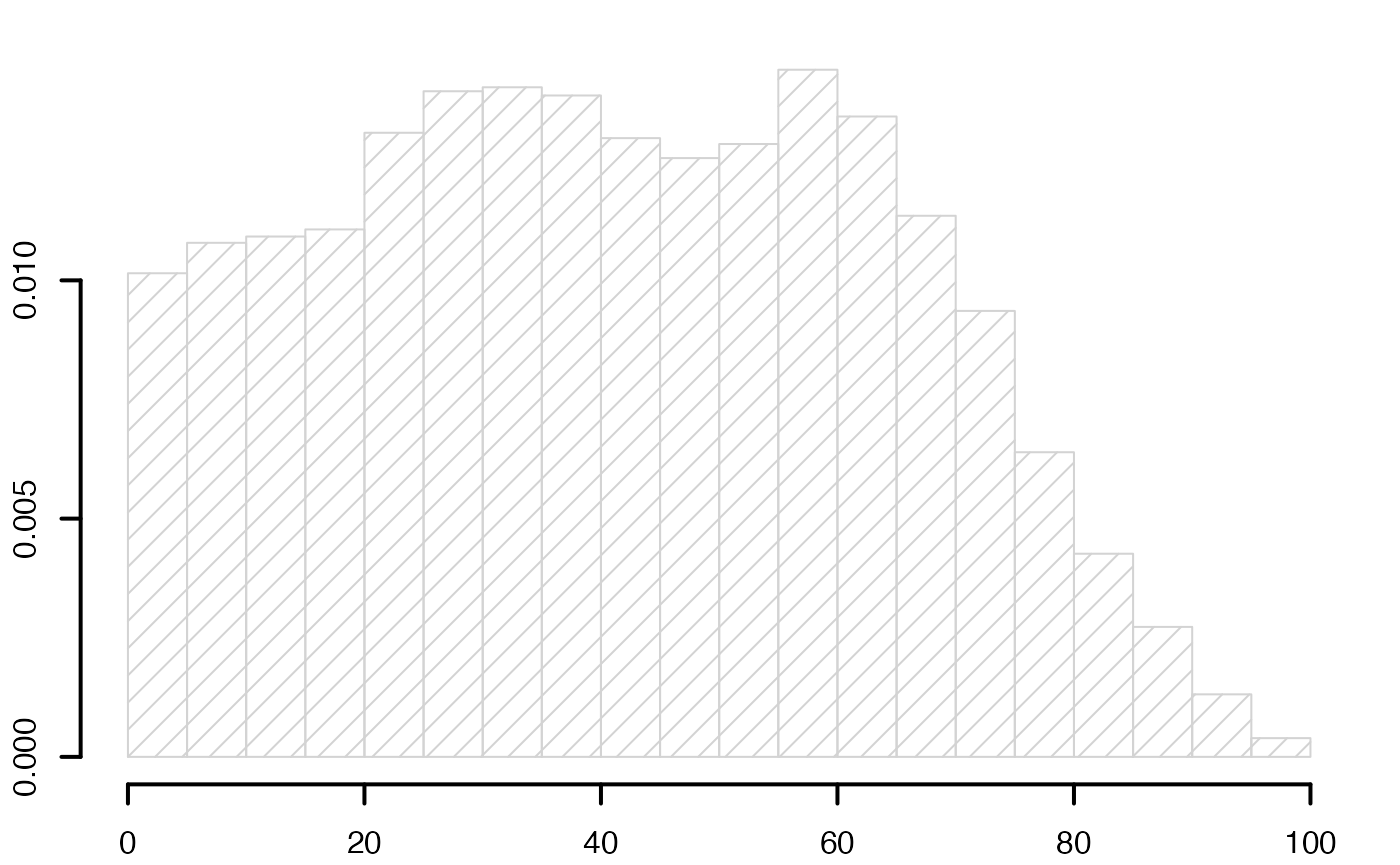
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histofrenew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
dev.off()
#> agg_png
#> 2
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-length(histofre$mids)],histofre$density[-length(histofre$density)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")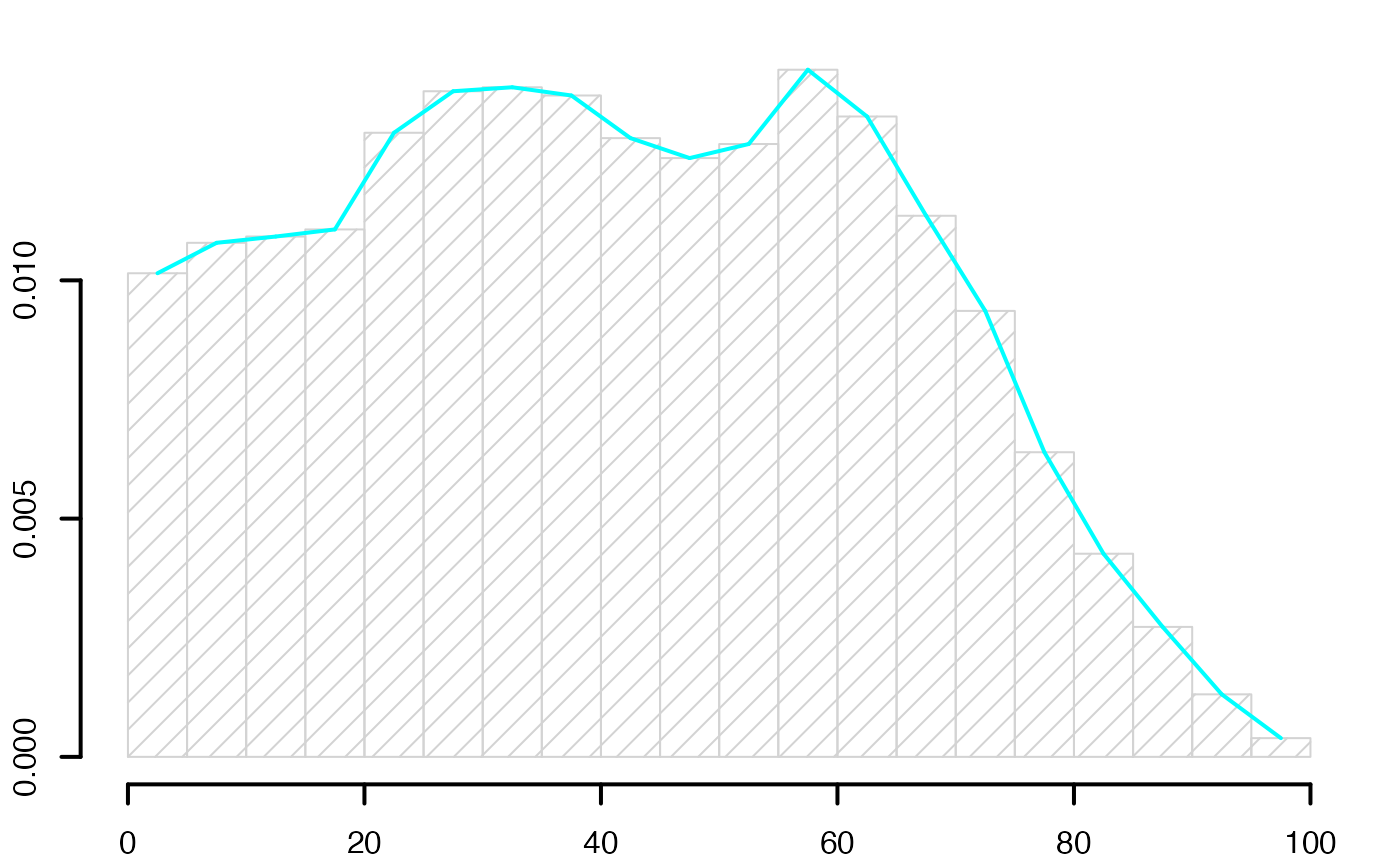
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrenew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-length(histofre$mids)],histofre$density[-length(histofre$density)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2
nihistsum <- rep(0,length(valeurs))
for (iii in 2:(length(valeurs)+1))
{
nihistsum[iii] <- nihistsum[iii-1]+(valeurs)[iii-1]
}
data4 <- rep(centres,cumsum(valeurs))
table(data4)
#> data4
#> 2.5 7.5 12.5 17.5 22.5 27.5 32.5 37.5
#> 1901542 3922769 5968683 8042554 10496446 13113610 15746391 18346920
#> 42.5 47.5 52.5 57.5 62.5 67.5 72.5 77.5
#> 20779741 23134103 25543804 28245595 30763497 32891054 34644497 35842342
#> 82.5 87.5 92.5 97.5
#> 36641095 37152143 37398021 37471746
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=limites,main="",freq=T,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$counts),labels=format(c(0,.2,.4,.6,.8,1)*max(histofrecum$counts),scientific = TRUE,digits = 3),lwd=2)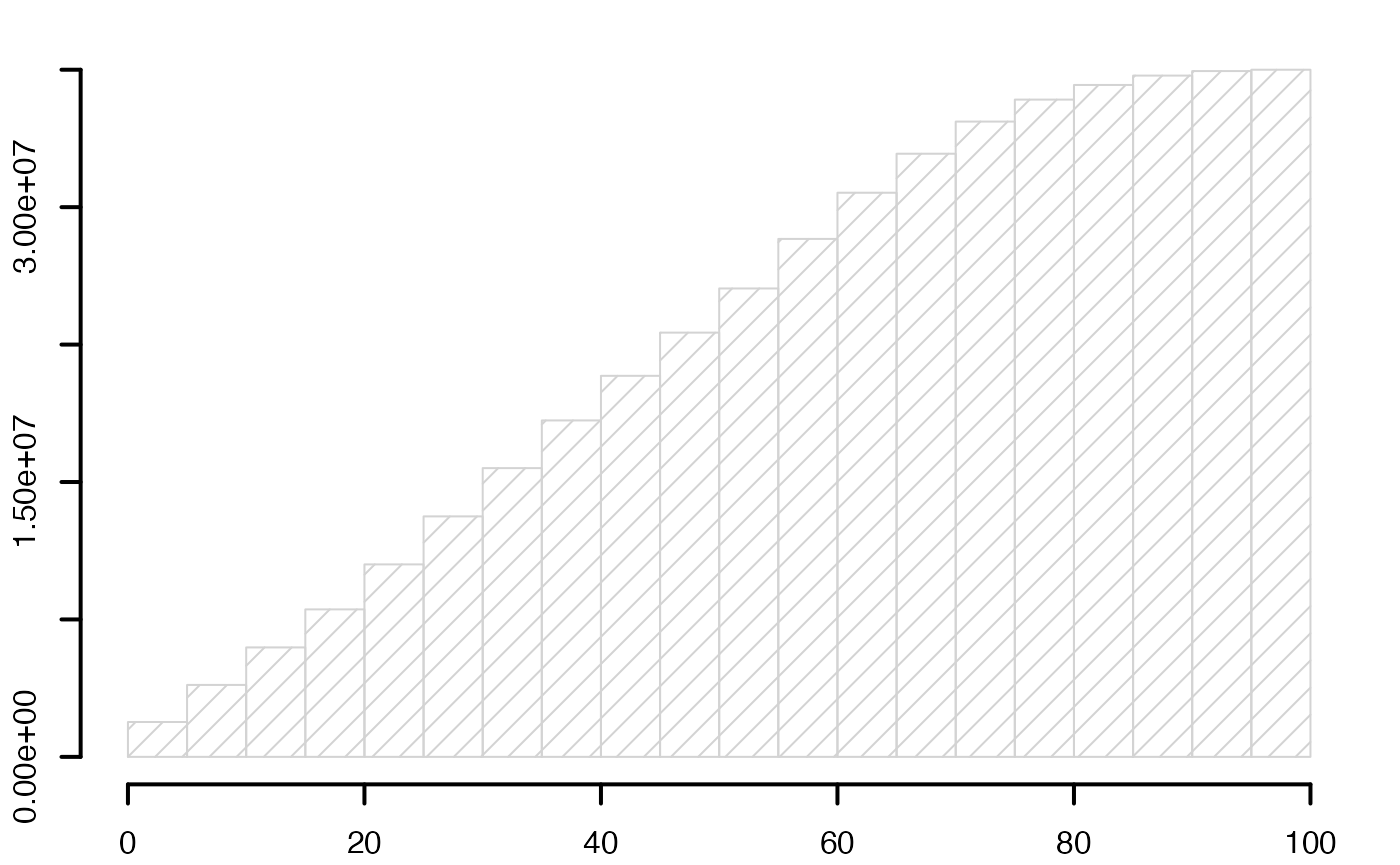
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histoeffcumnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=limites,main="",freq=T,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$counts),labels=round(c(0,.2,.4,.6,.8,1)*max(histofrecum$counts)),lwd=2)
dev.off()
#> agg_png
#> 2
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)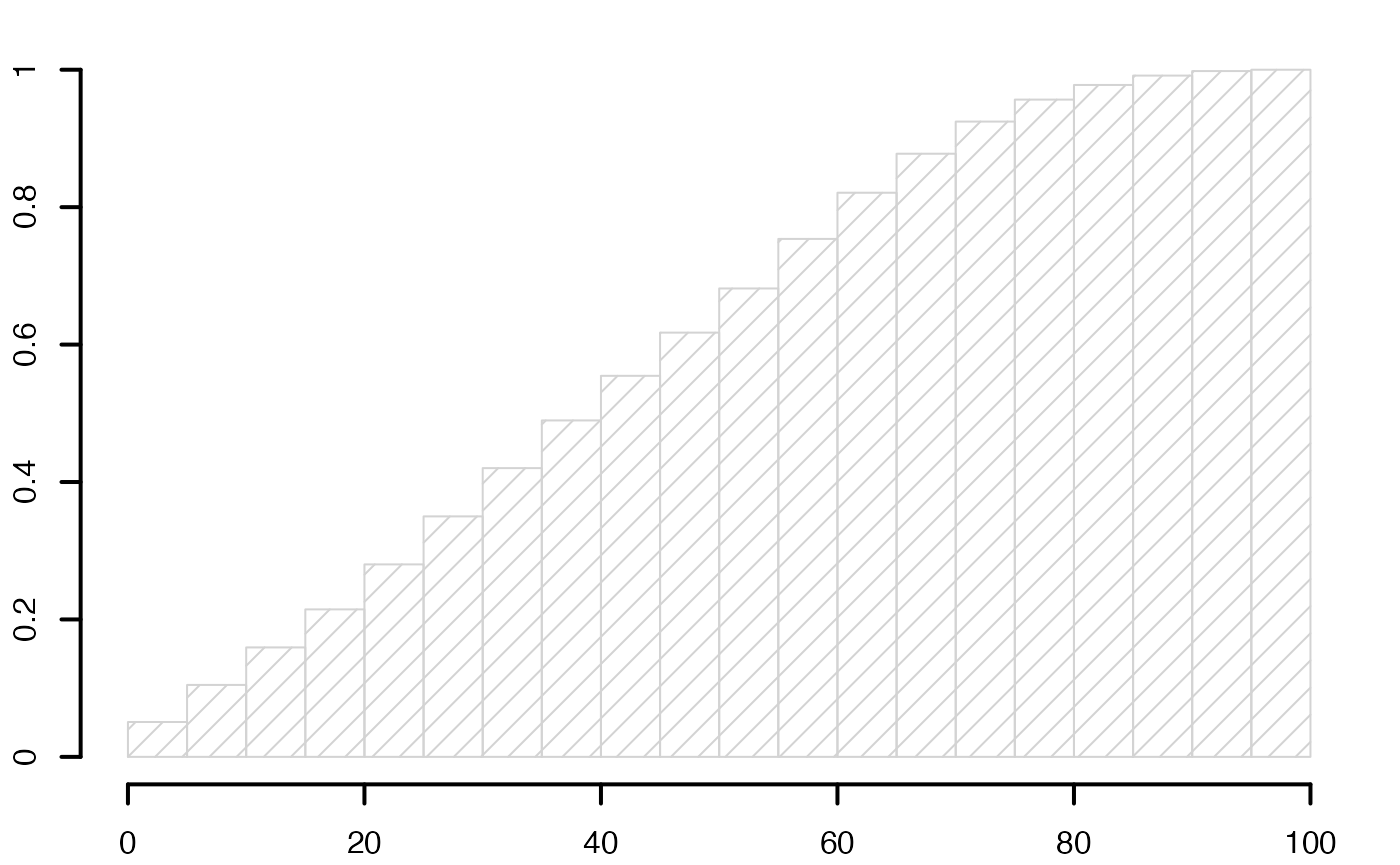
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histofrecumnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-length(histofrecum$mids)],histofrecum$density[-length(histofrecum$mids)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)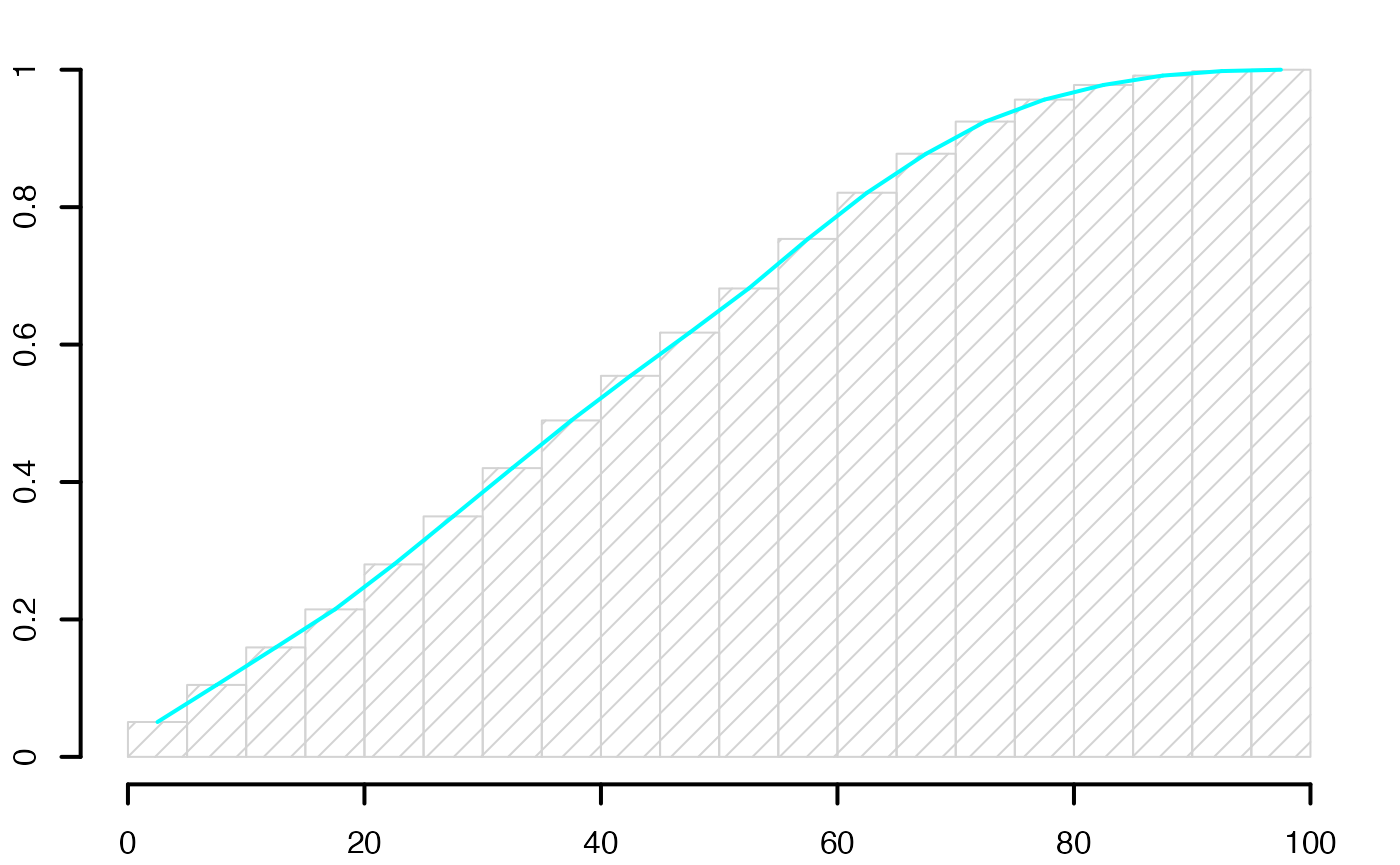
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrecumnew.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=limites,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-length(histofrecum$mids)],histofrecum$density[-length(histofrecum$mids)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Bonus : Sièges Voix
data(Sieges_Voix)
str(Sieges_Voix)
#> 'data.frame': 27 obs. of 4 variables:
#> $ Etats.Membres : chr "Allemagne" "Autriche" "Belgique" "Bulgarie" ...
#> $ Date.entrée : int 1957 1995 1957 2007 2004 1973 1986 2004 1995 1957 ...
#> $ Sièges.au.parlement: int 99 18 24 0 6 14 54 6 14 78 ...
#> $ Voix.au.conseil : int 29 10 12 0 4 7 27 4 7 29 ...
summary(Sieges_Voix)
#> Etats.Membres Date.entrée Sièges.au.parlement Voix.au.conseil
#> Length:27 Min. :1957 Min. : 0.00 Min. : 0.00
#> Class :character 1st Qu.:1973 1st Qu.: 8.00 1st Qu.: 4.00
#> Mode :character Median :1995 Median :18.00 Median :10.00
#> Mean :1987 Mean :27.11 Mean :11.89
#> 3rd Qu.:2004 3rd Qu.:25.50 3rd Qu.:12.50
#> Max. :2007 Max. :99.00 Max. :29.00
NameX <- Sieges_Voix$Etats.Membres
NombreSieges <- Sieges_Voix$Sièges.au.parlement
NombreVoix <- Sieges_Voix$Voix.au.conseil
NameXX <- names(table(Sieges_Voix$Date.entrée))
NombreAnnees <- as.vector(table(Sieges_Voix$Date.entrée))
names(NombreSieges) <- NameX
NombreSieges
#> Allemagne Autriche Belgique Bulgarie
#> 99 18 24 0
#> Chypre Danemark Espagne Estonie
#> 6 14 54 6
#> Finlande France Grèce Hongrie
#> 14 78 24 24
#> Irlande Italie Lettonie Lituanie
#> 13 78 9 13
#> Luxembourg Malte Pays-Bas Pologne
#> 6 5 27 54
#> Portugal République Tchèque Roumanie Royaume-Uni
#> 24 24 0 78
#> Slovaquie Slovénie Suède
#> 14 7 19
rep(NameX,NombreSieges)
#> [1] "Allemagne" "Allemagne" "Allemagne"
#> [4] "Allemagne" "Allemagne" "Allemagne"
#> [7] "Allemagne" "Allemagne" "Allemagne"
#> [10] "Allemagne" "Allemagne" "Allemagne"
#> [13] "Allemagne" "Allemagne" "Allemagne"
#> [16] "Allemagne" "Allemagne" "Allemagne"
#> [19] "Allemagne" "Allemagne" "Allemagne"
#> [22] "Allemagne" "Allemagne" "Allemagne"
#> [25] "Allemagne" "Allemagne" "Allemagne"
#> [28] "Allemagne" "Allemagne" "Allemagne"
#> [31] "Allemagne" "Allemagne" "Allemagne"
#> [34] "Allemagne" "Allemagne" "Allemagne"
#> [37] "Allemagne" "Allemagne" "Allemagne"
#> [40] "Allemagne" "Allemagne" "Allemagne"
#> [43] "Allemagne" "Allemagne" "Allemagne"
#> [46] "Allemagne" "Allemagne" "Allemagne"
#> [49] "Allemagne" "Allemagne" "Allemagne"
#> [52] "Allemagne" "Allemagne" "Allemagne"
#> [55] "Allemagne" "Allemagne" "Allemagne"
#> [58] "Allemagne" "Allemagne" "Allemagne"
#> [61] "Allemagne" "Allemagne" "Allemagne"
#> [64] "Allemagne" "Allemagne" "Allemagne"
#> [67] "Allemagne" "Allemagne" "Allemagne"
#> [70] "Allemagne" "Allemagne" "Allemagne"
#> [73] "Allemagne" "Allemagne" "Allemagne"
#> [76] "Allemagne" "Allemagne" "Allemagne"
#> [79] "Allemagne" "Allemagne" "Allemagne"
#> [82] "Allemagne" "Allemagne" "Allemagne"
#> [85] "Allemagne" "Allemagne" "Allemagne"
#> [88] "Allemagne" "Allemagne" "Allemagne"
#> [91] "Allemagne" "Allemagne" "Allemagne"
#> [94] "Allemagne" "Allemagne" "Allemagne"
#> [97] "Allemagne" "Allemagne" "Allemagne"
#> [100] "Autriche" "Autriche" "Autriche"
#> [103] "Autriche" "Autriche" "Autriche"
#> [106] "Autriche" "Autriche" "Autriche"
#> [109] "Autriche" "Autriche" "Autriche"
#> [112] "Autriche" "Autriche" "Autriche"
#> [115] "Autriche" "Autriche" "Autriche"
#> [118] "Belgique" "Belgique" "Belgique"
#> [121] "Belgique" "Belgique" "Belgique"
#> [124] "Belgique" "Belgique" "Belgique"
#> [127] "Belgique" "Belgique" "Belgique"
#> [130] "Belgique" "Belgique" "Belgique"
#> [133] "Belgique" "Belgique" "Belgique"
#> [136] "Belgique" "Belgique" "Belgique"
#> [139] "Belgique" "Belgique" "Belgique"
#> [142] "Chypre" "Chypre" "Chypre"
#> [145] "Chypre" "Chypre" "Chypre"
#> [148] "Danemark" "Danemark" "Danemark"
#> [151] "Danemark" "Danemark" "Danemark"
#> [154] "Danemark" "Danemark" "Danemark"
#> [157] "Danemark" "Danemark" "Danemark"
#> [160] "Danemark" "Danemark" "Espagne"
#> [163] "Espagne" "Espagne" "Espagne"
#> [166] "Espagne" "Espagne" "Espagne"
#> [169] "Espagne" "Espagne" "Espagne"
#> [172] "Espagne" "Espagne" "Espagne"
#> [175] "Espagne" "Espagne" "Espagne"
#> [178] "Espagne" "Espagne" "Espagne"
#> [181] "Espagne" "Espagne" "Espagne"
#> [184] "Espagne" "Espagne" "Espagne"
#> [187] "Espagne" "Espagne" "Espagne"
#> [190] "Espagne" "Espagne" "Espagne"
#> [193] "Espagne" "Espagne" "Espagne"
#> [196] "Espagne" "Espagne" "Espagne"
#> [199] "Espagne" "Espagne" "Espagne"
#> [202] "Espagne" "Espagne" "Espagne"
#> [205] "Espagne" "Espagne" "Espagne"
#> [208] "Espagne" "Espagne" "Espagne"
#> [211] "Espagne" "Espagne" "Espagne"
#> [214] "Espagne" "Espagne" "Estonie"
#> [217] "Estonie" "Estonie" "Estonie"
#> [220] "Estonie" "Estonie" "Finlande"
#> [223] "Finlande" "Finlande" "Finlande"
#> [226] "Finlande" "Finlande" "Finlande"
#> [229] "Finlande" "Finlande" "Finlande"
#> [232] "Finlande" "Finlande" "Finlande"
#> [235] "Finlande" "France" "France"
#> [238] "France" "France" "France"
#> [241] "France" "France" "France"
#> [244] "France" "France" "France"
#> [247] "France" "France" "France"
#> [250] "France" "France" "France"
#> [253] "France" "France" "France"
#> [256] "France" "France" "France"
#> [259] "France" "France" "France"
#> [262] "France" "France" "France"
#> [265] "France" "France" "France"
#> [268] "France" "France" "France"
#> [271] "France" "France" "France"
#> [274] "France" "France" "France"
#> [277] "France" "France" "France"
#> [280] "France" "France" "France"
#> [283] "France" "France" "France"
#> [286] "France" "France" "France"
#> [289] "France" "France" "France"
#> [292] "France" "France" "France"
#> [295] "France" "France" "France"
#> [298] "France" "France" "France"
#> [301] "France" "France" "France"
#> [304] "France" "France" "France"
#> [307] "France" "France" "France"
#> [310] "France" "France" "France"
#> [313] "France" "Grèce" "Grèce"
#> [316] "Grèce" "Grèce" "Grèce"
#> [319] "Grèce" "Grèce" "Grèce"
#> [322] "Grèce" "Grèce" "Grèce"
#> [325] "Grèce" "Grèce" "Grèce"
#> [328] "Grèce" "Grèce" "Grèce"
#> [331] "Grèce" "Grèce" "Grèce"
#> [334] "Grèce" "Grèce" "Grèce"
#> [337] "Grèce" "Hongrie" "Hongrie"
#> [340] "Hongrie" "Hongrie" "Hongrie"
#> [343] "Hongrie" "Hongrie" "Hongrie"
#> [346] "Hongrie" "Hongrie" "Hongrie"
#> [349] "Hongrie" "Hongrie" "Hongrie"
#> [352] "Hongrie" "Hongrie" "Hongrie"
#> [355] "Hongrie" "Hongrie" "Hongrie"
#> [358] "Hongrie" "Hongrie" "Hongrie"
#> [361] "Hongrie" "Irlande" "Irlande"
#> [364] "Irlande" "Irlande" "Irlande"
#> [367] "Irlande" "Irlande" "Irlande"
#> [370] "Irlande" "Irlande" "Irlande"
#> [373] "Irlande" "Irlande" "Italie"
#> [376] "Italie" "Italie" "Italie"
#> [379] "Italie" "Italie" "Italie"
#> [382] "Italie" "Italie" "Italie"
#> [385] "Italie" "Italie" "Italie"
#> [388] "Italie" "Italie" "Italie"
#> [391] "Italie" "Italie" "Italie"
#> [394] "Italie" "Italie" "Italie"
#> [397] "Italie" "Italie" "Italie"
#> [400] "Italie" "Italie" "Italie"
#> [403] "Italie" "Italie" "Italie"
#> [406] "Italie" "Italie" "Italie"
#> [409] "Italie" "Italie" "Italie"
#> [412] "Italie" "Italie" "Italie"
#> [415] "Italie" "Italie" "Italie"
#> [418] "Italie" "Italie" "Italie"
#> [421] "Italie" "Italie" "Italie"
#> [424] "Italie" "Italie" "Italie"
#> [427] "Italie" "Italie" "Italie"
#> [430] "Italie" "Italie" "Italie"
#> [433] "Italie" "Italie" "Italie"
#> [436] "Italie" "Italie" "Italie"
#> [439] "Italie" "Italie" "Italie"
#> [442] "Italie" "Italie" "Italie"
#> [445] "Italie" "Italie" "Italie"
#> [448] "Italie" "Italie" "Italie"
#> [451] "Italie" "Italie" "Lettonie"
#> [454] "Lettonie" "Lettonie" "Lettonie"
#> [457] "Lettonie" "Lettonie" "Lettonie"
#> [460] "Lettonie" "Lettonie" "Lituanie"
#> [463] "Lituanie" "Lituanie" "Lituanie"
#> [466] "Lituanie" "Lituanie" "Lituanie"
#> [469] "Lituanie" "Lituanie" "Lituanie"
#> [472] "Lituanie" "Lituanie" "Lituanie"
#> [475] "Luxembourg" "Luxembourg" "Luxembourg"
#> [478] "Luxembourg" "Luxembourg" "Luxembourg"
#> [481] "Malte" "Malte" "Malte"
#> [484] "Malte" "Malte" "Pays-Bas"
#> [487] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [490] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [493] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [496] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [499] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [502] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [505] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [508] "Pays-Bas" "Pays-Bas" "Pays-Bas"
#> [511] "Pays-Bas" "Pays-Bas" "Pologne"
#> [514] "Pologne" "Pologne" "Pologne"
#> [517] "Pologne" "Pologne" "Pologne"
#> [520] "Pologne" "Pologne" "Pologne"
#> [523] "Pologne" "Pologne" "Pologne"
#> [526] "Pologne" "Pologne" "Pologne"
#> [529] "Pologne" "Pologne" "Pologne"
#> [532] "Pologne" "Pologne" "Pologne"
#> [535] "Pologne" "Pologne" "Pologne"
#> [538] "Pologne" "Pologne" "Pologne"
#> [541] "Pologne" "Pologne" "Pologne"
#> [544] "Pologne" "Pologne" "Pologne"
#> [547] "Pologne" "Pologne" "Pologne"
#> [550] "Pologne" "Pologne" "Pologne"
#> [553] "Pologne" "Pologne" "Pologne"
#> [556] "Pologne" "Pologne" "Pologne"
#> [559] "Pologne" "Pologne" "Pologne"
#> [562] "Pologne" "Pologne" "Pologne"
#> [565] "Pologne" "Pologne" "Portugal"
#> [568] "Portugal" "Portugal" "Portugal"
#> [571] "Portugal" "Portugal" "Portugal"
#> [574] "Portugal" "Portugal" "Portugal"
#> [577] "Portugal" "Portugal" "Portugal"
#> [580] "Portugal" "Portugal" "Portugal"
#> [583] "Portugal" "Portugal" "Portugal"
#> [586] "Portugal" "Portugal" "Portugal"
#> [589] "Portugal" "Portugal" "République Tchèque"
#> [592] "République Tchèque" "République Tchèque" "République Tchèque"
#> [595] "République Tchèque" "République Tchèque" "République Tchèque"
#> [598] "République Tchèque" "République Tchèque" "République Tchèque"
#> [601] "République Tchèque" "République Tchèque" "République Tchèque"
#> [604] "République Tchèque" "République Tchèque" "République Tchèque"
#> [607] "République Tchèque" "République Tchèque" "République Tchèque"
#> [610] "République Tchèque" "République Tchèque" "République Tchèque"
#> [613] "République Tchèque" "République Tchèque" "Royaume-Uni"
#> [616] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [619] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [622] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [625] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [628] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [631] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [634] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [637] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [640] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [643] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [646] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [649] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [652] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [655] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [658] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [661] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [664] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [667] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [670] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [673] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [676] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [679] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [682] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [685] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [688] "Royaume-Uni" "Royaume-Uni" "Royaume-Uni"
#> [691] "Royaume-Uni" "Royaume-Uni" "Slovaquie"
#> [694] "Slovaquie" "Slovaquie" "Slovaquie"
#> [697] "Slovaquie" "Slovaquie" "Slovaquie"
#> [700] "Slovaquie" "Slovaquie" "Slovaquie"
#> [703] "Slovaquie" "Slovaquie" "Slovaquie"
#> [706] "Slovaquie" "Slovénie" "Slovénie"
#> [709] "Slovénie" "Slovénie" "Slovénie"
#> [712] "Slovénie" "Slovénie" "Suède"
#> [715] "Suède" "Suède" "Suède"
#> [718] "Suède" "Suède" "Suède"
#> [721] "Suède" "Suède" "Suède"
#> [724] "Suède" "Suède" "Suède"
#> [727] "Suède" "Suède" "Suède"
#> [730] "Suède" "Suède" "Suède"
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(NombreSieges,labels=NameX,pch=19,col="#00FFFF")
dotchart3(NombreSieges,labels=NameX,pch=19,col="#00FFFF",cex=1.6,cex.axis=.8)
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"Sieges_pointshoriz_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(NombreSieges/sum(NombreSieges),labels=NameX,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dev.off()
#> agg_png
#> 2
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(NombreVoix,labels=NameX,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dotchart3(NombreVoix,labels=NameX,pch=19,col="#00FFFF")
pdf(file = paste(Chemin,"Voix_pointshoriz_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(NombreVoix/sum(NombreVoix),labels=NameX,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dev.off()
#> agg_png
#> 2
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(NombreAnnees,labels=NameXX,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)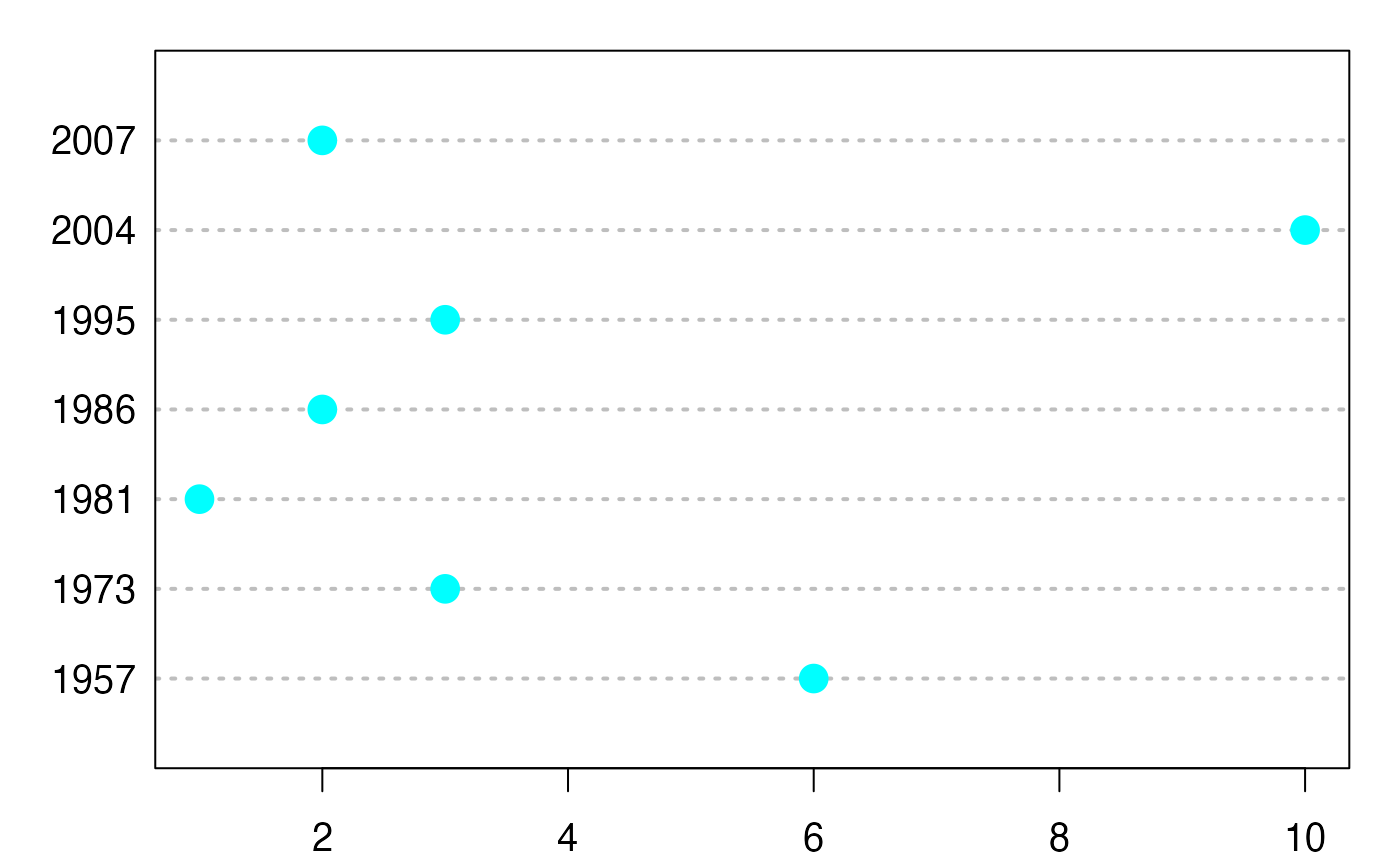
dotchart3(NombreAnnees,labels=NameXX,pch=19,col="#00FFFF")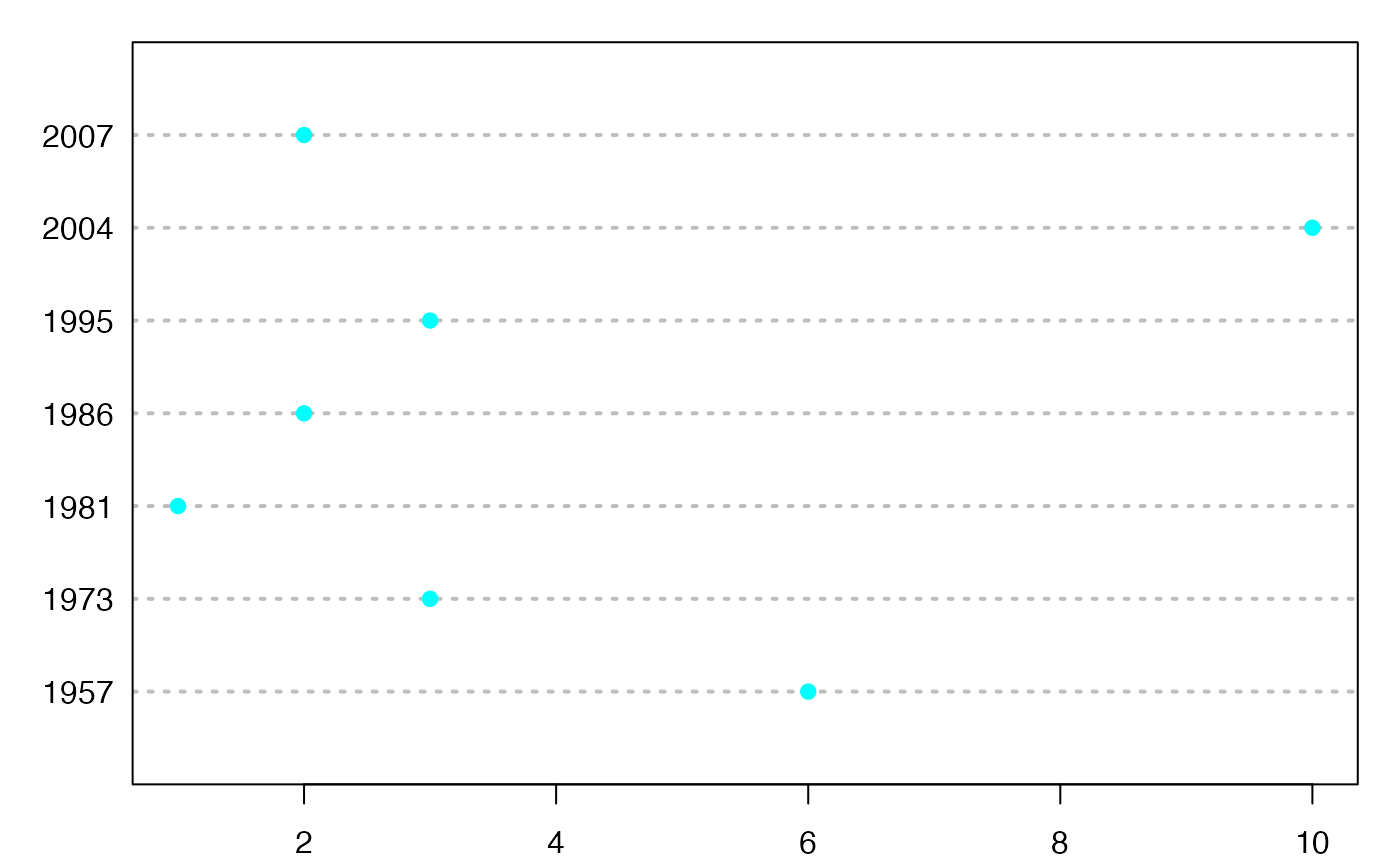
pdf(file = paste(Chemin,"Annees_pointshoriz_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
dotchart3(NombreAnnees/sum(NombreAnnees),labels=NameXX,pch=19,col="#00FFFF",cex=1.6,cex.axis=1.2)
dev.off()
#> agg_png
#> 2
NameXbar <- NameX
par(mar = c(9.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(NombreSieges/sum(NombreSieges),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)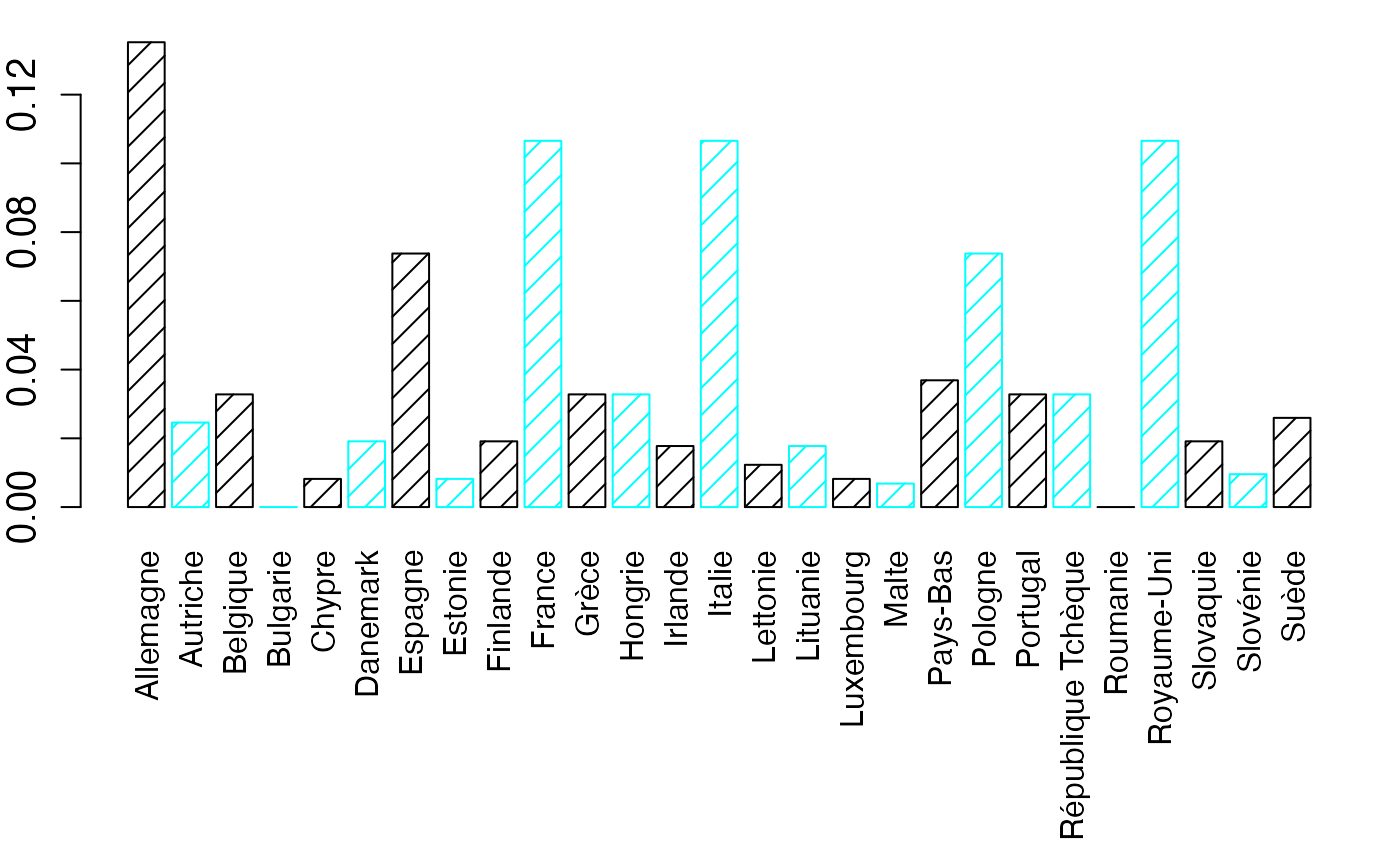
pdf(file = paste(Chemin,"Sieges_barvertibis_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(9.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(NombreSieges/sum(NombreSieges),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)
dev.off()
#> agg_png
#> 2
NameXbar <- NameX
par(mar = c(9.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(NombreVoix/sum(NombreVoix),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)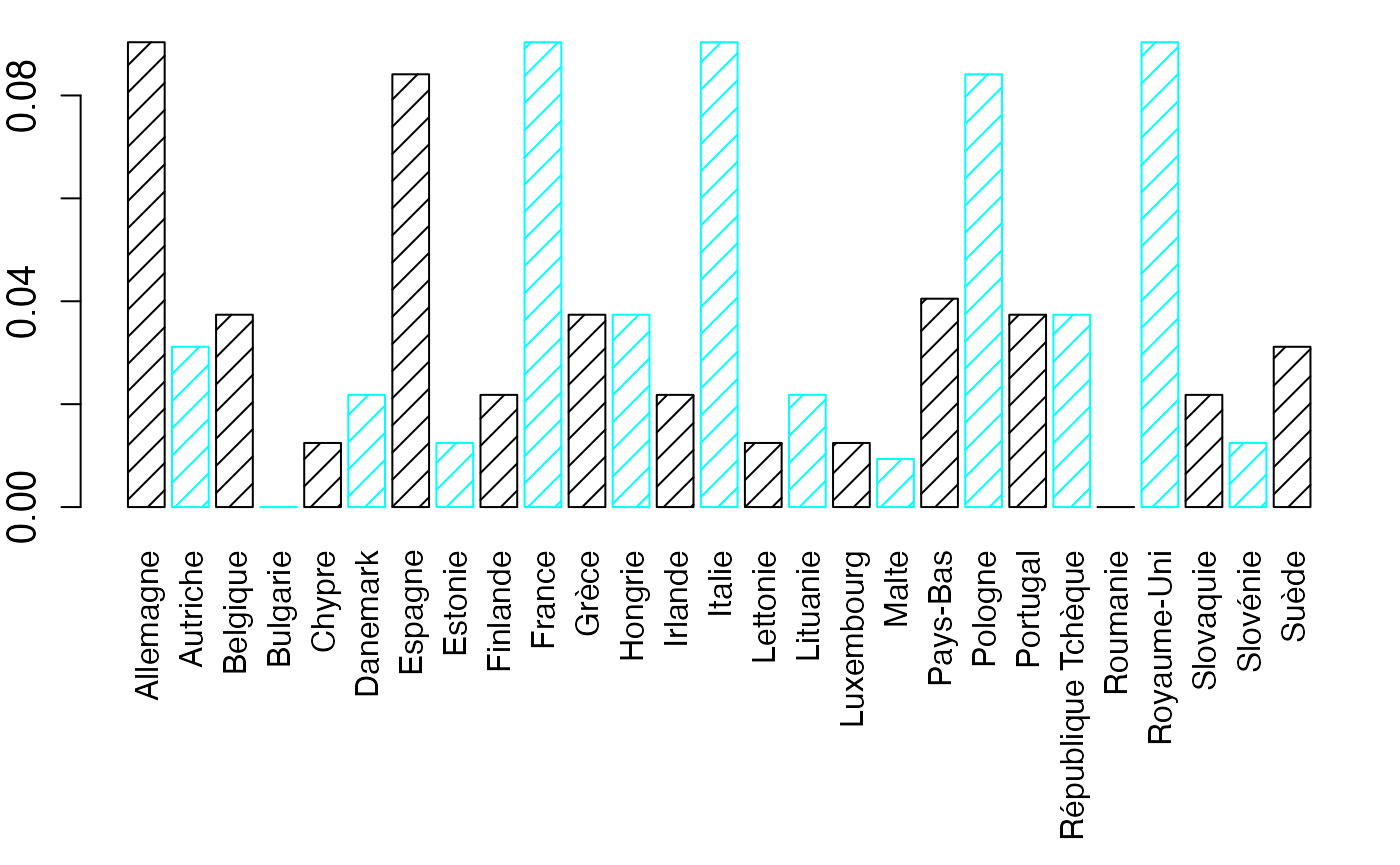
pdf(file = paste(Chemin,"Voix_barvertibis_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(9.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(NombreVoix/sum(NombreVoix),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)
dev.off()
#> agg_png
#> 2
NameXbar <- NameXX
par(mar = c(3.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(NombreAnnees/sum(NombreAnnees),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)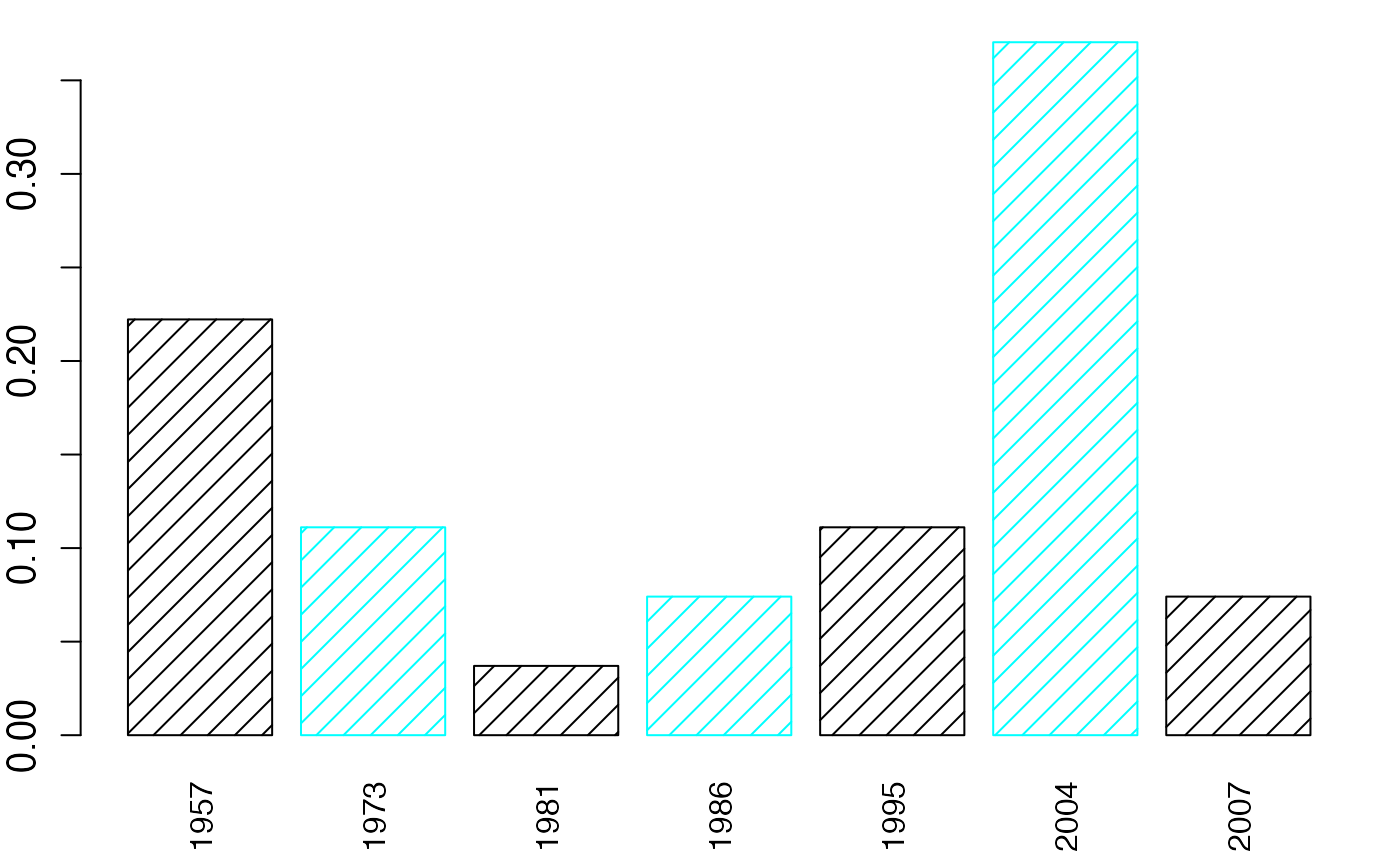
pdf(file = paste(Chemin,"Annees_barvertibis_fre.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(3.1, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
barplot(NombreAnnees/sum(NombreAnnees),names.arg=NameXbar,col=c("black","#00FFFF","black","#00FFFF"),density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex.axis=1.2,las=3)
dev.off()
#> agg_png
#> 2
NameX[22] <- "République\nTchèque"
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(NombreSieges,labels=NameX,col=c("black","#00FFFF","black","#00FFFF"),init.angle = 70,density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1)#,density = c(4*(4:1))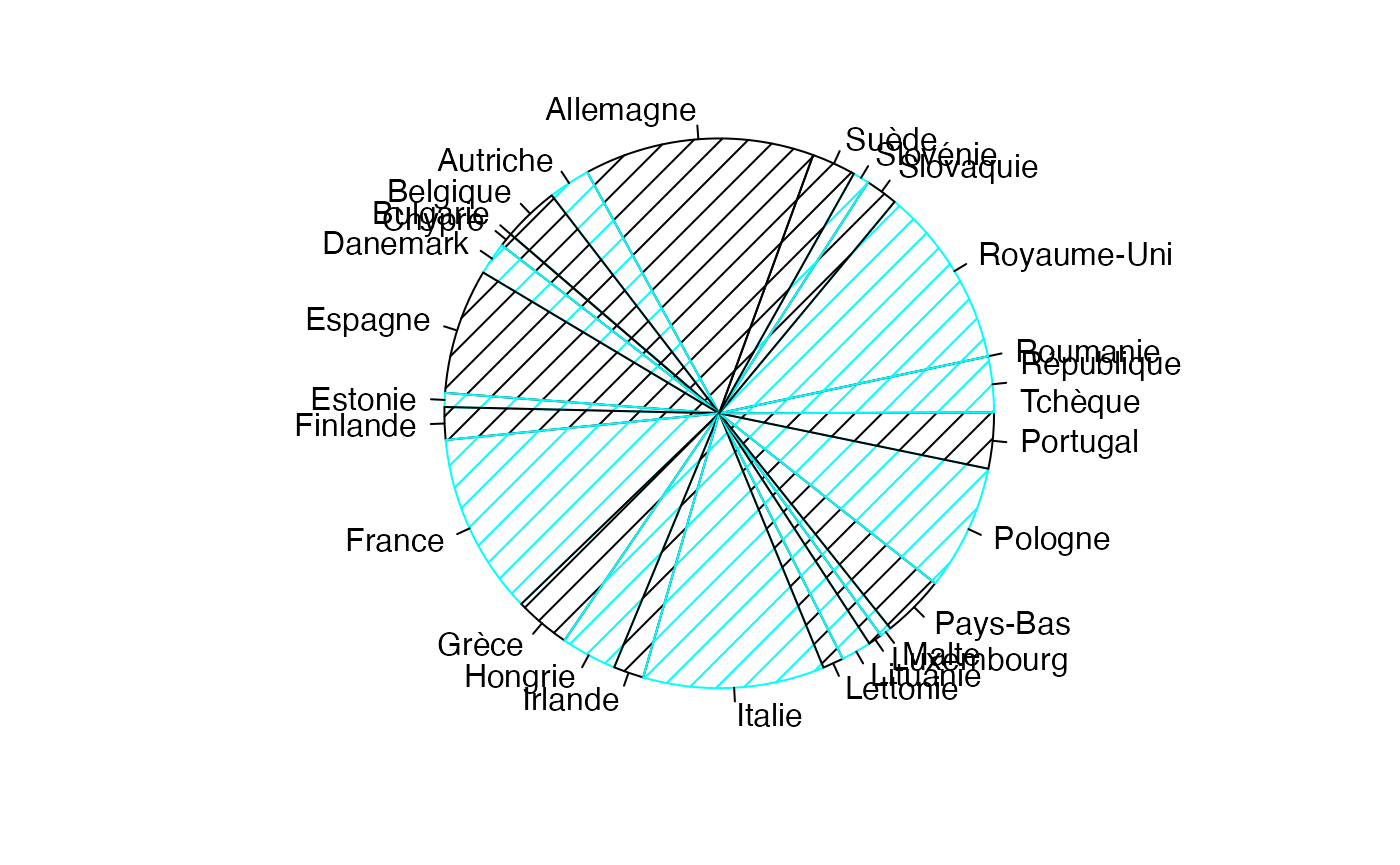
pdf(file = paste(Chemin,"Sieges_diagcircubis.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(NombreSieges,labels=NameX,col=c("black","#00FFFF","black","#00FFFF"),init.angle = 70,density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1)#,density = c(4*(4:1))
dev.off()
#> agg_png
#> 2
NameX <- Sieges_Voix$Etats.Membres
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(NombreVoix,labels=NameX,col=c("black","#00FFFF","black","#00FFFF"),init.angle = 0,density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1)#,density = c(4*(4:1))
pdf(file = paste(Chemin,"Voix_diagcircubis.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(NombreVoix,labels=NameX,col=c("black","#00FFFF","black","#00FFFF"),init.angle = 0,density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1)#,density = c(4*(4:1))
dev.off()
#> agg_png
#> 2
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(NombreAnnees,labels=NameXX,col=c("black","#00FFFF","black","#00FFFF"),init.angle = 0,density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1)#,density = c(4*(4:1))
pdf(file = paste(Chemin,"Annees_diagcircubis.pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
pie(NombreAnnees,labels=NameXX,col=c("black","#00FFFF","black","#00FFFF"),init.angle = 0,density = 10,border=c("black","#00FFFF","black","#00FFFF"),cex=1)#,density = c(4*(4:1))
dev.off()
#> agg_png
#> 2Europe Emploi Salariés
Boîte à moustaches
head(europe)
#> [[1]]
#> NULL
#>
#> $Durée
#> [1] 40.2 41.1 39.1 40.7 41.2 40.4 37.6 39.6 40.2 39.4 39.1 40.7 40.4 39.4 43.4
#> [16] 39.0 40.2 39.9 40.2 41.6 41.3 44.6 38.4 38.9 40.8 40.8 40.6 42.0 42.8 40.5
#> [31] 40.7 39.9 41.8 40.7 48.1
str(europe)
#> List of 2
#> $ : NULL
#> $ Durée: num [1:35] 40.2 41.1 39.1 40.7 41.2 40.4 37.6 39.6 40.2 39.4 ...
summary(europe$Durée)
#> Min. 1st Qu. Median Mean 3rd Qu. Max.
#> 37.60 39.75 40.50 40.72 41.15 48.10
range(europe$Durée)
#> [1] 37.6 48.1
sd(europe$Durée)
#> [1] 1.873984
qts <- quantile(europe$Durée,c(.25,.5,.75),type=6)
ltms <- c(qts[1]-3/2*(qts[3]-qts[1]),qts[3]+3/2*(qts[3]-qts[1]))
lms <- c(min(europe$Durée[europe$Durée>=qts[1]-3/2*(qts[3]-qts[1])]),max(europe$Durée[europe$Durée<=qts[3]+3/2*(qts[3]-qts[1])]))
conf <- qts[2] + c(-1.58, 1.58) * (qts[3]-qts[1])/sqrt(sum(!is.na(europe$Durée)))
extremes <- c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
)
#bltr
oldpar <- par()
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="Durée (heures)",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(rep(1,length(extremes)),extremes,pch=8,cex=1.25,lwd=2.5)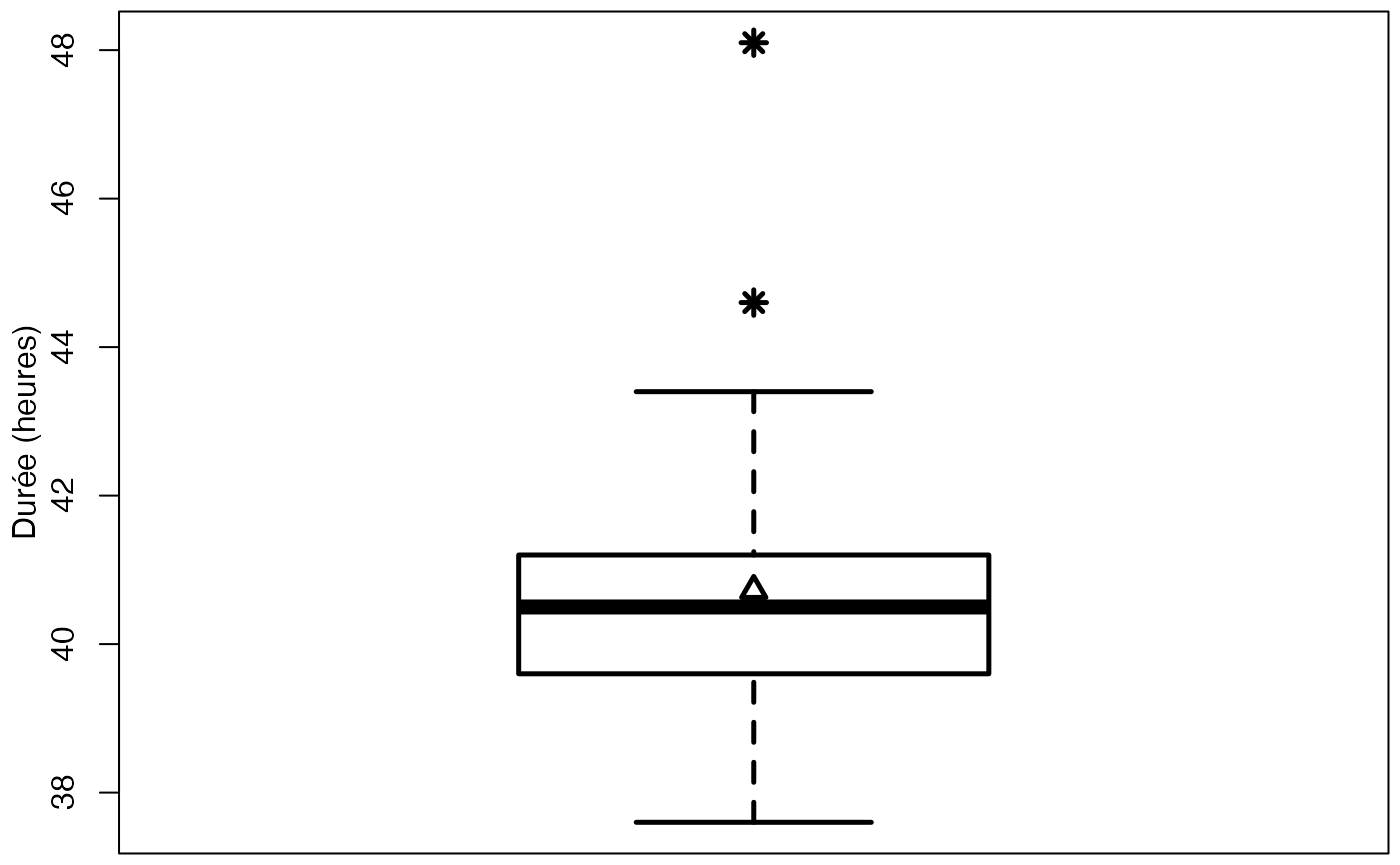
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"boxplot2019v3.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "europeSalaries boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="Durée (heures)",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(rep(1,length(extremes)),extremes,pch=8,cex=1.25,lwd=2.5)
dev.off()
#> agg_png
#> 2
#suppressWarnings(par(oldpar))Statistiques descriptives
mean(europe$Durée)
#> [1] 40.72286
quantile(europe$Durée,probs=c(.25,.5,.75),type=6)
#> 25% 50% 75%
#> 39.6 40.5 41.2
range(europe$Durée)
#> [1] 37.6 48.1
diff(quantile(europe$Durée,probs=c(.25,.75),type=6))
#> 75%
#> 1.6
diff(range(europe$Durée))
#> [1] 10.5
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2)
#> [1] 3.411478
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))
#> [1] 1.847019
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))/mean(europe$Durée)*100
#> [1] 4.535582
mean(sort(abs(europe$Durée-mean(sort(europe$Durée)[c(14)])))[c(14)])
#> [1] 0.5
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2))
#> [1] 1.912403
(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2)))^2
#> [1] 3.657286
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))
#> [1] 8.419112
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))-3
#> [1] 5.419112Europe Emploi Non-Salariés
Boîte à moustaches
head(europe)
#> [[1]]
#> NULL
#>
#> $Durée
#> [1] 47.6 51.2 52.5 43.0 44.9 44.6 46.3 47.0 43.2 46.4 50.1 50.4 41.5 49.4 48.5
#> [16] 46.0 41.5 40.6 46.9 45.3 47.4 48.3 45.0 47.9 45.7 47.6 38.9 45.2 51.2 45.1
#> [31] 45.7 47.9 49.4 45.9 49.6
str(europe)
#> List of 2
#> $ : NULL
#> $ Durée: num [1:35] 47.6 51.2 52.5 43 44.9 44.6 46.3 47 43.2 46.4 ...
summary(europe$Durée)
#> Min. 1st Qu. Median Mean 3rd Qu. Max.
#> 38.90 45.05 46.40 46.51 48.40 52.50
range(europe$Durée)
#> [1] 38.9 52.5
sd(europe$Durée)
#> [1] 3.118912
qts <- quantile(europe$Durée,c(.25,.5,.75),type=6)
ltms <- c(qts[1]-3/2*(qts[3]-qts[1]),qts[3]+3/2*(qts[3]-qts[1]))
lms <- c(min(europe$Durée[europe$Durée>=qts[1]-3/2*(qts[3]-qts[1])]),max(europe$Durée[europe$Durée<=qts[3]+3/2*(qts[3]-qts[1])]))
conf <- qts[2] + c(-1.58, 1.58) * (qts[3]-qts[1])/sqrt(sum(!is.na(europe$Durée)))
extremes <- c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
)
#bltr
oldpar <- par()
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="Durée (heures)",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(rep(1,length(extremes)),extremes,pch=8,cex=1.25,lwd=2.5)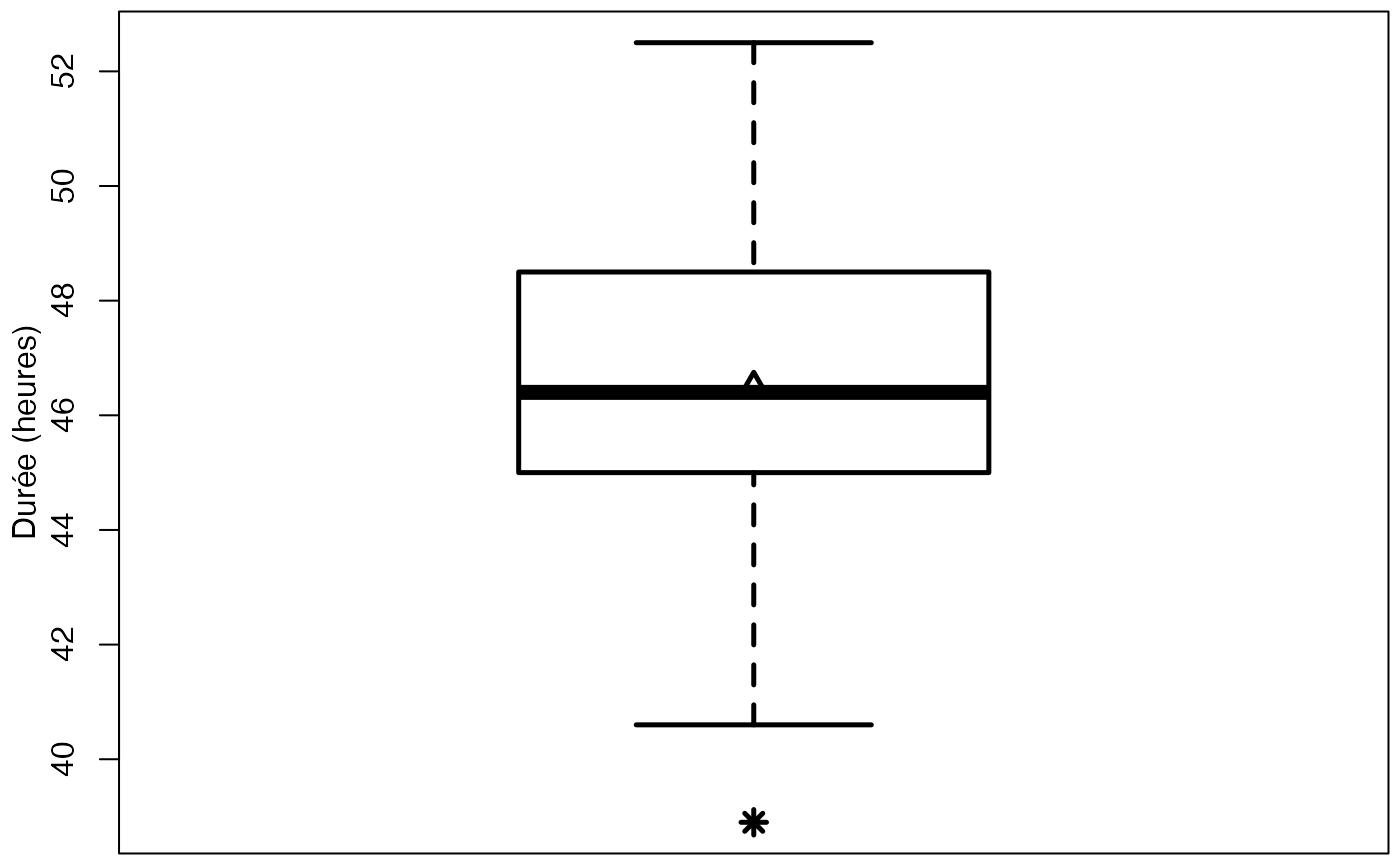
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"boxplot2019v3_app2.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "europeSalaries boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="Durée (heures)",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(rep(1,length(extremes)),extremes,pch=8,cex=1.25,lwd=2.5)
dev.off()
#> agg_png
#> 2
#suppressWarnings(par(oldpar))Statistiques descriptives
mean(europe$Durée)
#> [1] 46.50571
quantile(europe$Durée,probs=c(.25,.5,.75),type=6)
#> 25% 50% 75%
#> 45.0 46.4 48.5
range(europe$Durée)
#> [1] 38.9 52.5
diff(quantile(europe$Durée,probs=c(.25,.75),type=6))
#> 75%
#> 3.5
diff(range(europe$Durée))
#> [1] 13.6
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2)
#> [1] 9.449682
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))
#> [1] 3.074033
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))/mean(europe$Durée)*100
#> [1] 6.610012
mean(sort(abs(europe$Durée-mean(sort(europe$Durée)[c(14)])))[c(14)])
#> [1] 1.3
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2))
#> [1] -0.3640395
(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2)))^2
#> [1] 0.1325248
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))
#> [1] 2.896399
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))-3
#> [1] -0.1036008Europe Emploi Partiel_Ens
data(Europe)
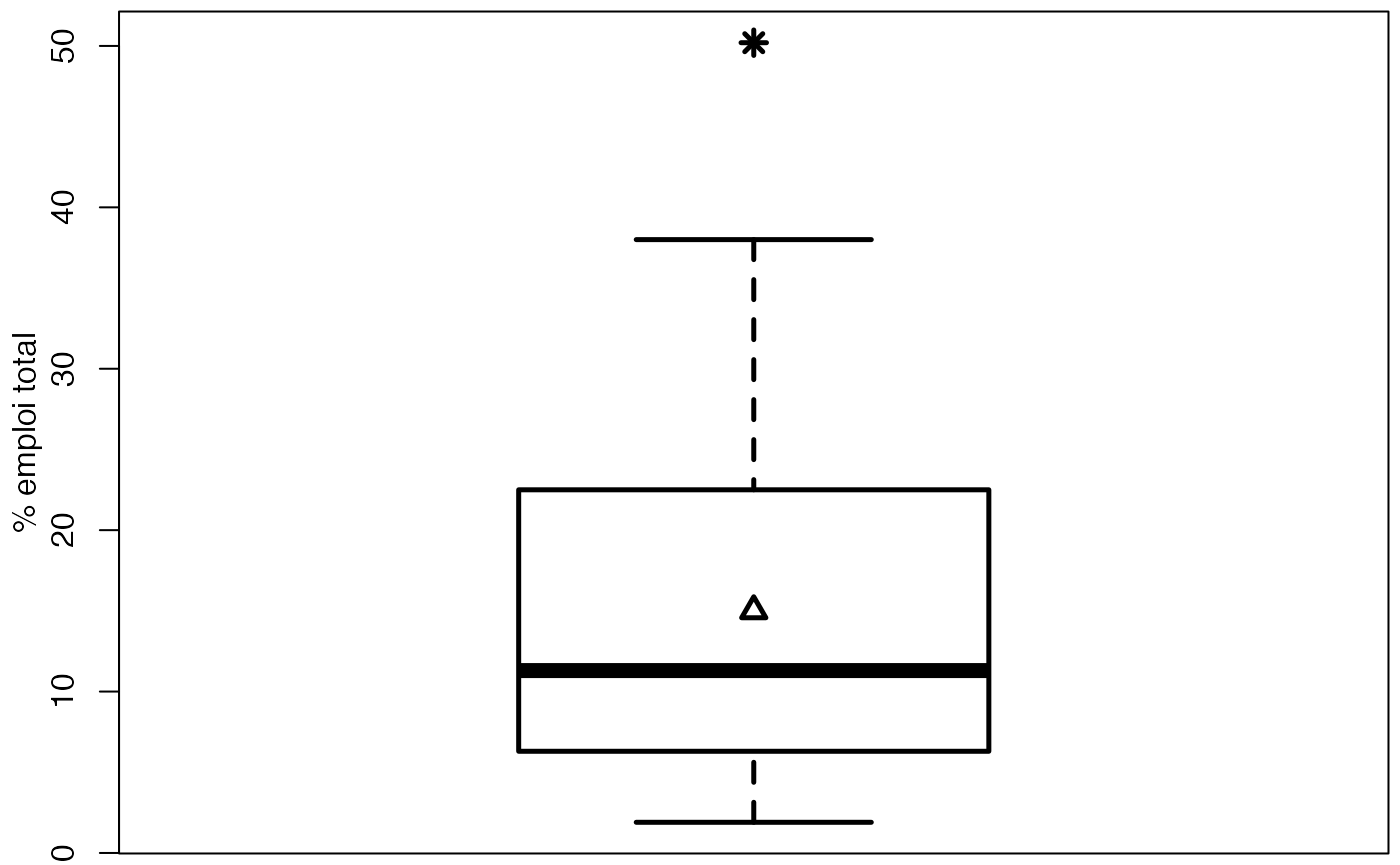
europe <- vector("list",1)
europe$Durée <- Europe$Partiel_Ens
head(europe)
#> [[1]]
#> NULL
#>
#> $Durée
#> [1] 27.2 27.2 24.9 1.9 10.2 4.8 24.2 14.5 11.3 15.5 17.5 9.1 4.4 19.7 21.5
#> [16] 18.7 8.4 6.4 17.0 4.1 12.2 4.5 25.8 50.2 6.1 8.1 6.1 24.4 9.7 4.5
#> [31] 8.4 22.5 38.0 6.3 9.9
str(europe)
#> List of 2
#> $ : NULL
#> $ Durée: num [1:35] 27.2 27.2 24.9 1.9 10.2 4.8 24.2 14.5 11.3 15.5 ...
summary(europe)
#> Length Class Mode
#> 0 -none- NULL
#> Durée 35 -none- numeric
range(europe$Durée)
#> [1] 1.9 50.2
sd(europe$Durée)
#> [1] 10.71283
rangePartiel_Ens <-
cbind(range(Europe$Partiel_Ens),
range(Europe$Partiel_H),
range(Europe$Partiel_F))
ylimits = c(min(rangePartiel_Ens[1,]),
max(rangePartiel_Ens[2,]))Boîte à moustaches
qts <- quantile(europe$Durée,c(.25,.5,.75),type=6)
ltms <- c(qts[1]-3/2*(qts[3]-qts[1]),qts[3]+3/2*(qts[3]-qts[1]))
lms <- c(min(europe$Durée[europe$Durée>=qts[1]-3/2*(qts[3]-qts[1])]),max(europe$Durée[europe$Durée<=qts[3]+3/2*(qts[3]-qts[1])]))
conf <- qts[2] + c(-1.58, 1.58) * (qts[3]-qts[1])/sqrt(sum(!is.na(europe$Durée)))
#bltr
oldpar <- par()
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"boxplotPartiel_Ens.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "Europe boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"boxplotPartiel_Ens_same_scale.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "Europe boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=ylimits)
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
dev.off()
#> agg_png
#> 2Statistiques descriptives
mean(europe$Durée)
#> [1] 15.00571
quantile(europe$Durée,probs=c(.25,.5,.75),type=6)
#> 25% 50% 75%
#> 6.3 11.3 22.5
range(europe$Durée)
#> [1] 1.9 50.2
diff(quantile(europe$Durée,probs=c(.25,.75),type=6))
#> 75%
#> 16.2
diff(range(europe$Durée))
#> [1] 48.3
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2)
#> [1] 111.4857
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))
#> [1] 10.55868
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))/mean(europe$Durée)*100
#> [1] 70.36438
mean(sort(abs(europe$Durée-mean(sort(europe$Durée)[c(14)])))[c(14)])
#> [1] 4.3
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2))
#> [1] 1.254157
(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2)))^2
#> [1] 1.57291
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))
#> [1] 4.658759
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))-3
#> [1] 1.658759Europe Emploi Partiel_H
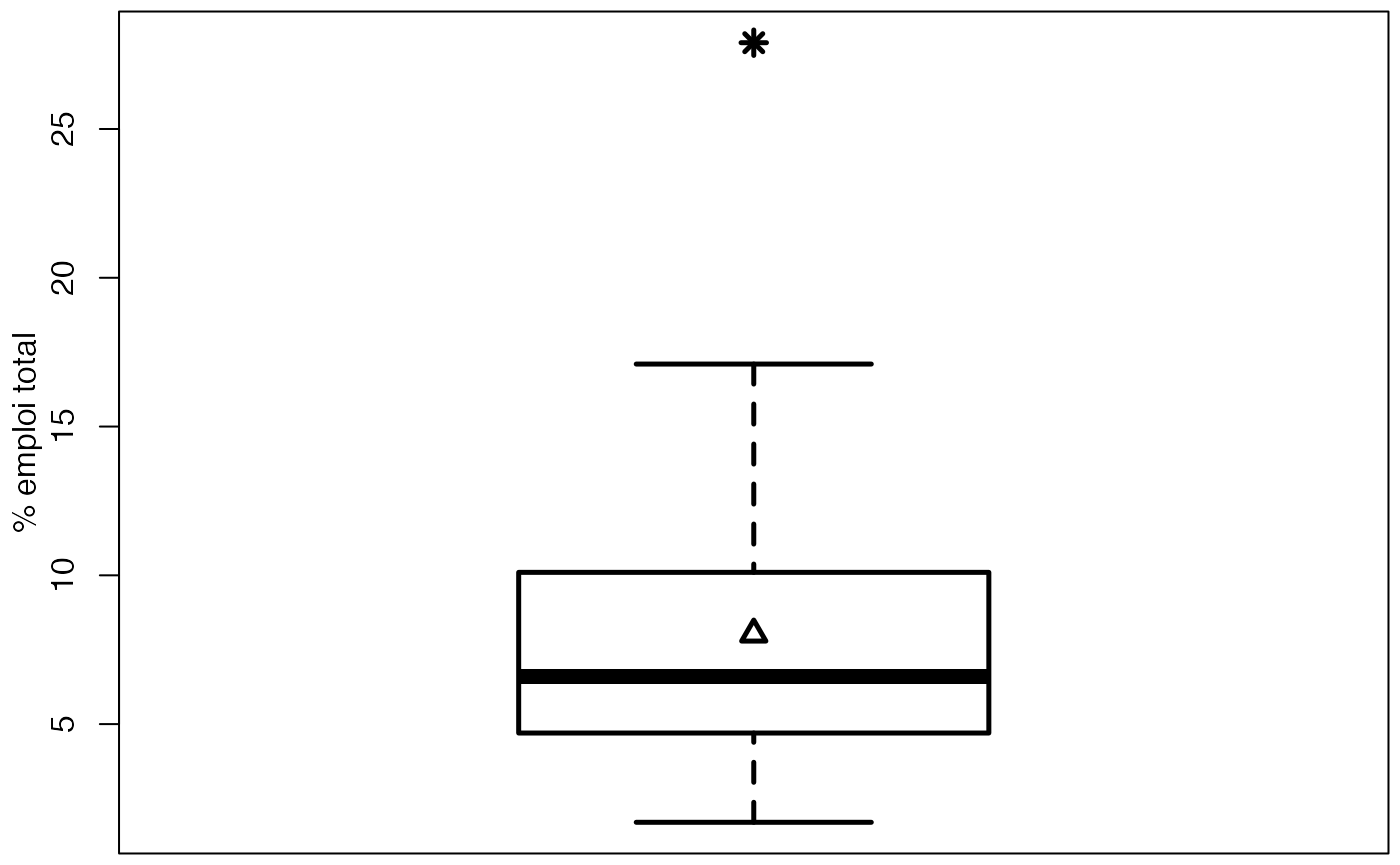
data(Europe)
europe <- vector("list",1)
europe$Durée <- Europe$Partiel_H
head(europe)
#> [[1]]
#> NULL
#>
#> $Durée
#> [1] 9.9 9.5 10.5 1.7 6.3 3.1 15.3 6.8 7.1 10.1 7.5 5.9 2.5 10.1 10.3
#> [16] 8.2 5.8 4.7 5.6 4.1 5.9 4.7 15.2 27.9 3.5 5.4 6.0 10.8 8.9 2.9
#> [31] 4.8 13.4 17.1 2.8 6.6
str(europe)
#> List of 2
#> $ : NULL
#> $ Durée: num [1:35] 9.9 9.5 10.5 1.7 6.3 3.1 15.3 6.8 7.1 10.1 ...
summary(europe)
#> Length Class Mode
#> 0 -none- NULL
#> Durée 35 -none- numeric
range(europe$Durée)
#> [1] 1.7 27.9
sd(europe$Durée)
#> [1] 5.144977Boîte à moustaches
qts <- quantile(europe$Durée,c(.25,.5,.75),type=6)
ltms <- c(qts[1]-3/2*(qts[3]-qts[1]),qts[3]+3/2*(qts[3]-qts[1]))
lms <- c(min(europe$Durée[europe$Durée>=qts[1]-3/2*(qts[3]-qts[1])]),max(europe$Durée[europe$Durée<=qts[3]+3/2*(qts[3]-qts[1])]))
conf <- qts[2] + c(-1.58, 1.58) * (qts[3]-qts[1])/sqrt(sum(!is.na(europe$Durée)))
#bltr
oldpar <- par()
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"boxplotPartiel_H.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "Europe boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"boxplotPartiel_H_same_scale.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "Europe boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=ylimits)
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
dev.off()
#> agg_png
#> 2Statistiques descriptives
mean(europe$Durée)
#> [1] 8.025714
quantile(europe$Durée,probs=c(.25,.5,.75),type=6)
#> 25% 50% 75%
#> 4.7 6.6 10.1
range(europe$Durée)
#> [1] 1.7 27.9
diff(quantile(europe$Durée,probs=c(.25,.75),type=6))
#> 75%
#> 5.4
diff(range(europe$Durée))
#> [1] 26.2
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2)
#> [1] 25.71448
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))
#> [1] 5.070945
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))/mean(europe$Durée)*100
#> [1] 63.18372
mean(sort(abs(europe$Durée-mean(sort(europe$Durée)[c(14)])))[c(14)])
#> [1] 1.6
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2))
#> [1] 1.845463
(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2)))^2
#> [1] 3.405734
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))
#> [1] 7.561845
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))-3
#> [1] 4.561845Europe Emploi Partiel_F
data(Europe)
europe <- vector("list",1)
europe$Durée <- Europe$Partiel_F
head(europe)
#> [[1]]
#> NULL
#>
#> $Durée
#> [1] 46.7 47.1 41.0 2.1 14.6 6.7 33.9 23.7 15.9 21.3 28.0 13.5 6.8 30.6 34.1
#> [16] 32.9 10.9 8.0 30.4 4.3 21.4 4.1 37.7 75.2 9.3 10.9 6.2 39.4 10.6 6.5
#> [31] 12.7 32.5 61.7 10.6 17.0
str(europe)
#> List of 2
#> $ : NULL
#> $ Durée: num [1:35] 46.7 47.1 41 2.1 14.6 6.7 33.9 23.7 15.9 21.3 ...
summary(europe)
#> Length Class Mode
#> 0 -none- NULL
#> Durée 35 -none- numeric
range(europe$Durée)
#> [1] 2.1 75.2
sd(europe$Durée)
#> [1] 17.43912Boîte à moustaches
qts <- quantile(europe$Durée,c(.25,.5,.75),type=6)
ltms <- c(qts[1]-3/2*(qts[3]-qts[1]),qts[3]+3/2*(qts[3]-qts[1]))
lms <- c(min(europe$Durée[europe$Durée>=qts[1]-3/2*(qts[3]-qts[1])]),max(europe$Durée[europe$Durée<=qts[3]+3/2*(qts[3]-qts[1])]))
conf <- qts[2] + c(-1.58, 1.58) * (qts[3]-qts[1])/sqrt(sum(!is.na(europe$Durée)))
#bltr
oldpar <- par()
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)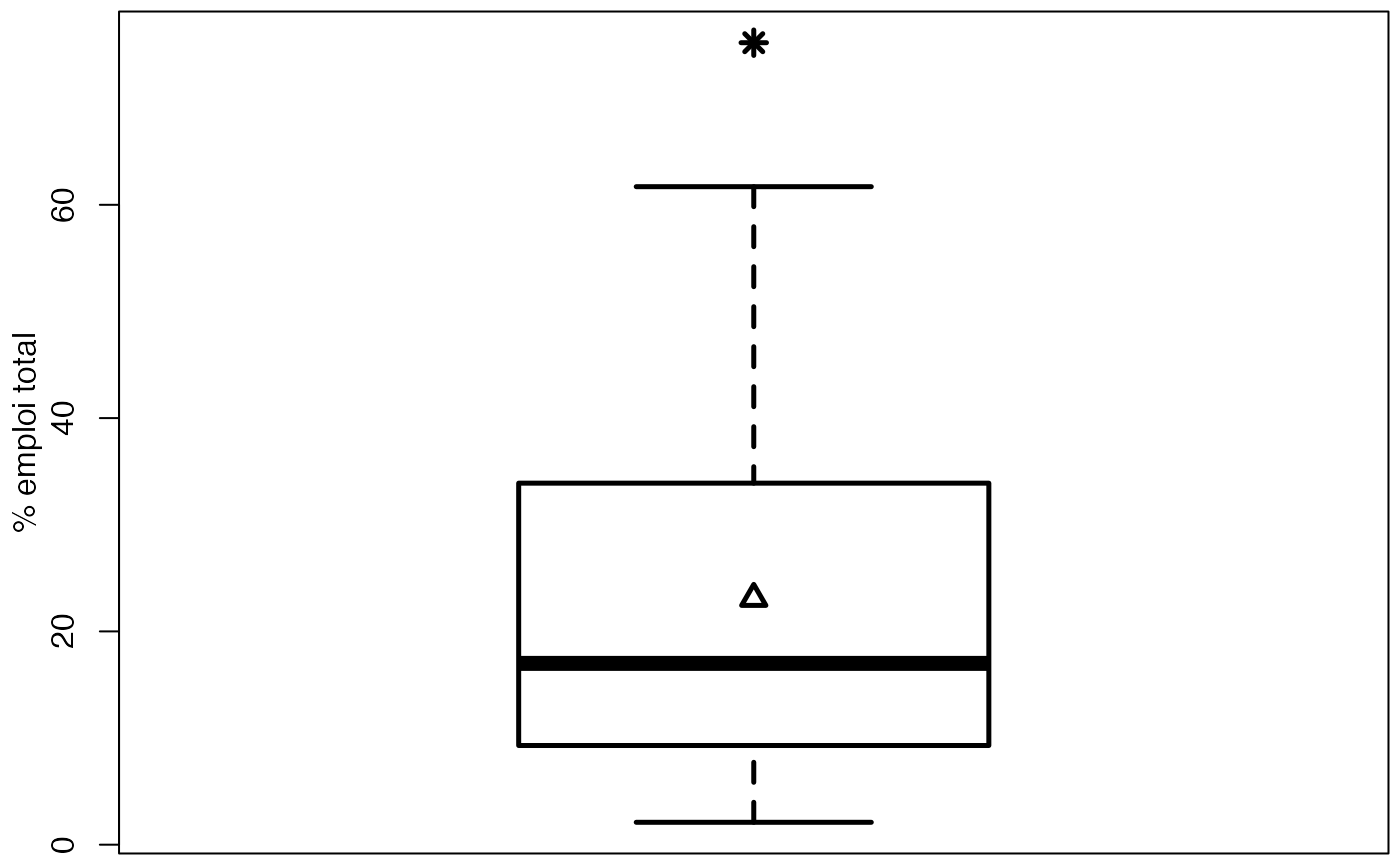
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"boxplotPartiel_F.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "Europe boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=range(europe$Durée))
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
dev.off()
#> agg_png
#> 2
pdf(file = paste(Chemin,"boxplotPartiel_F_same_scale.pdf",sep=""),
width = 6, height = 6, onefile = TRUE, family = "Helvetica",
title = "Europe boxplot", paper = "special")
par(mar = c(0.2, 3, 0.2, 0.2) + 0.1, mgp = c(2, 1, 0))
bxp(list(stats=t(t(c(lms[1],qts,lms[2]))),n=sum(!is.na(europe$Durée)),conf=matrix(conf,ncol=1)),ylab="% emploi total",lwd=2.5,cex.axis=1,cex.lab=1,ylim=ylimits)
points(1,mean(europe$Durée),pch=2,lwd=2.5,cex=1.25)
points(1,
c(
europe$Durée[!(europe$Durée>=qts[1]-3/2*(qts[3]-qts[1]))],
europe$Durée[!(europe$Durée<=qts[3]+3/2*(qts[3]-qts[1]))]
),pch=8,cex=1.25,lwd=2.5
)
dev.off()
#> agg_png
#> 2Statistiques descriptives
mean(europe$Durée)
#> [1] 23.09429
quantile(europe$Durée,probs=c(.25,.5,.75),type=6)
#> 25% 50% 75%
#> 9.3 17.0 33.9
range(europe$Durée)
#> [1] 2.1 75.2
diff(quantile(europe$Durée,probs=c(.25,.75),type=6))
#> 75%
#> 24.6
diff(range(europe$Durée))
#> [1] 73.1
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2)
#> [1] 295.4337
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))
#> [1] 17.18818
sqrt(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))/mean(europe$Durée)*100
#> [1] 74.42614
mean(sort(abs(europe$Durée-mean(sort(europe$Durée)[c(14)])))[c(14)])
#> [1] 6.2
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2))
#> [1] 1.051022
(1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^3)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(3/2)))^2
#> [1] 1.104648
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))
#> [1] 3.753343
1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^4)/((1/length(europe$Durée)*sum((europe$Durée-mean(europe$Durée))^2))^(4/2))-3
#> [1] 0.753343Partiel Ensemble
data(Europe)
var1<- Europe$Partiel_Ens
varN <- "_Ens"
xi <- unique(sort(var1))
ni <- as.vector(table(var1))
data2 <- cbind(xi,ni)Diagramme en bâtons des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
#axis(1, at = 1:6, labels = c(1:5,6))
#axis(1, at = 1:6, labels = c(1:5,"6 et plus"))
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"batoneffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"batonfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs et polygone des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs et polygone des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")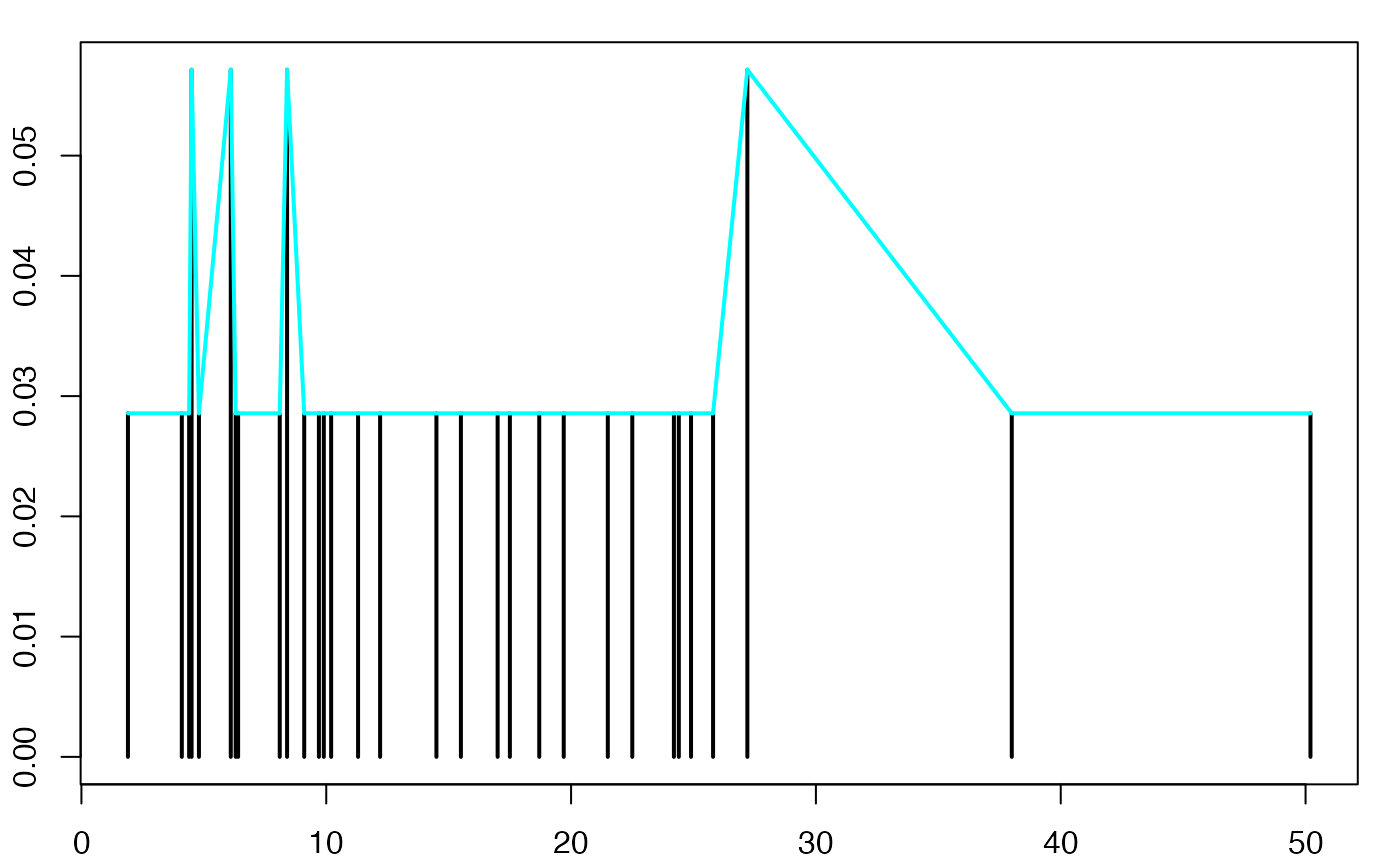
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés
nisum <- rep(ni[1],length(ni))
for (iii in 2:length(ni))
{
nisum[iii] <- nisum[iii-1]+ni[iii]
}
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")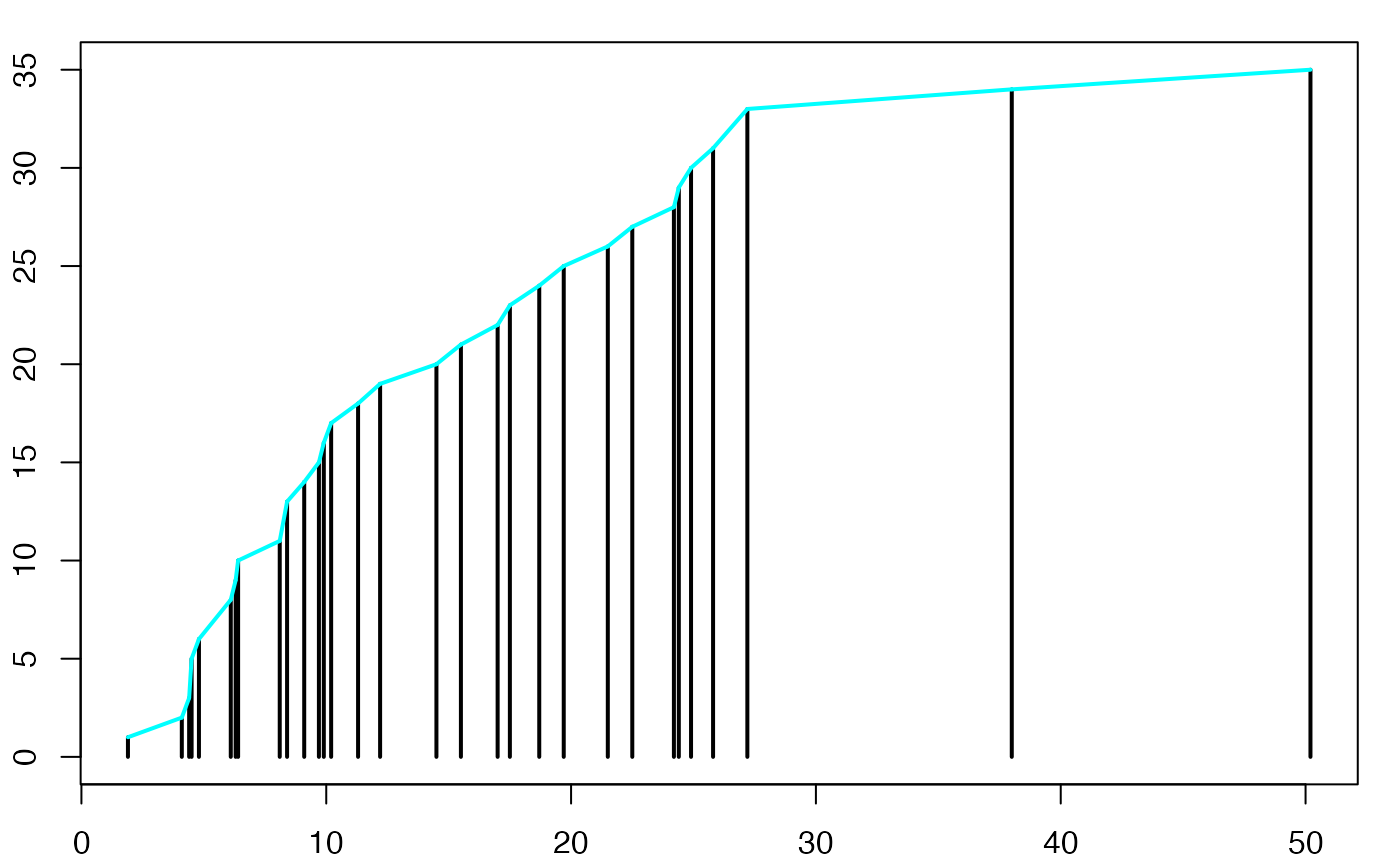
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"frecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"effcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Histogramme des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)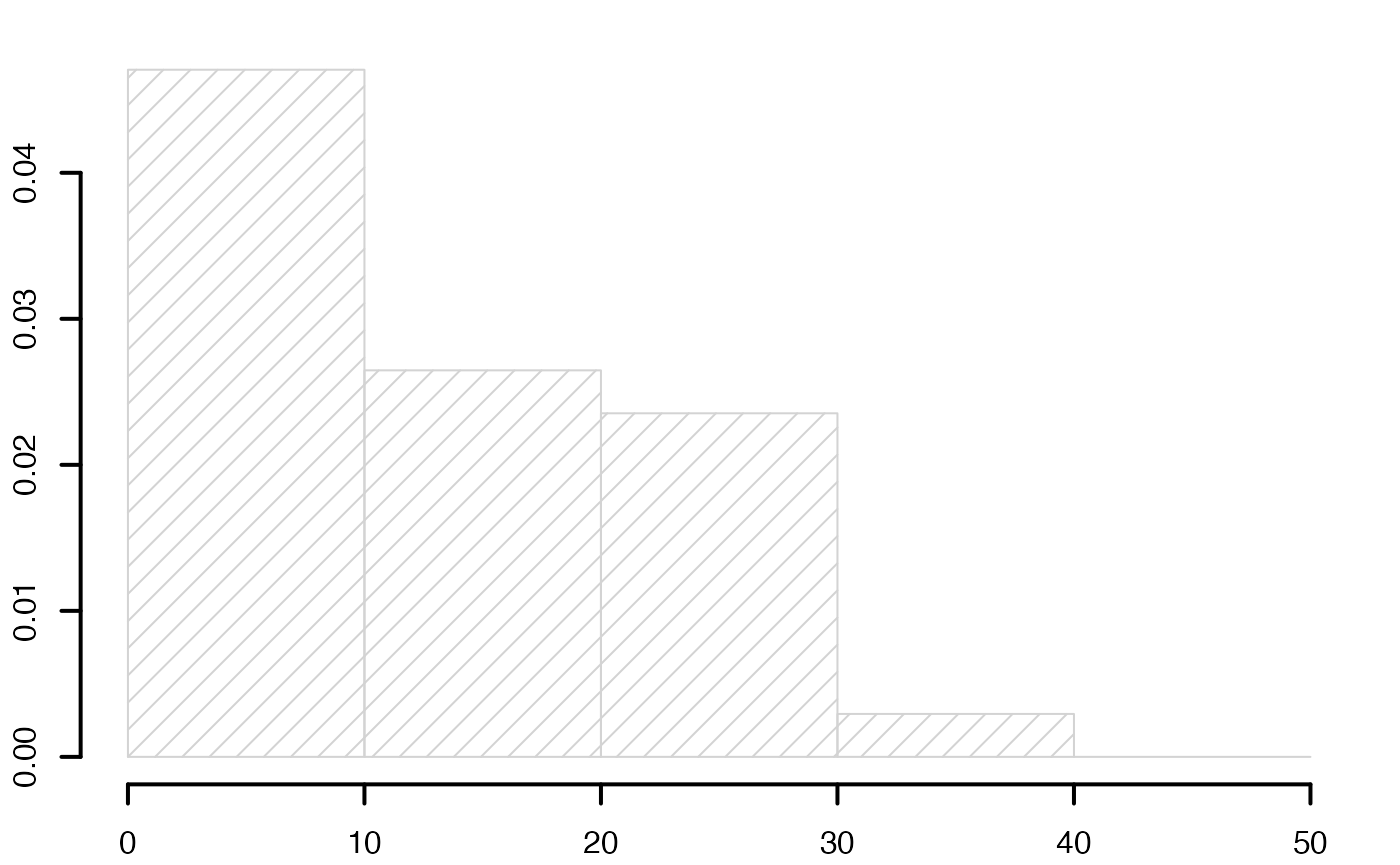
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histofrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
dev.off()
#> agg_png
#> 2Histogramme des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")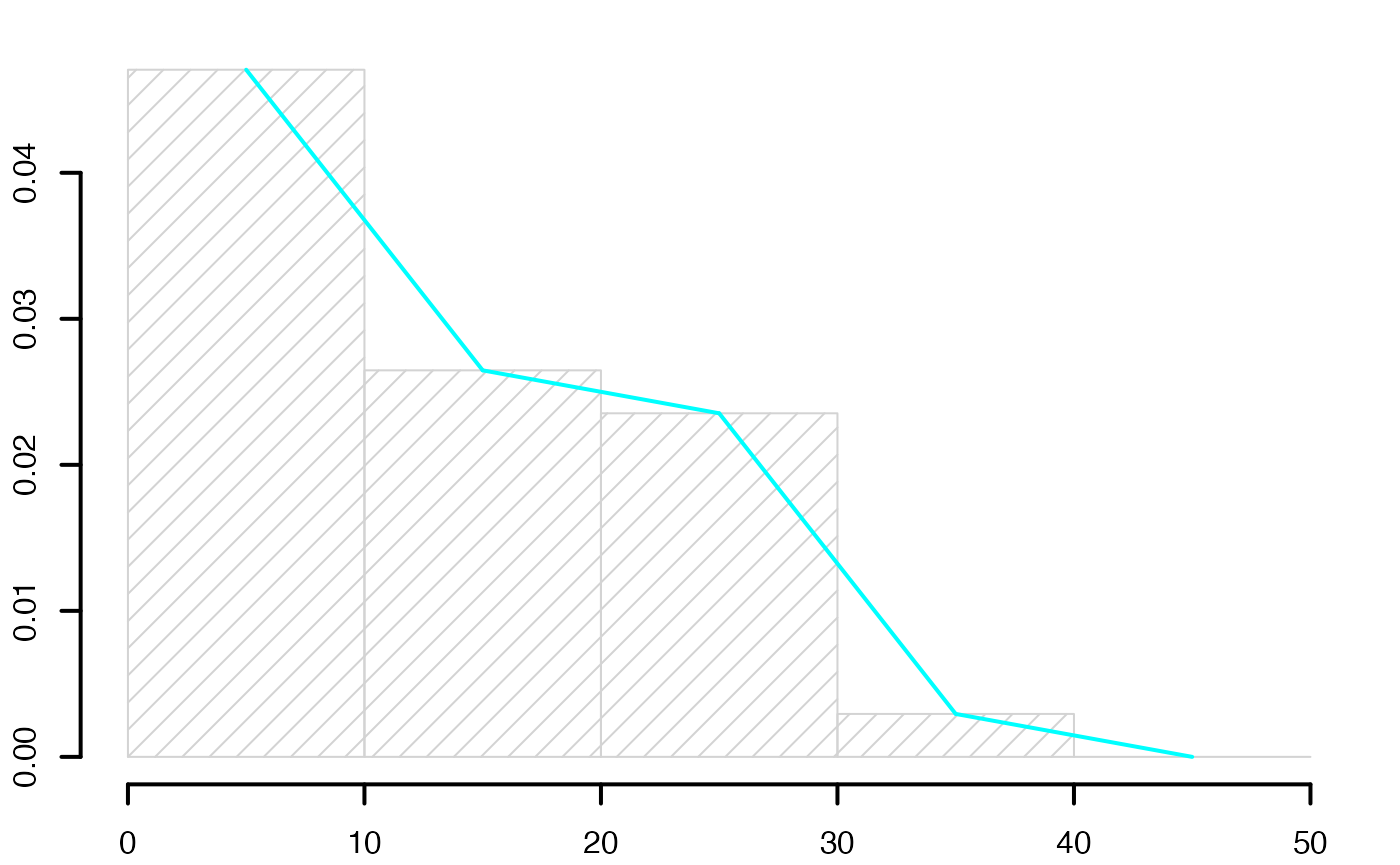
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histopolfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulés
data4 <- rep((breaks0[-length(breaks0)]+diff(breaks0)/2),cumsum(table(cut(var1,breaks0))))
table(data4)
#> data4
#> 5 15 25 35 45
#> 16 25 33 34 34
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histofrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulées et polygone des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)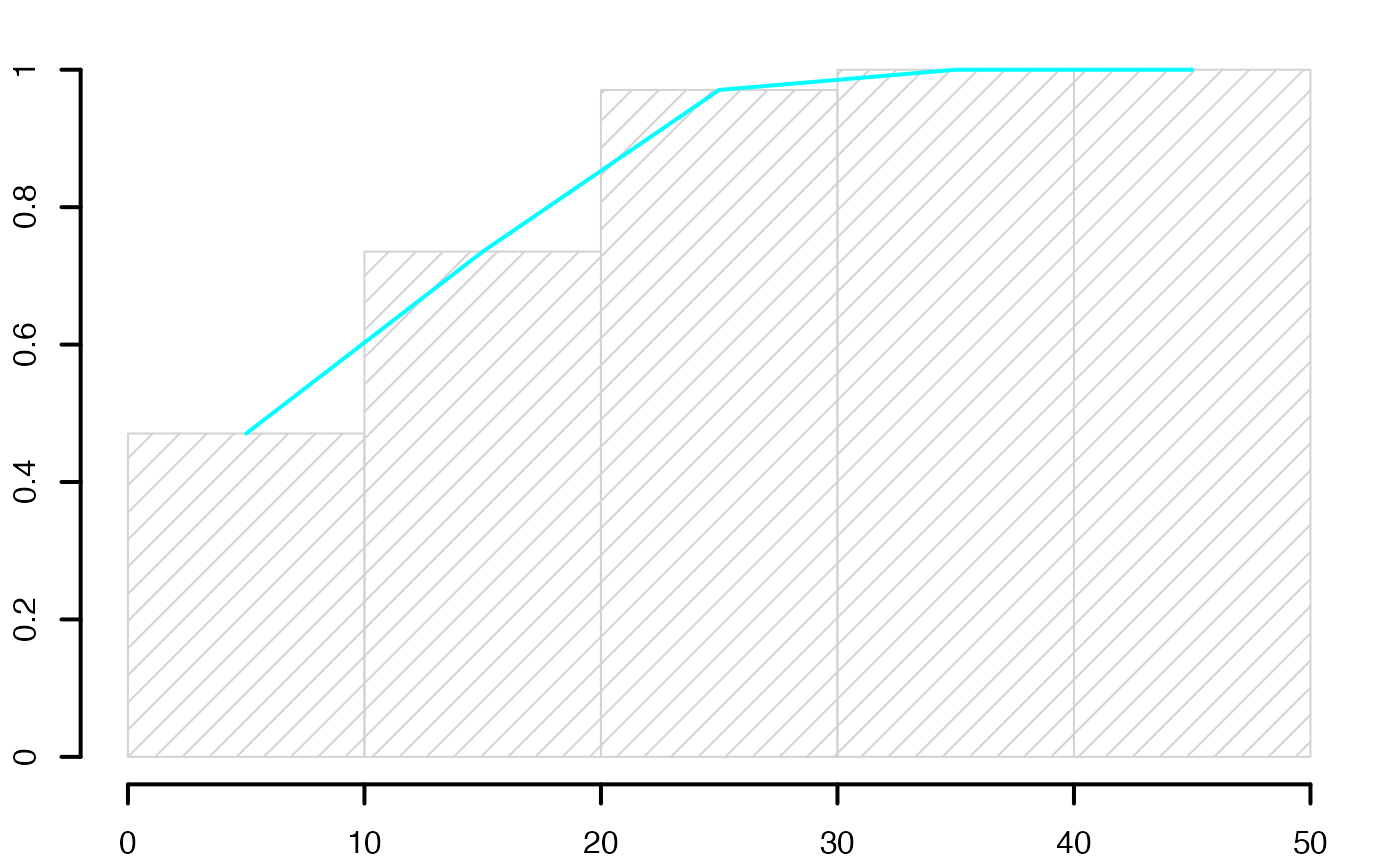
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulées et polygone des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des effectifs cumulés et polygone des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")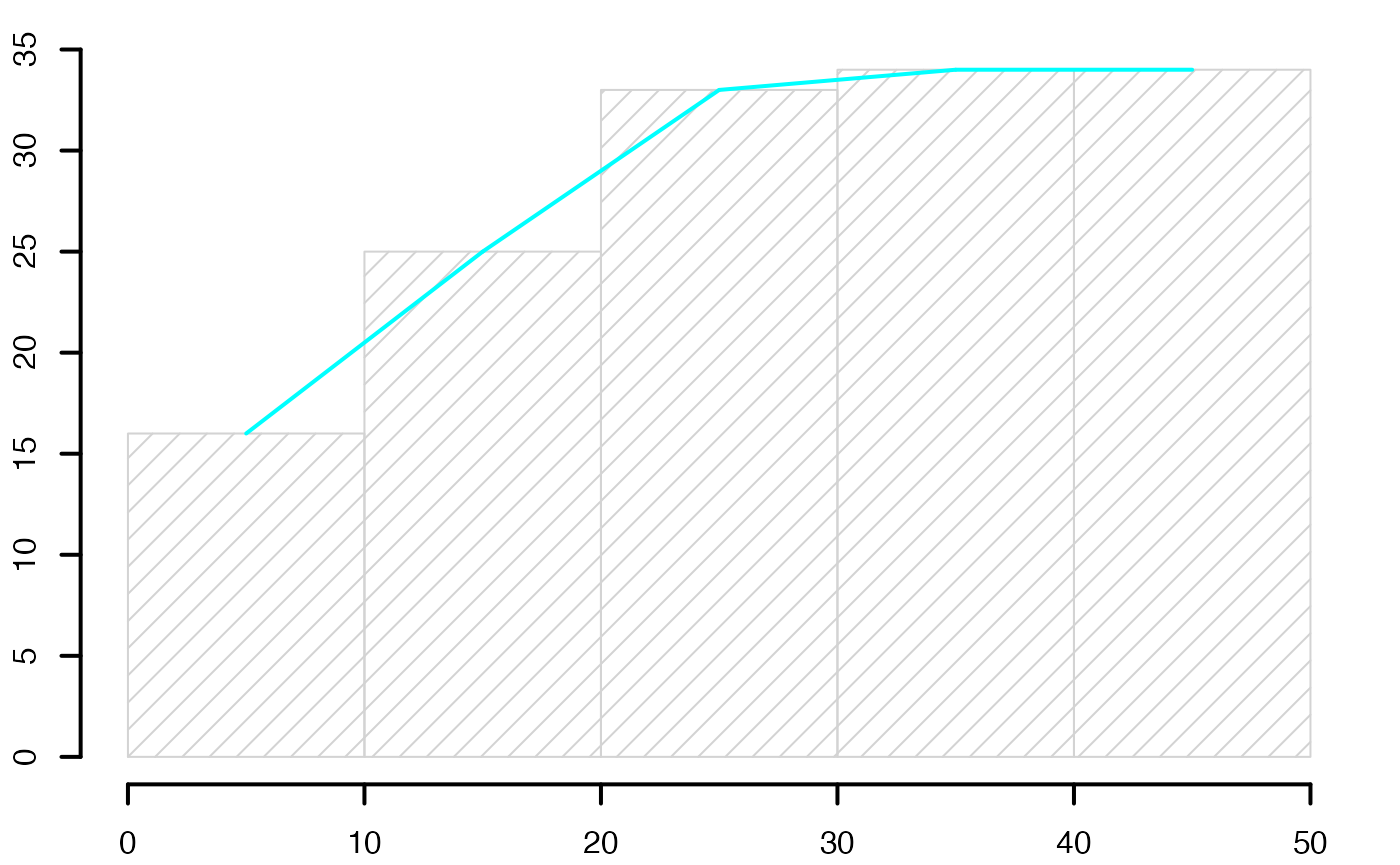
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopoleffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
dev.off()
#> agg_png
#> 2Partiel Hommes
data(Europe)
var1<- Europe$Partiel_H
varN <- "_H"
xi <- unique(sort(var1))
ni <- as.vector(table(var1))
data2 <- cbind(xi,ni)Diagramme en bâtons des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
#axis(1, at = 1:6, labels = c(1:5,6))
#axis(1, at = 1:6, labels = c(1:5,"6 et plus"))
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"batoneffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"batonfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs et polygone des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs et polygone des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés
nisum <- rep(ni[1],length(ni))
for (iii in 2:length(ni))
{
nisum[iii] <- nisum[iii-1]+ni[iii]
}
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"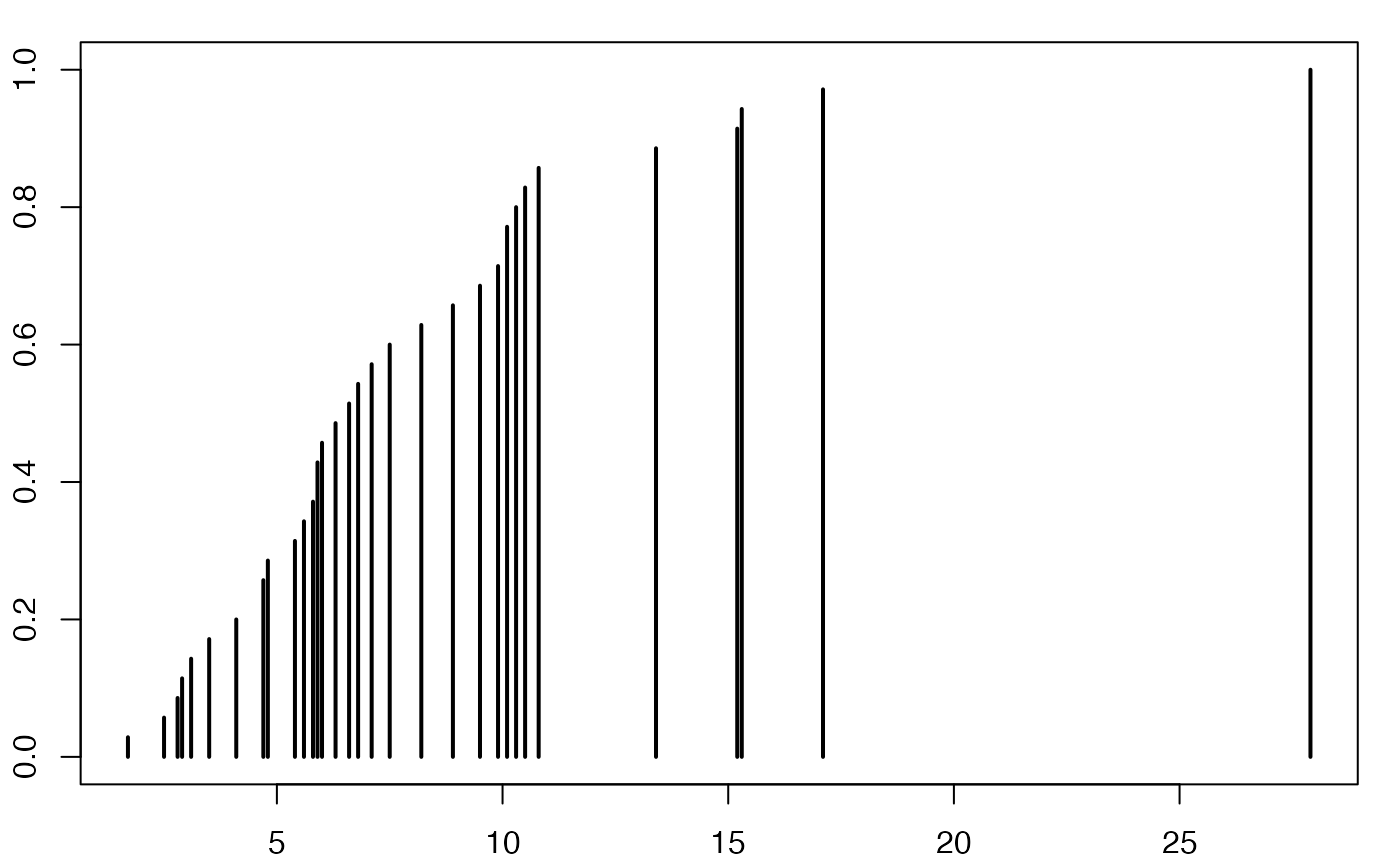
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"frecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"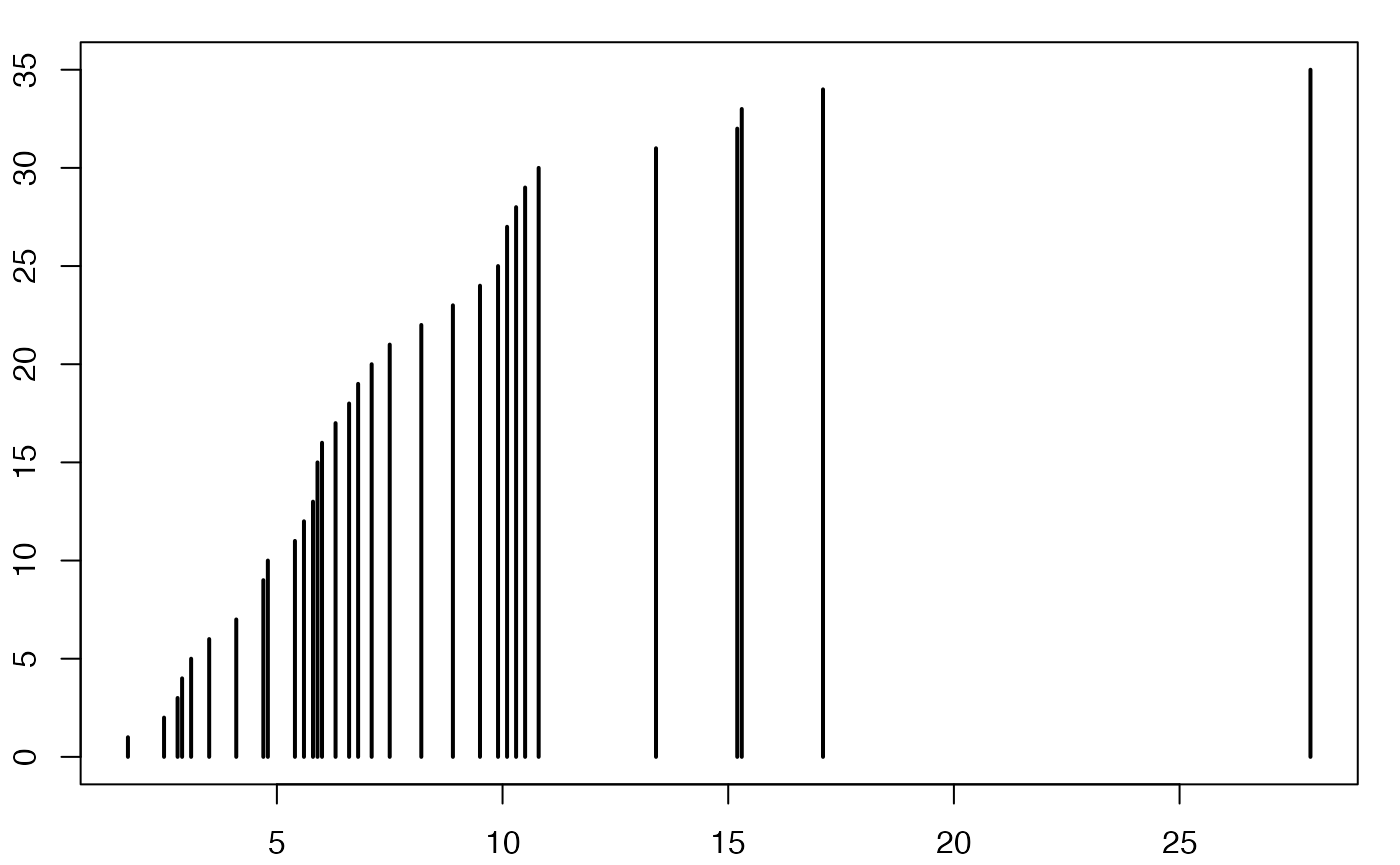
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"effcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Histogramme des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histofrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
dev.off()
#> agg_png
#> 2Histogramme des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")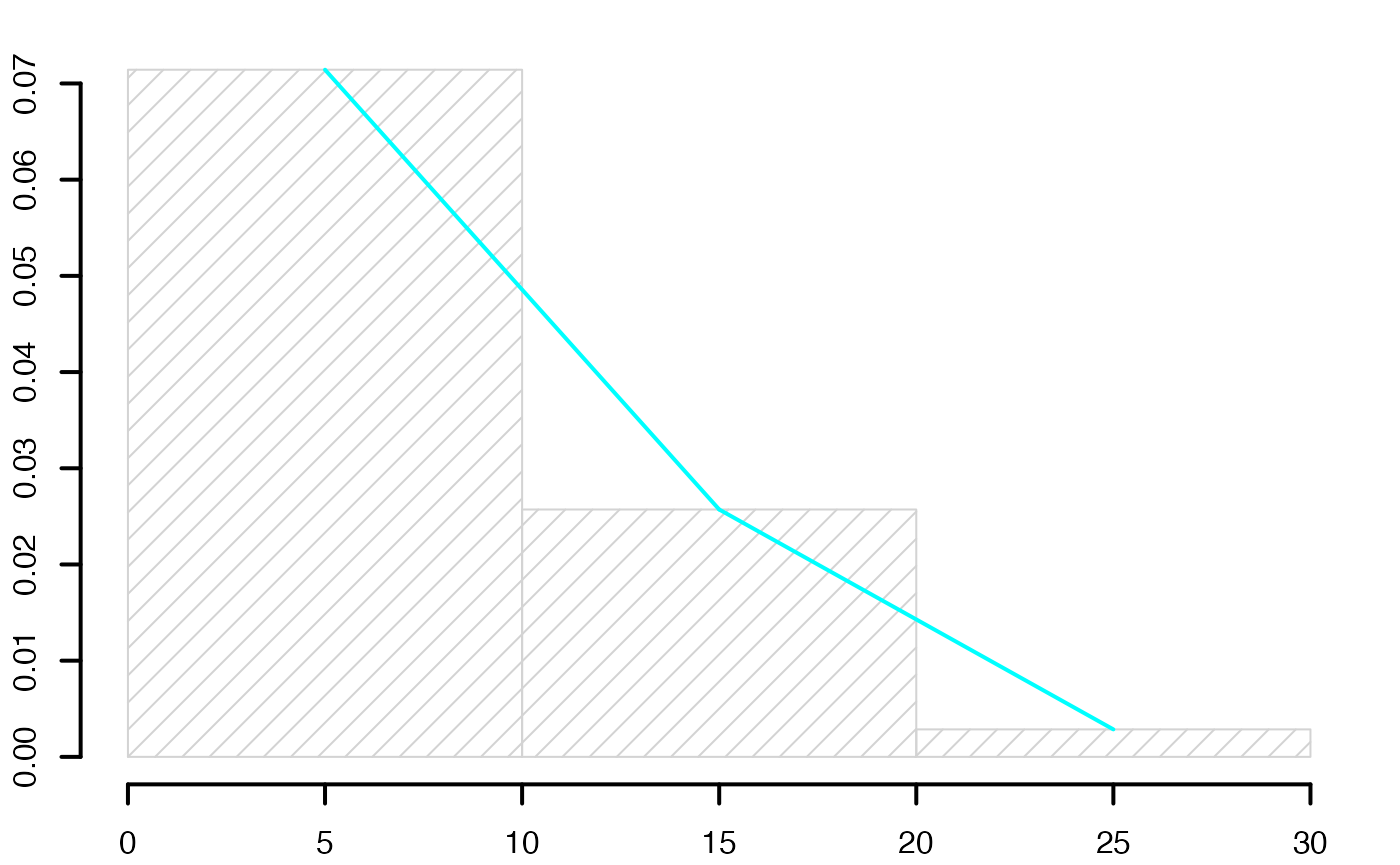
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulés
data4 <- rep((breaks0[-length(breaks0)]+diff(breaks0)/2)
,cumsum(table(cut(var1,breaks0))))
table(data4)
#> data4
#> 5 15 25
#> 25 34 35
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histofrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulées et polygone des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)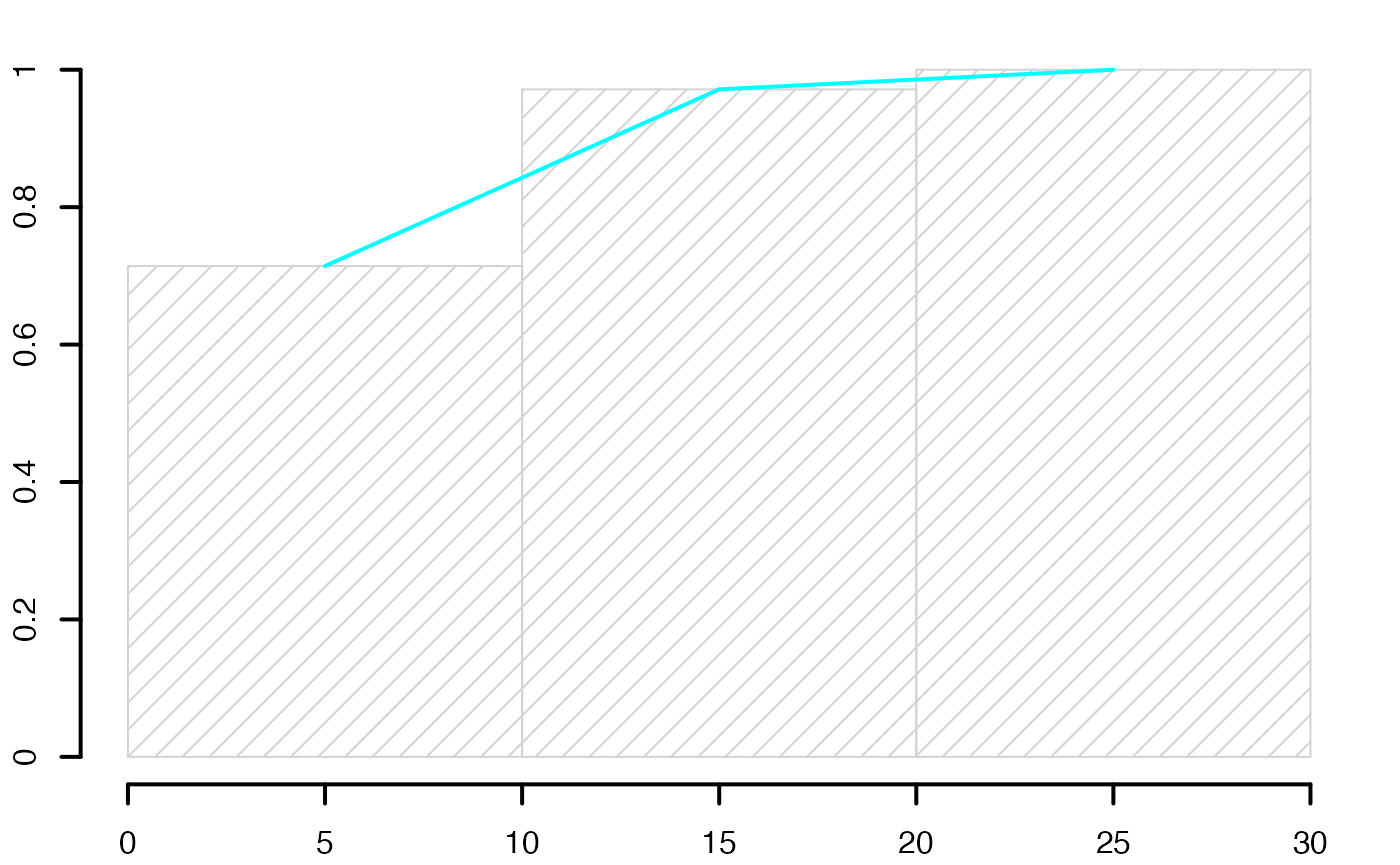
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulées et polygone des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des effectifs cumulés et polygone des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")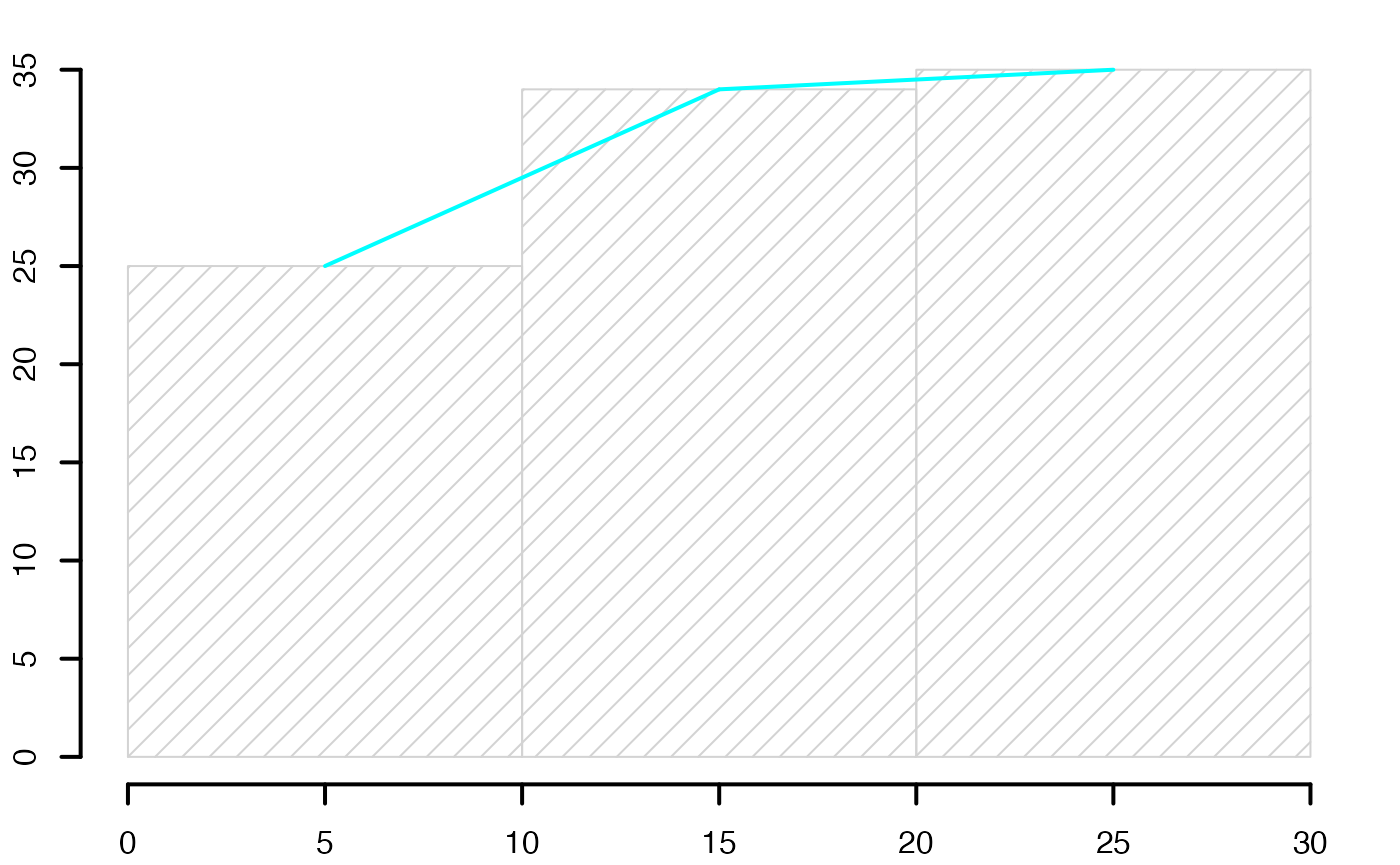
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopoleffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
dev.off()
#> agg_png
#> 2Partiel Femmes
data(Europe)
var1<- Europe$Partiel_F
varN <- "_F"
xi <- unique(sort(var1))
ni <- as.vector(table(var1))
data2 <- cbind(xi,ni)Diagramme en bâtons des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
#axis(1, at = 1:6, labels = c(1:5,6))
#axis(1, at = 1:6, labels = c(1:5,"6 et plus"))
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"batoneffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"batonfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs et polygone des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs et polygone des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés
nisum <- rep(ni[1],length(ni))
for (iii in 2:length(ni))
{
nisum[iii] <- nisum[iii-1]+ni[iii]
}
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"frecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"effcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Histogramme des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)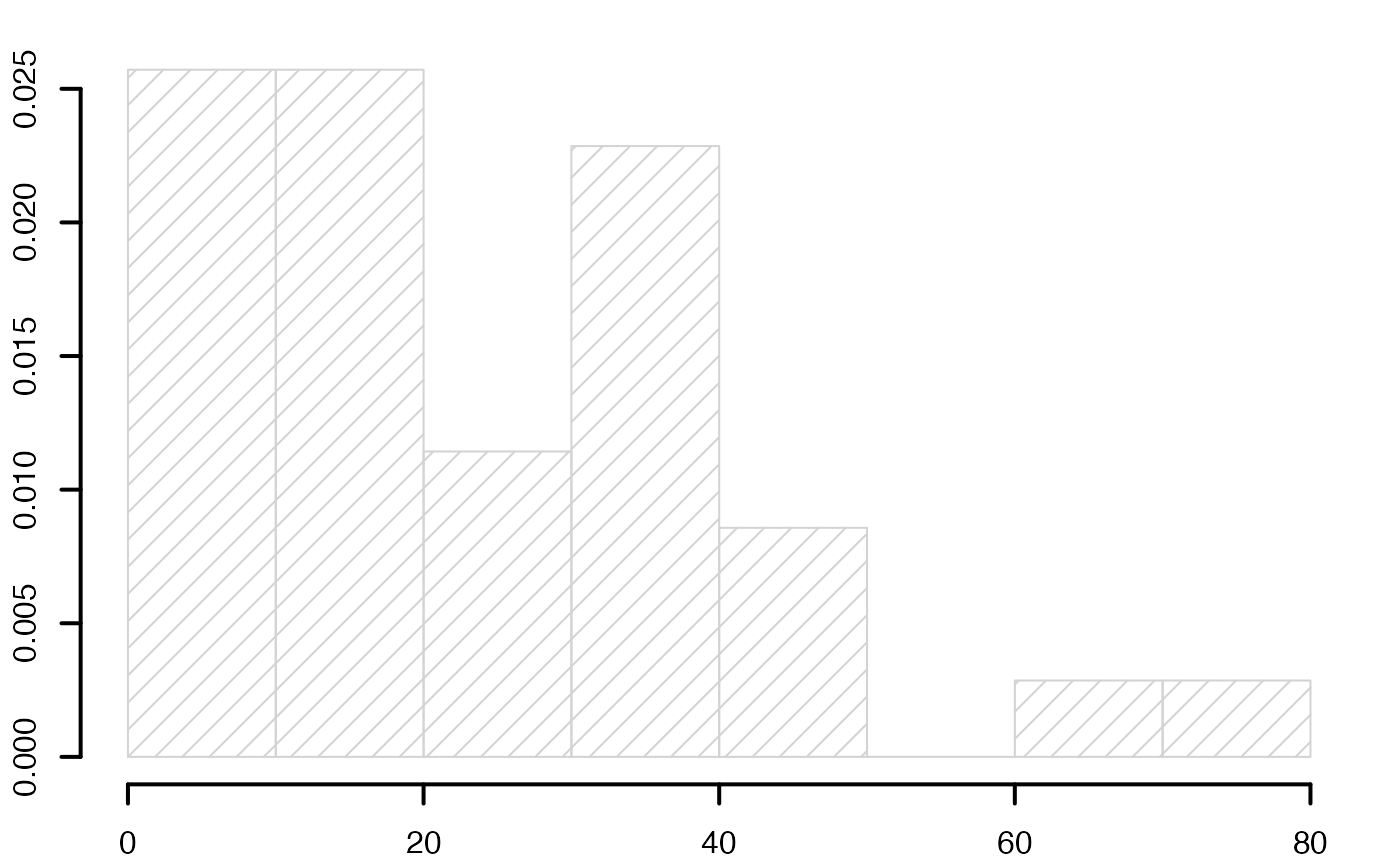
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histofrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
dev.off()
#> agg_png
#> 2Histogramme des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")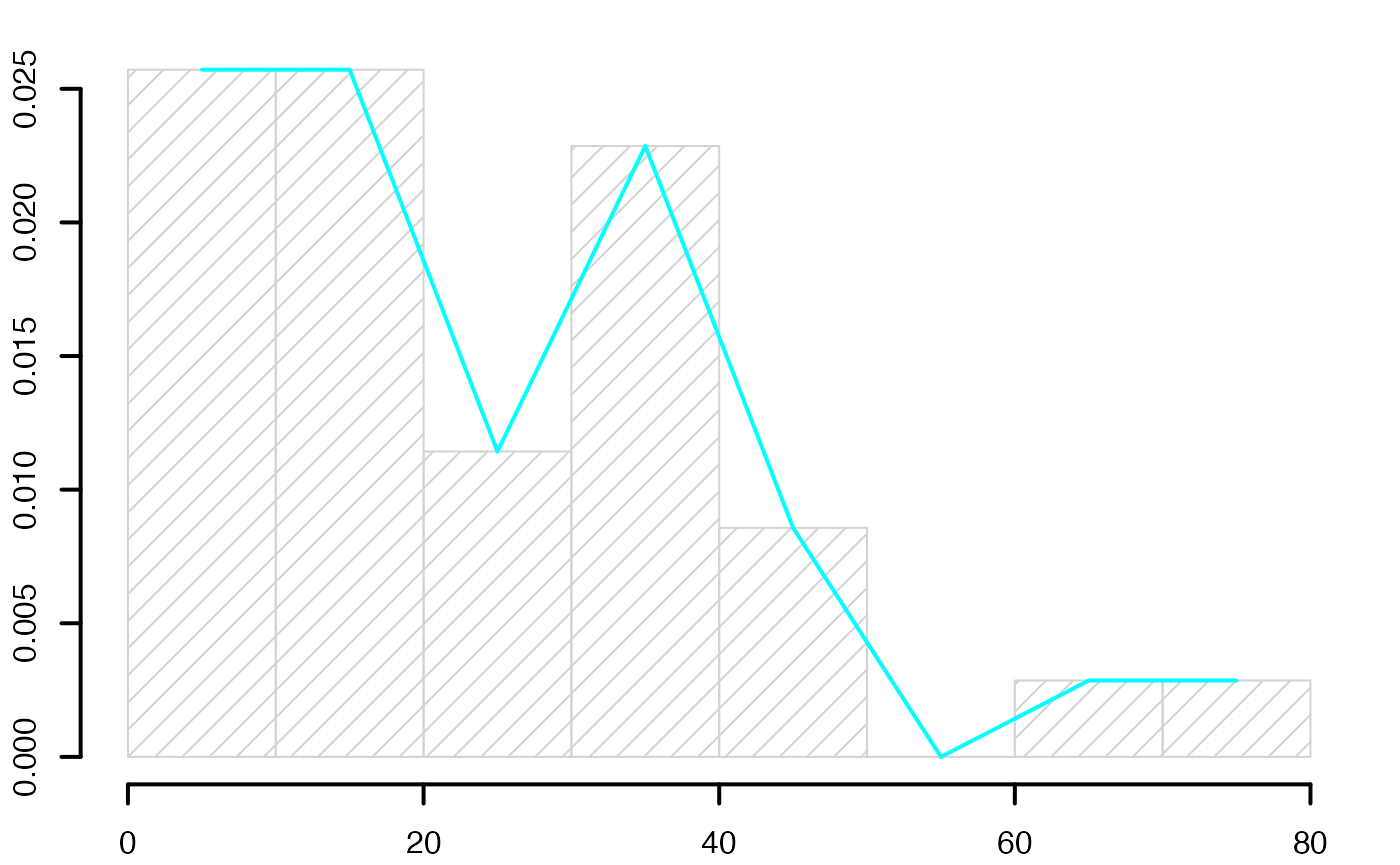
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulés
data4 <- rep((breaks0[-length(breaks0)]+diff(breaks0)/2)
,cumsum(table(cut(var1,breaks0))))
table(data4)
#> data4
#> 5 15 25 35 45 55 65 75
#> 9 18 22 30 33 33 34 35
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histofrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulées et polygone des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)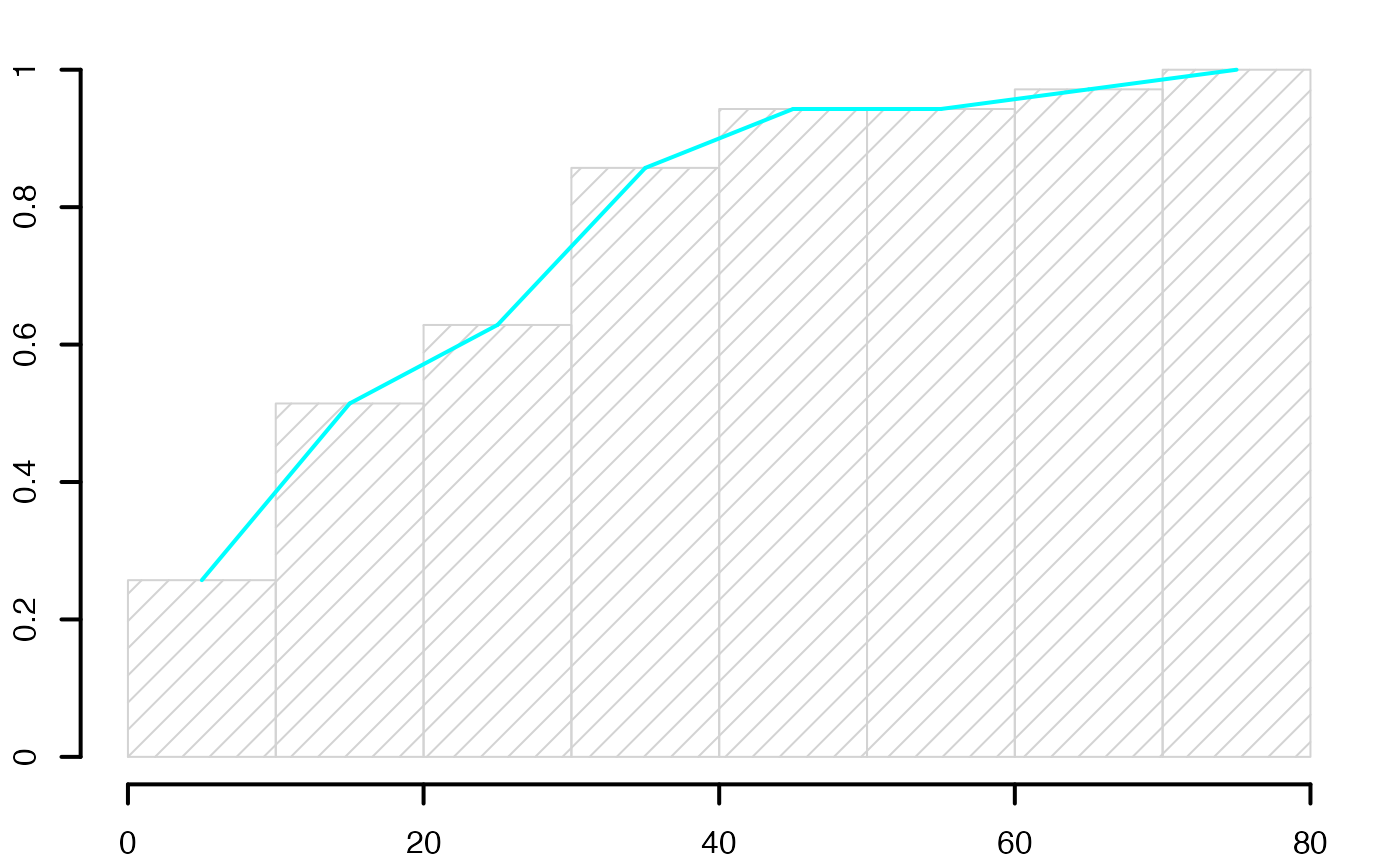
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulées et polygone des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des effectifs cumulés et polygone des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")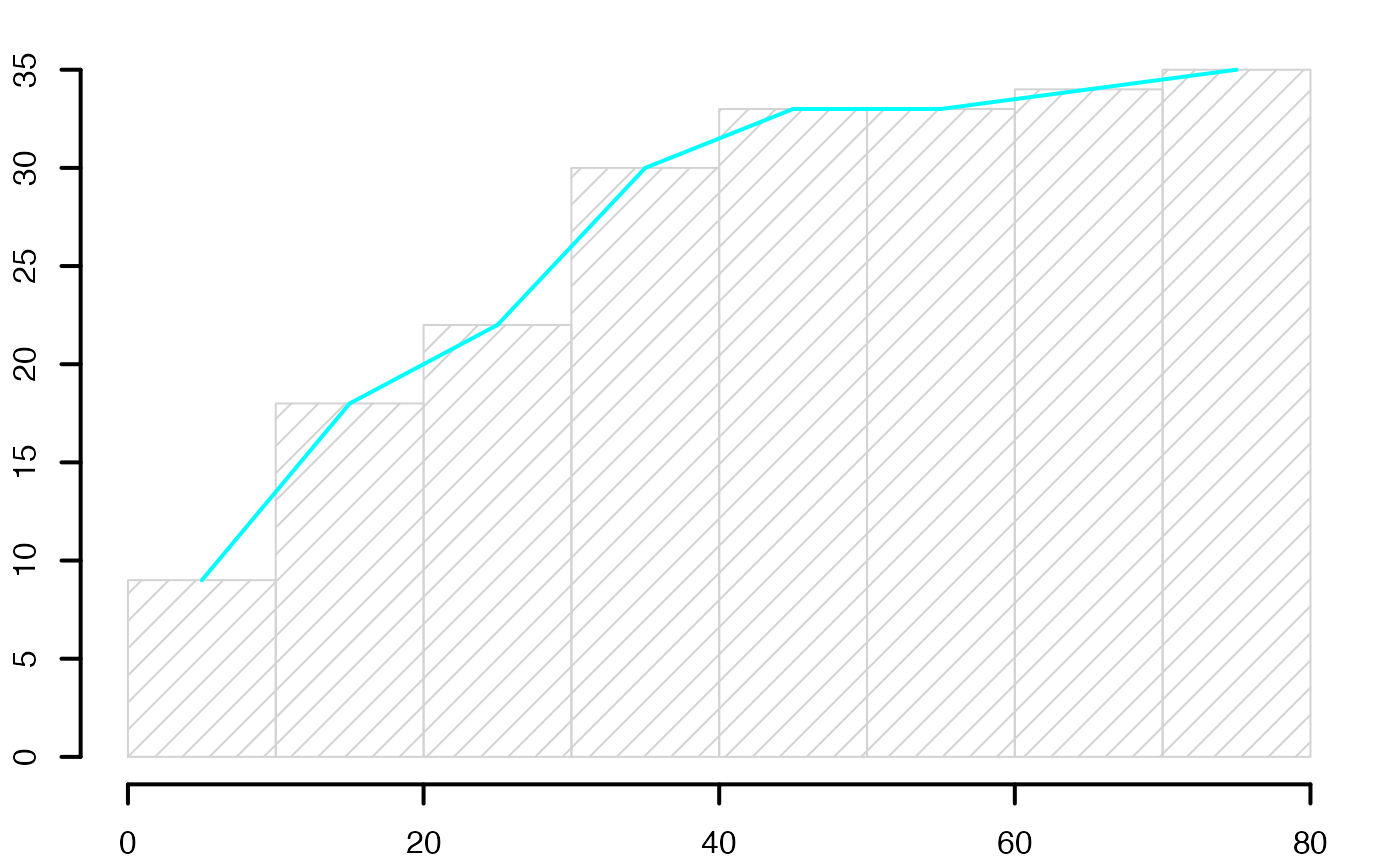
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopoleffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
dev.off()
#> agg_png
#> 2Europe Salariés
data(Europe)
var1<- Europe$Salariés
varN <- "_Sal"
xi <- unique(sort(var1))
ni <- as.vector(table(var1))
data2 <- cbind(xi,ni)Diagramme en bâtons des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"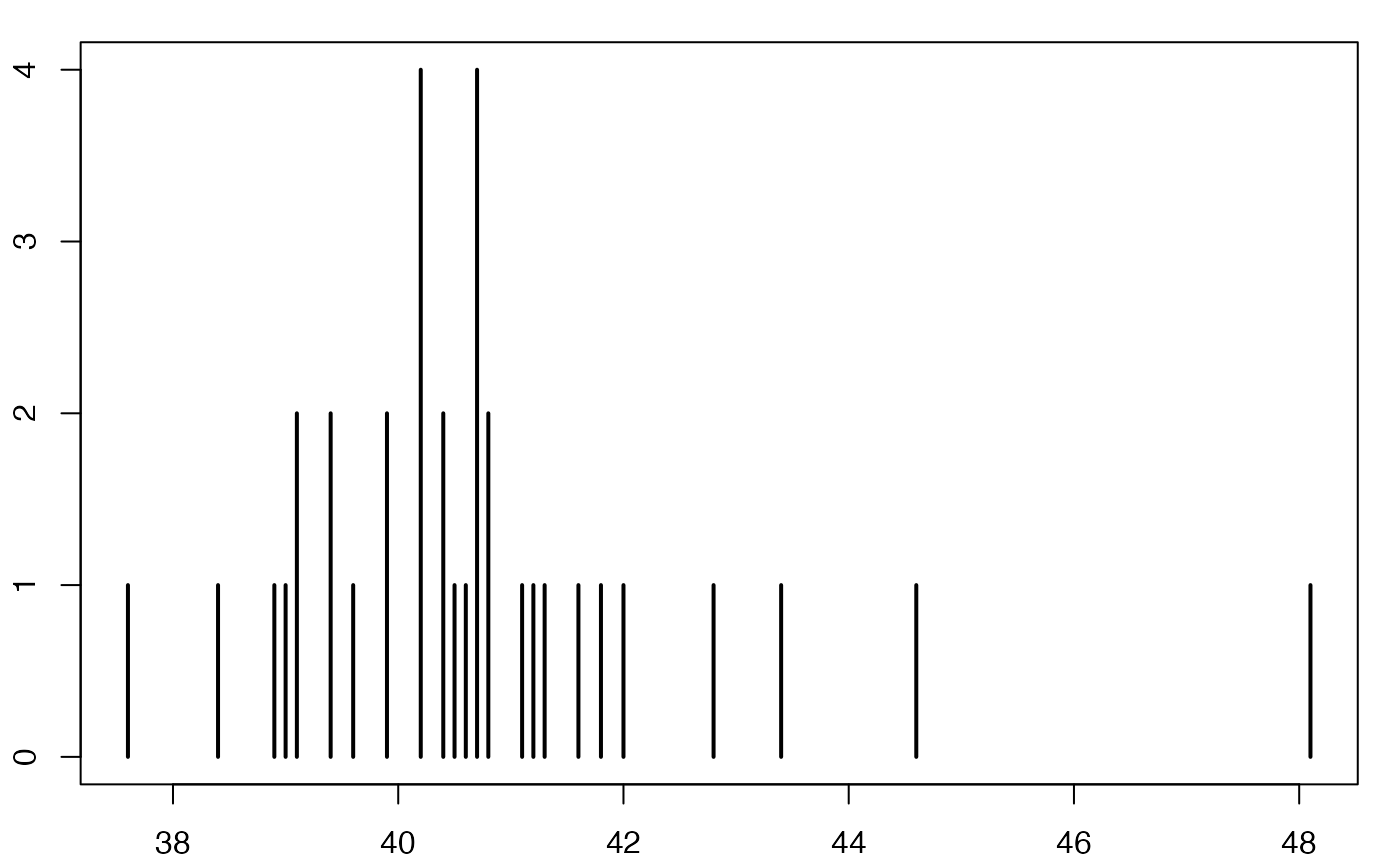
#axis(1, at = 1:6, labels = c(1:5,6))
#axis(1, at = 1:6, labels = c(1:5,"6 et plus"))
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"batoneffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs et polygone des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"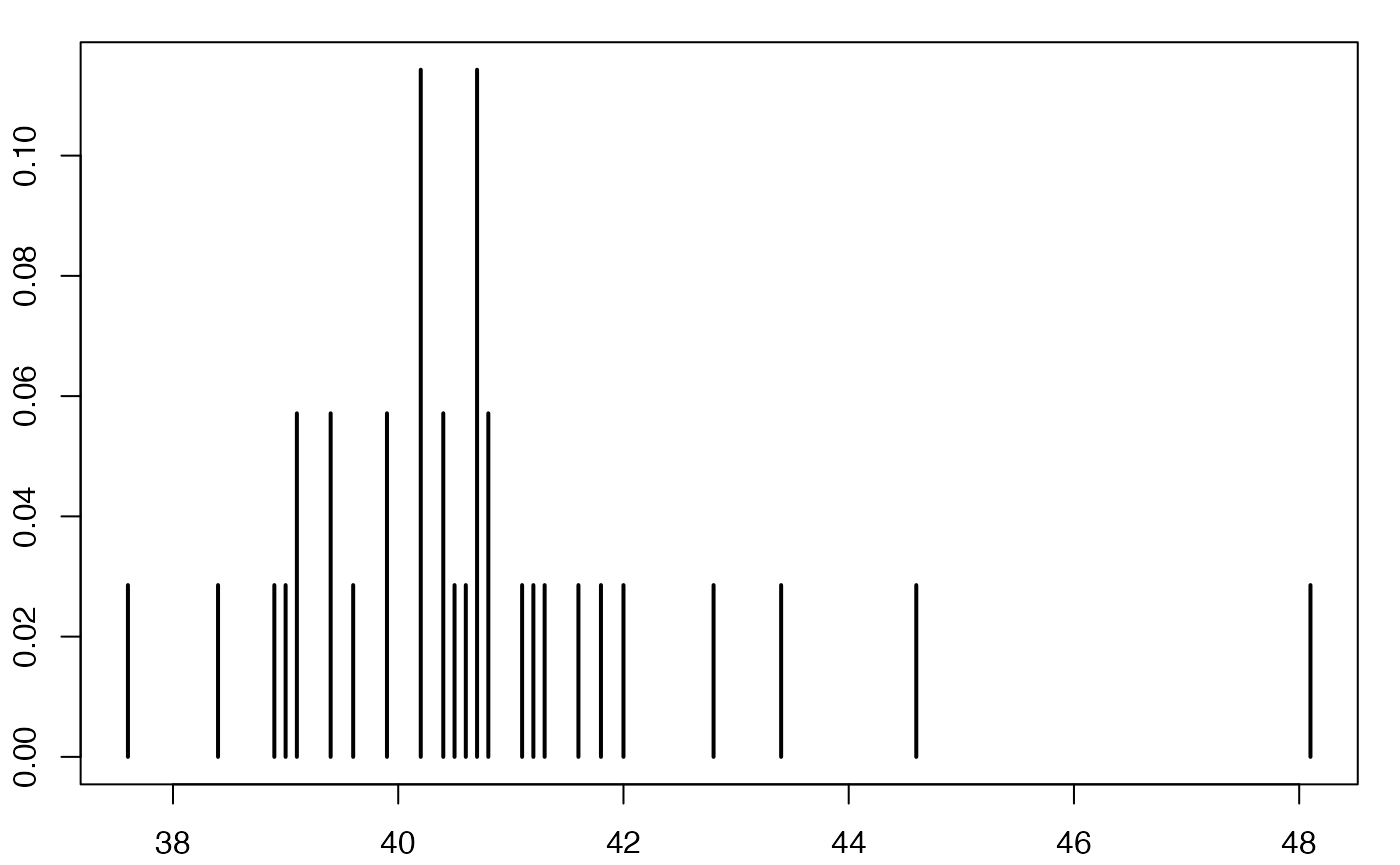
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"batonfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Probability or cumulative distribution graphs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs et polygone des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")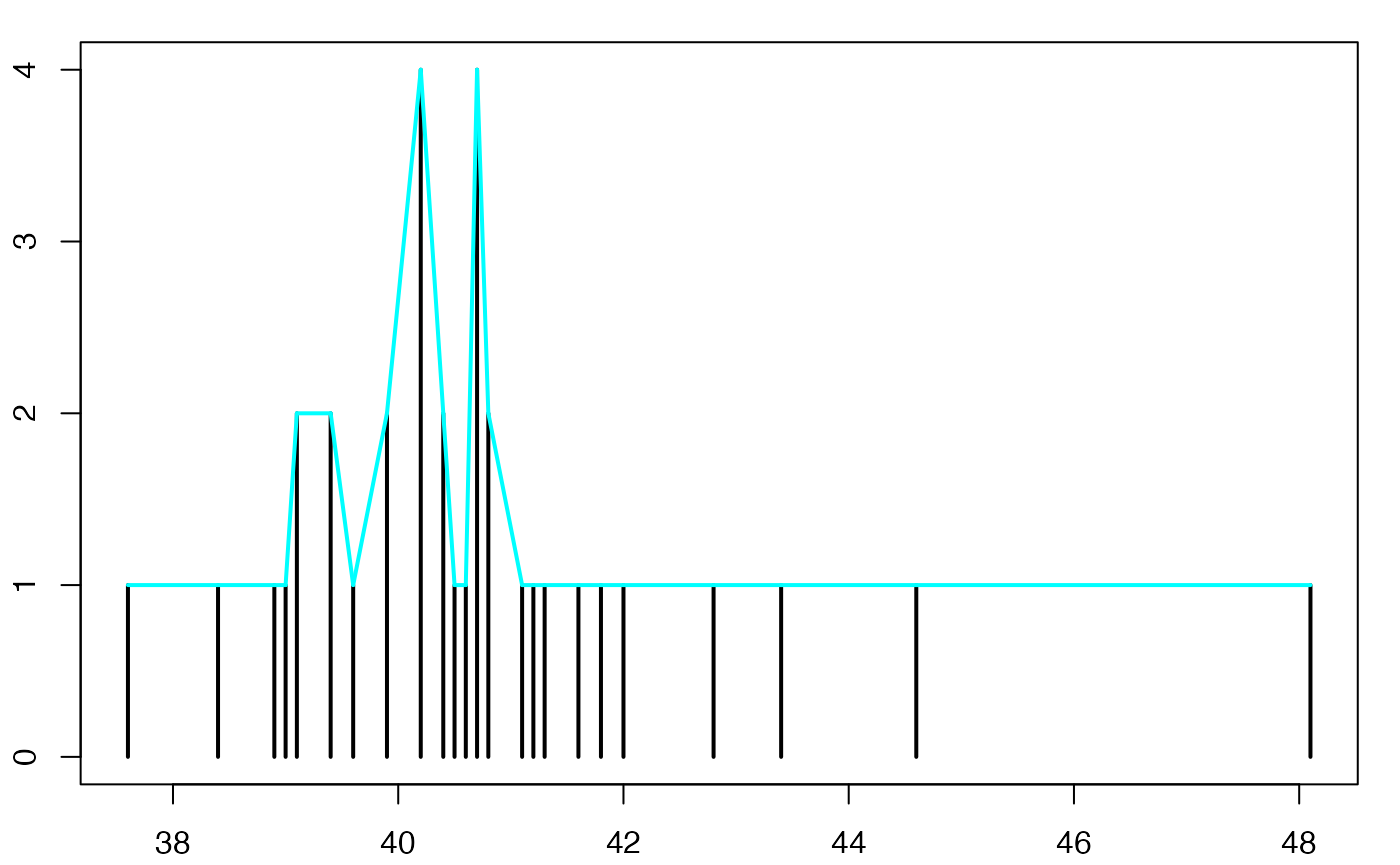
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs et polygone des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")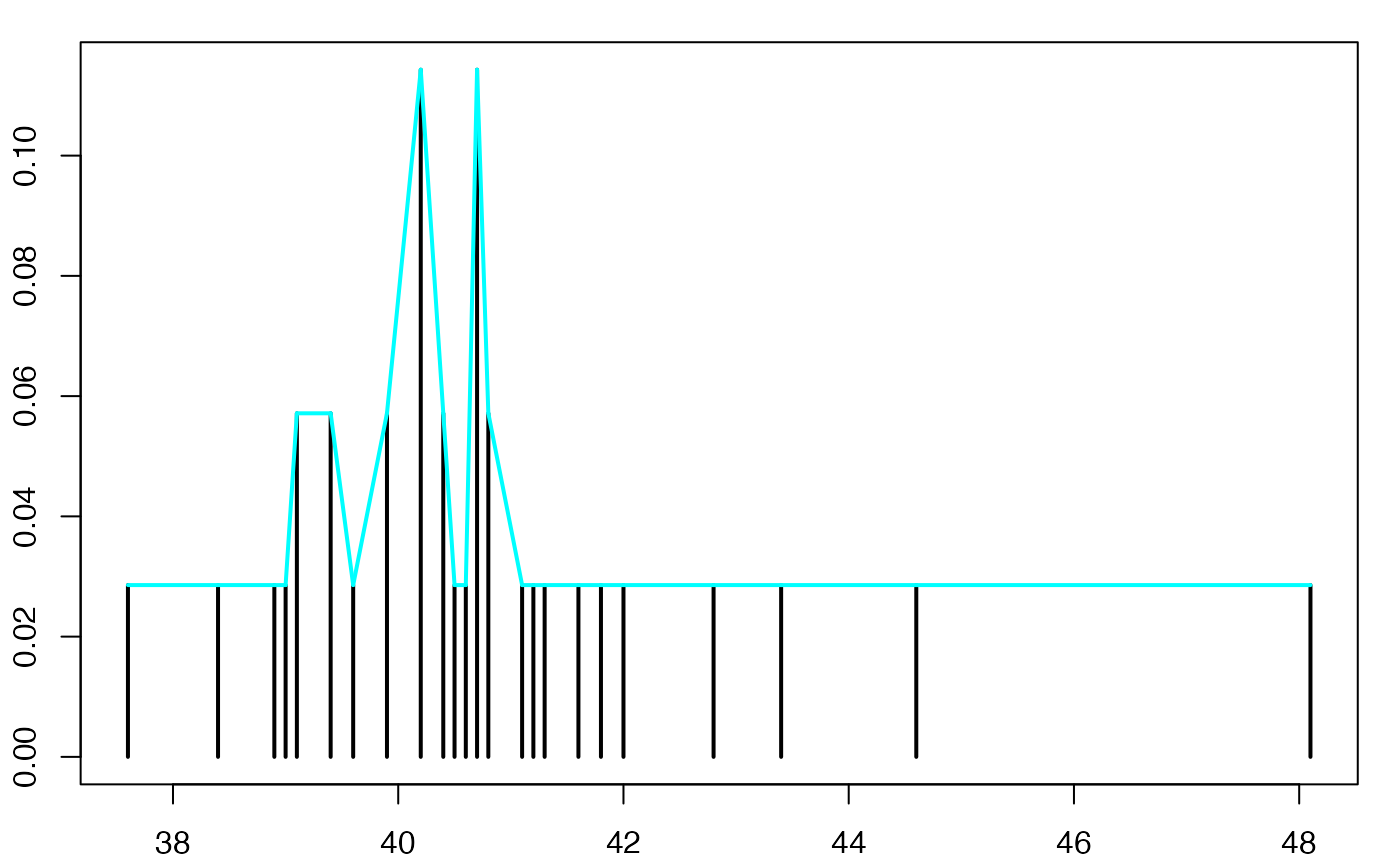
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés
nisum <- rep(ni[1],length(ni))
for (iii in 2:length(ni))
{
nisum[iii] <- nisum[iii-1]+ni[iii]
}
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")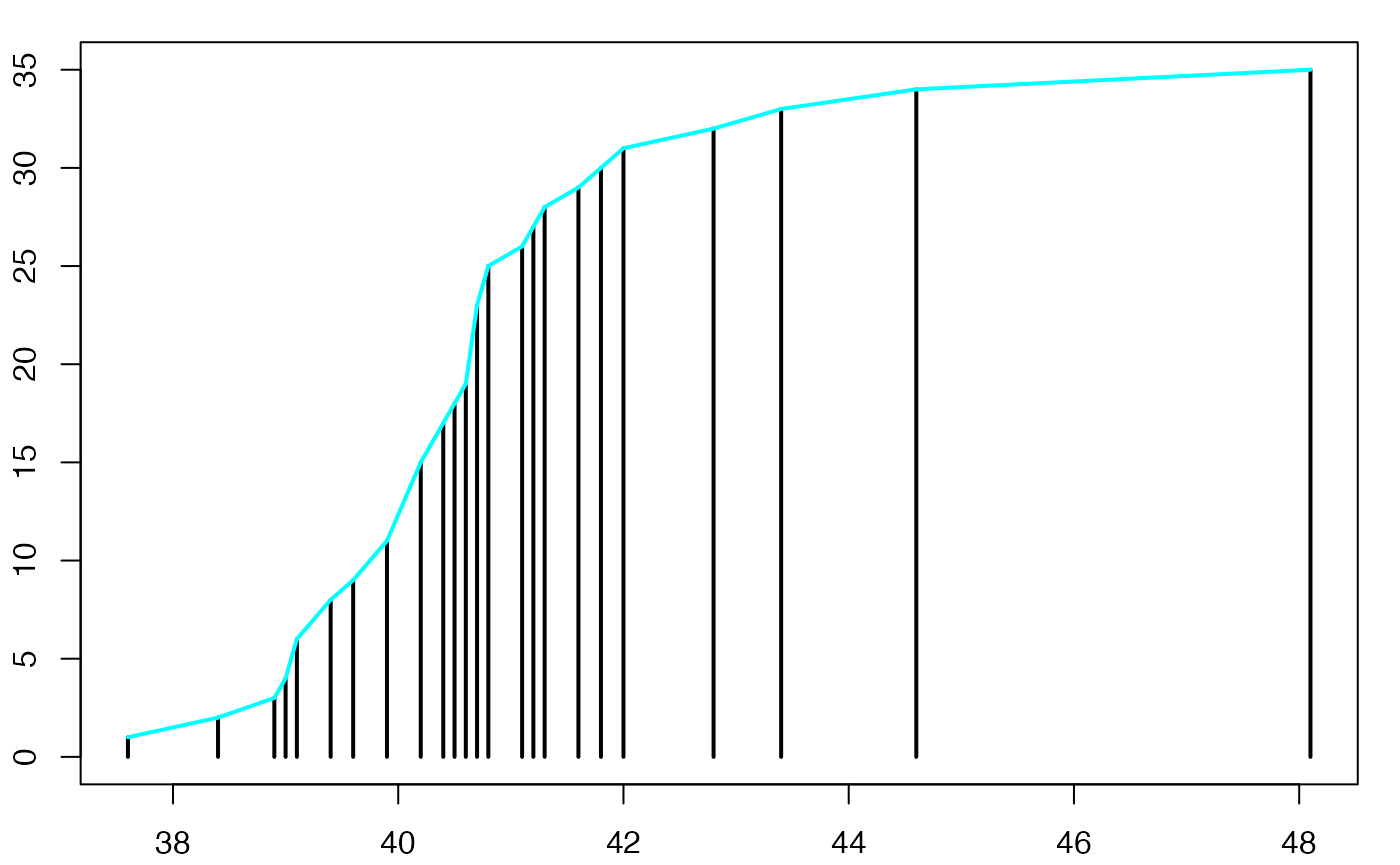
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"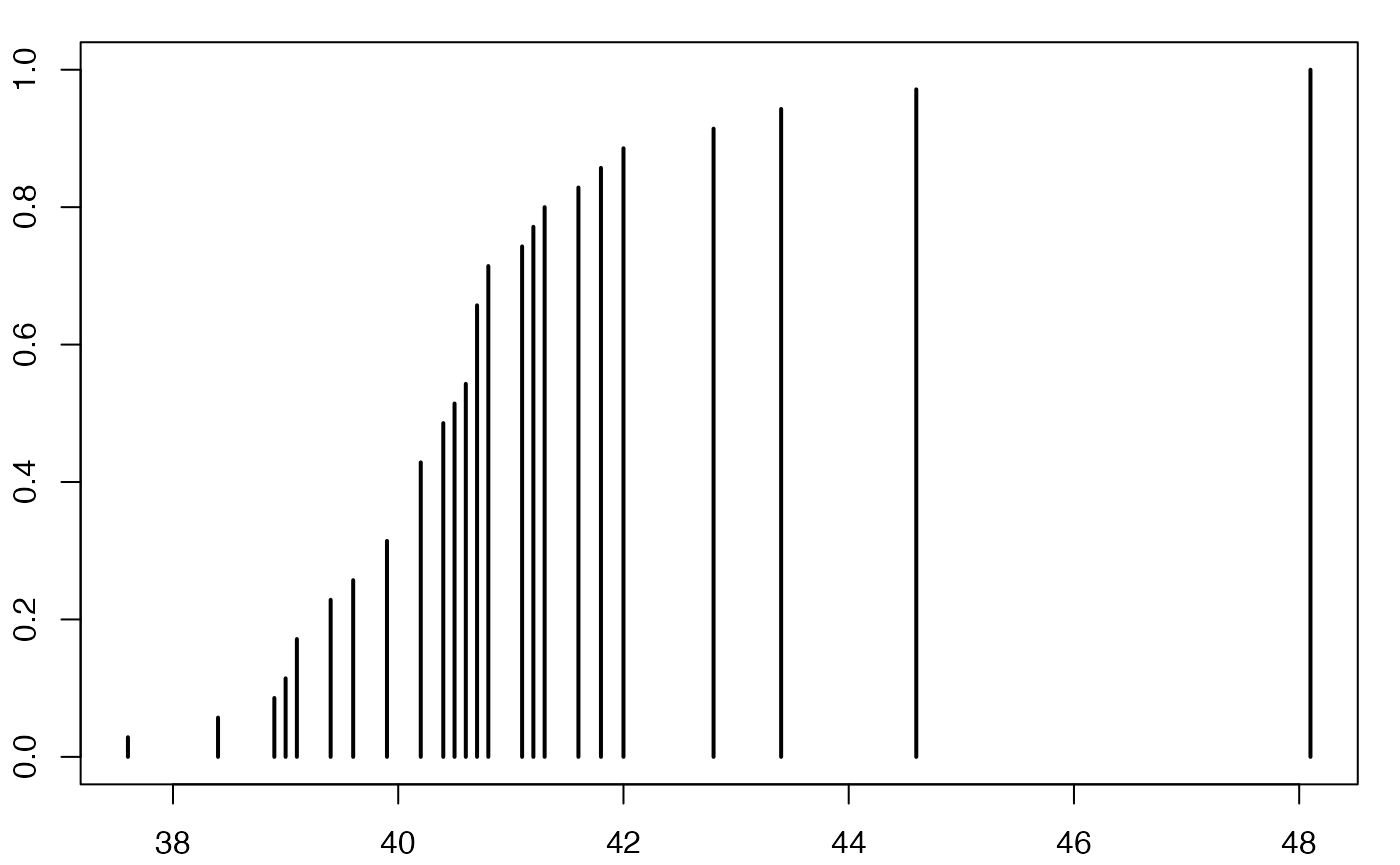
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"frecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"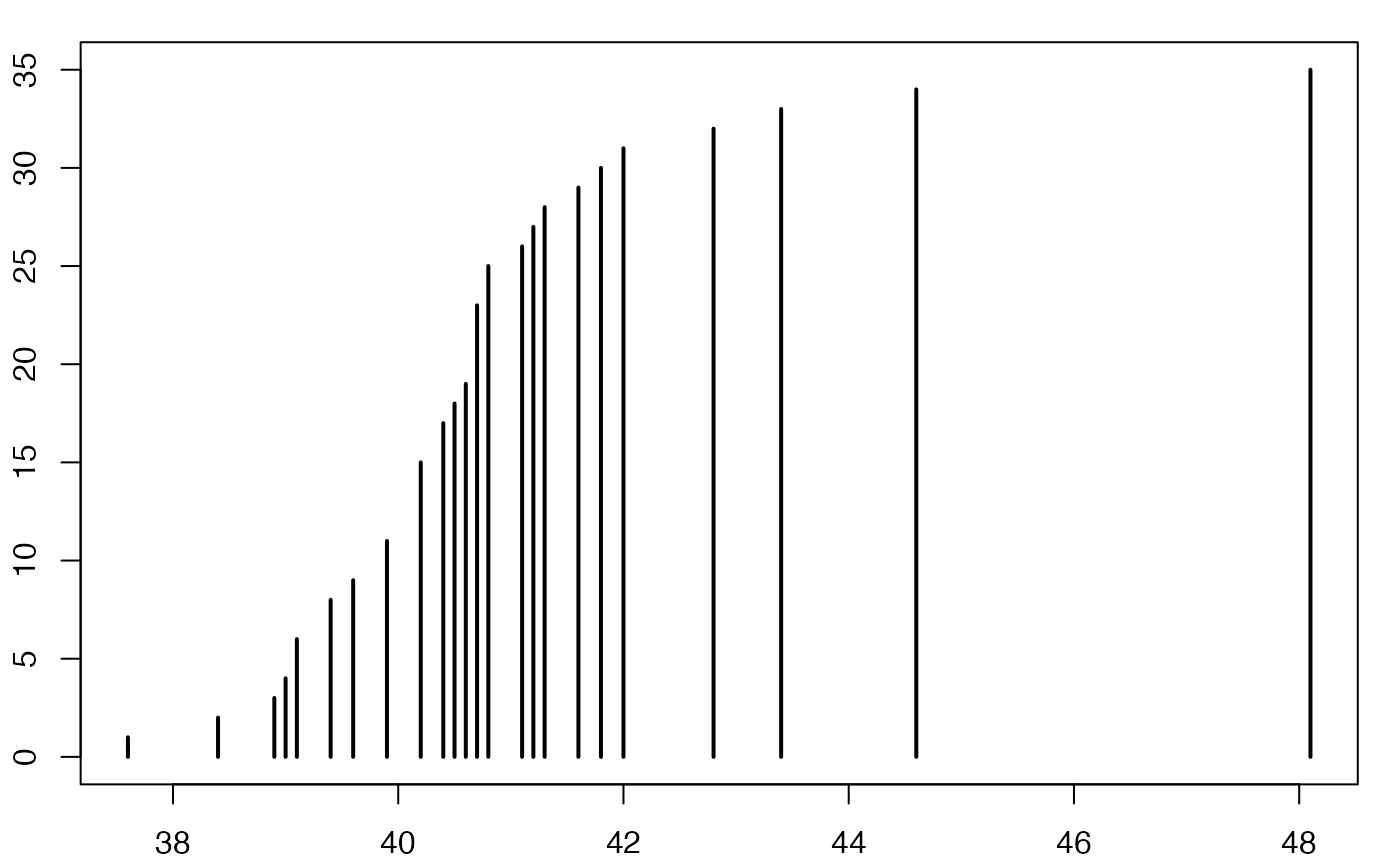
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"effcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Histogramme des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histofrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
dev.off()
#> agg_png
#> 2Histogramme des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulés
data4 <- rep((breaks0[-length(breaks0)]+diff(breaks0)/2)
,cumsum(table(cut(var1,breaks0))))
table(data4)
#> data4
#> 37.5 42.5
#> 11 34
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histofrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulées et polygone des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulées et polygone des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des effectifs cumulés et polygone des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopoleffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
dev.off()
#> agg_png
#> 2Europe Non-salariés
data(Europe)
var1<- Europe$NonSalariés
varN <- "_NonSal"
xi <- unique(sort(var1))
ni <- as.vector(table(var1))
data2 <- cbind(xi,ni)Diagramme en bâtons des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
#axis(1, at = 1:6, labels = c(1:5,6))
#axis(1, at = 1:6, labels = c(1:5,"6 et plus"))
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"batoneffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"batonfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs et polygone des effectifs
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")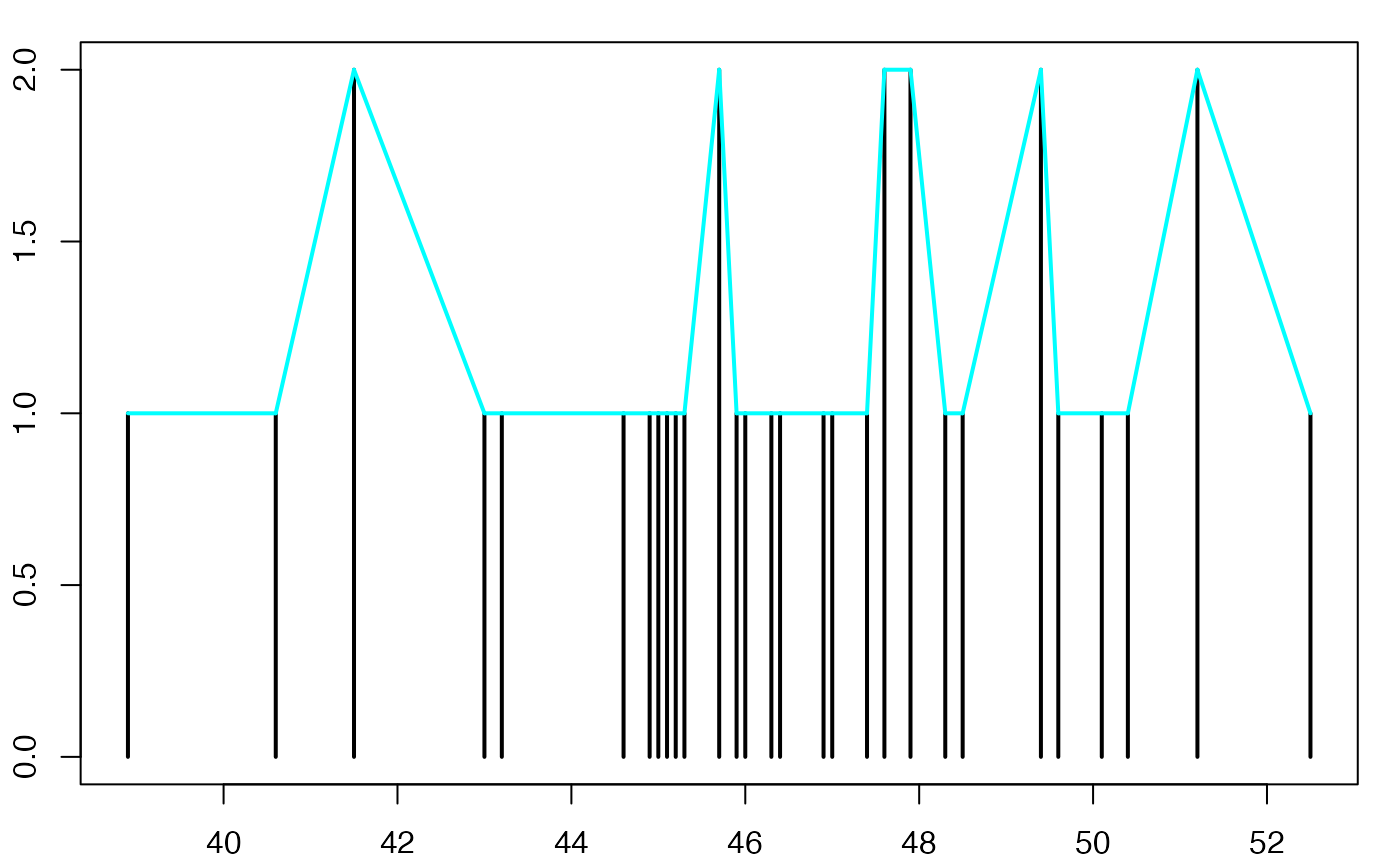
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs et polygone des effectifs", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni))) #,xaxt="n"
segments(xi[-length(xi)],ni[-length(xi)],xi[-1],ni[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")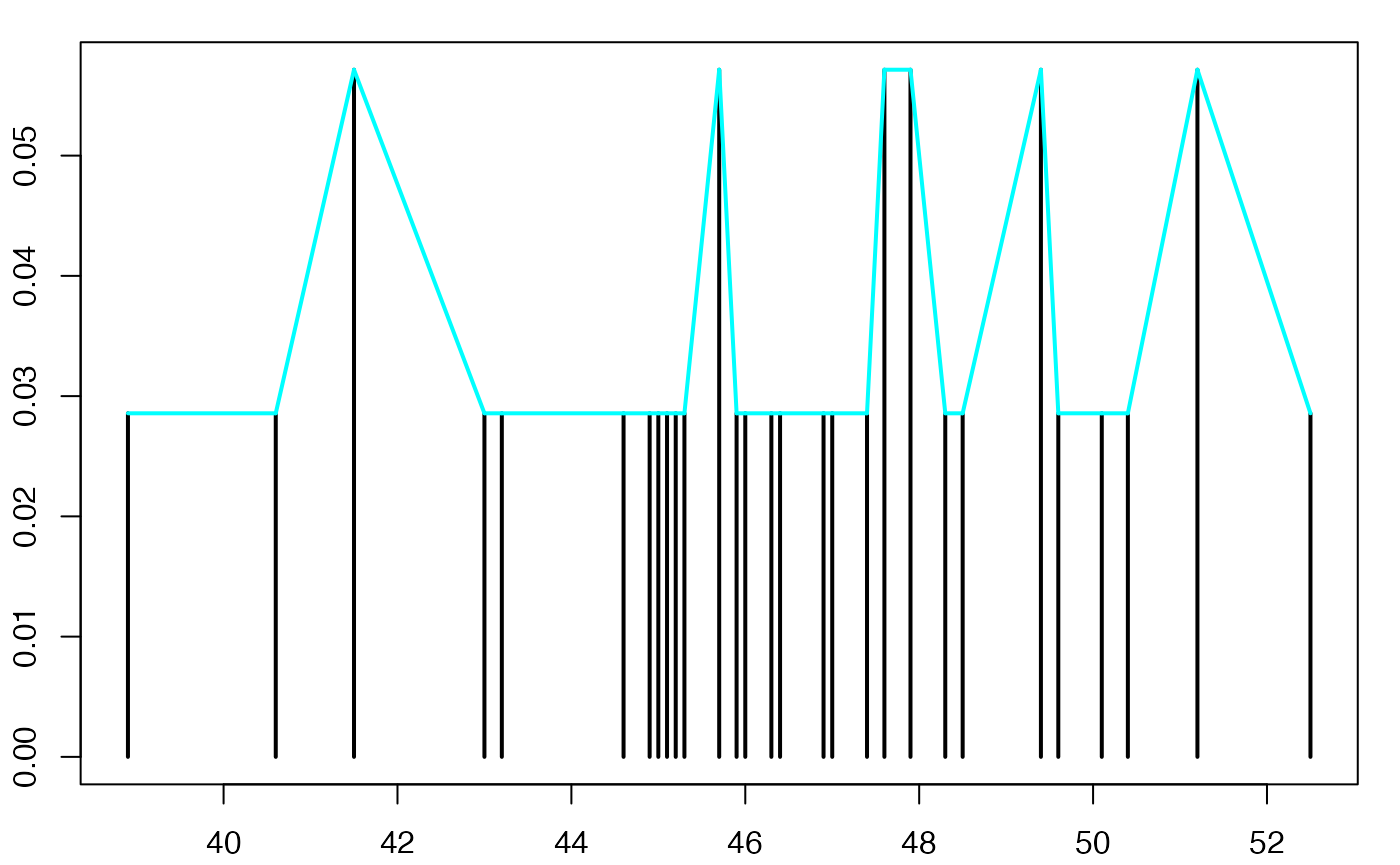
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,ni/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(ni/sum(ni))))
segments(xi[-length(xi)],ni[-length(xi)]/sum(ni),xi[-1],ni[-1]/sum(ni),lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés
nisum <- rep(ni[1],length(ni))
for (iii in 2:length(ni))
{
nisum[iii] <- nisum[iii-1]+ni[iii]
}
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")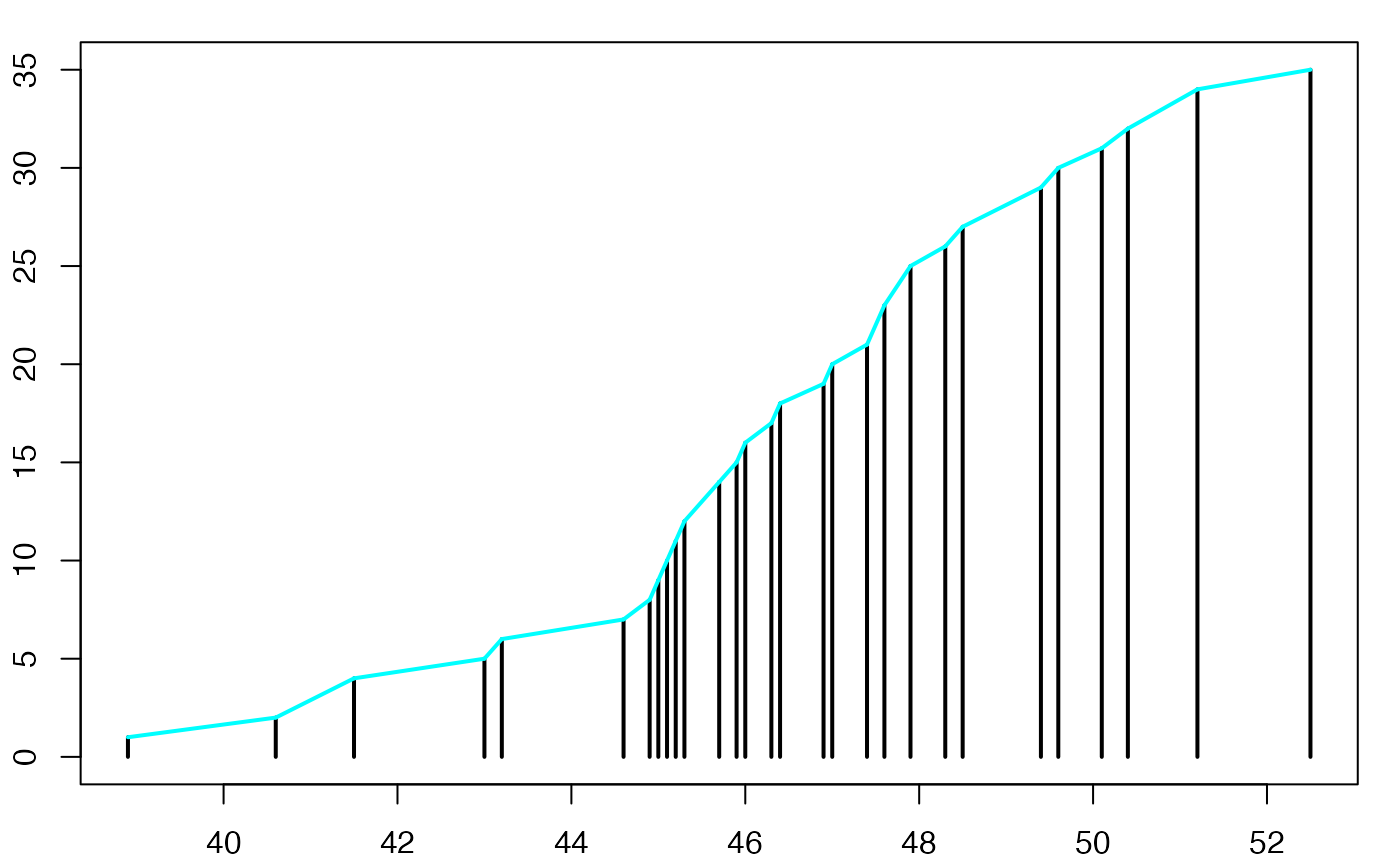
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"polyeffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
segments(xi[-length(xi)],nisum[-length(xi)],xi[-1],nisum[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Diagramme en bâtons des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"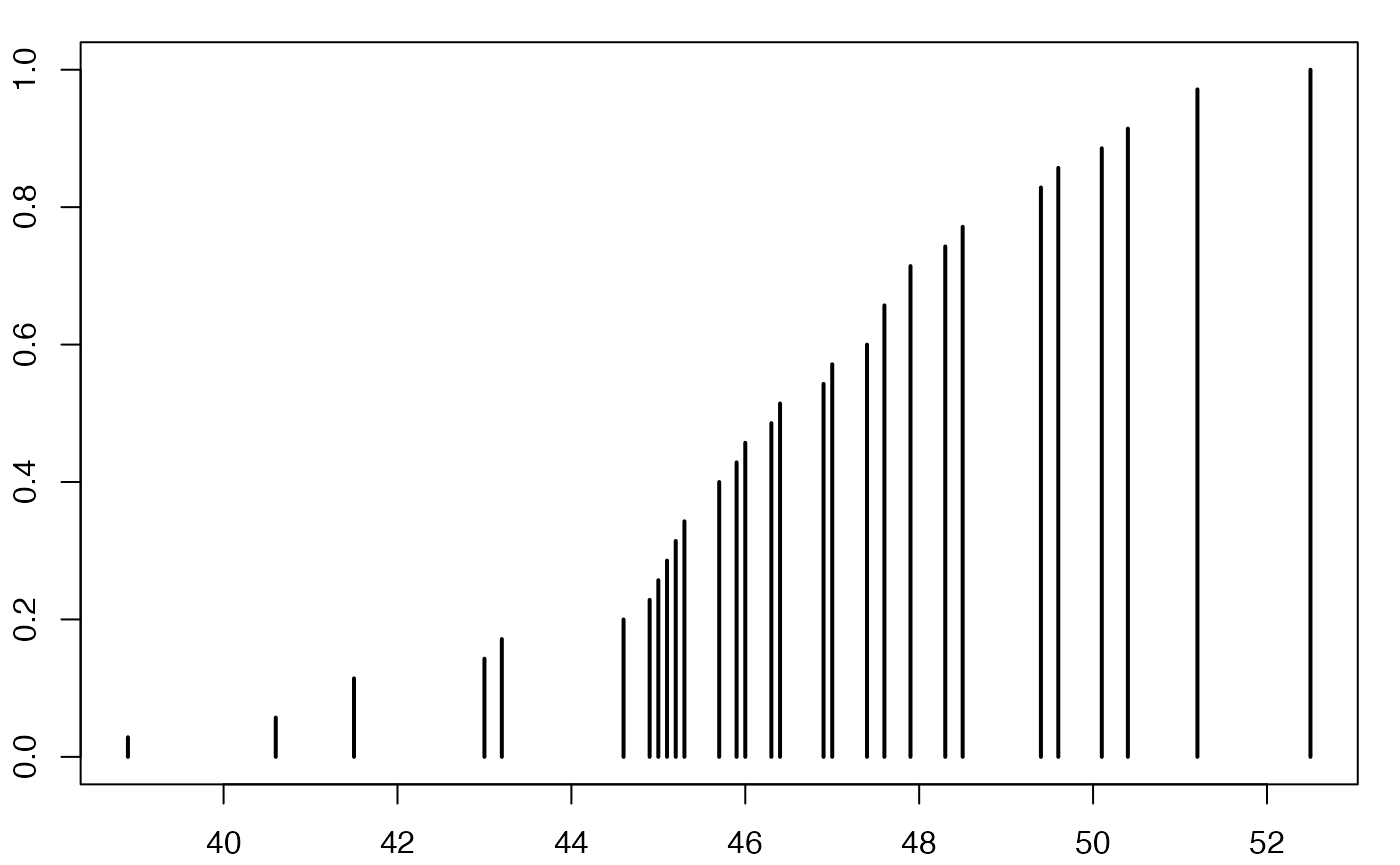
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"frecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum/sum(ni),type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum/sum(ni)))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Diagramme en bâtons des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"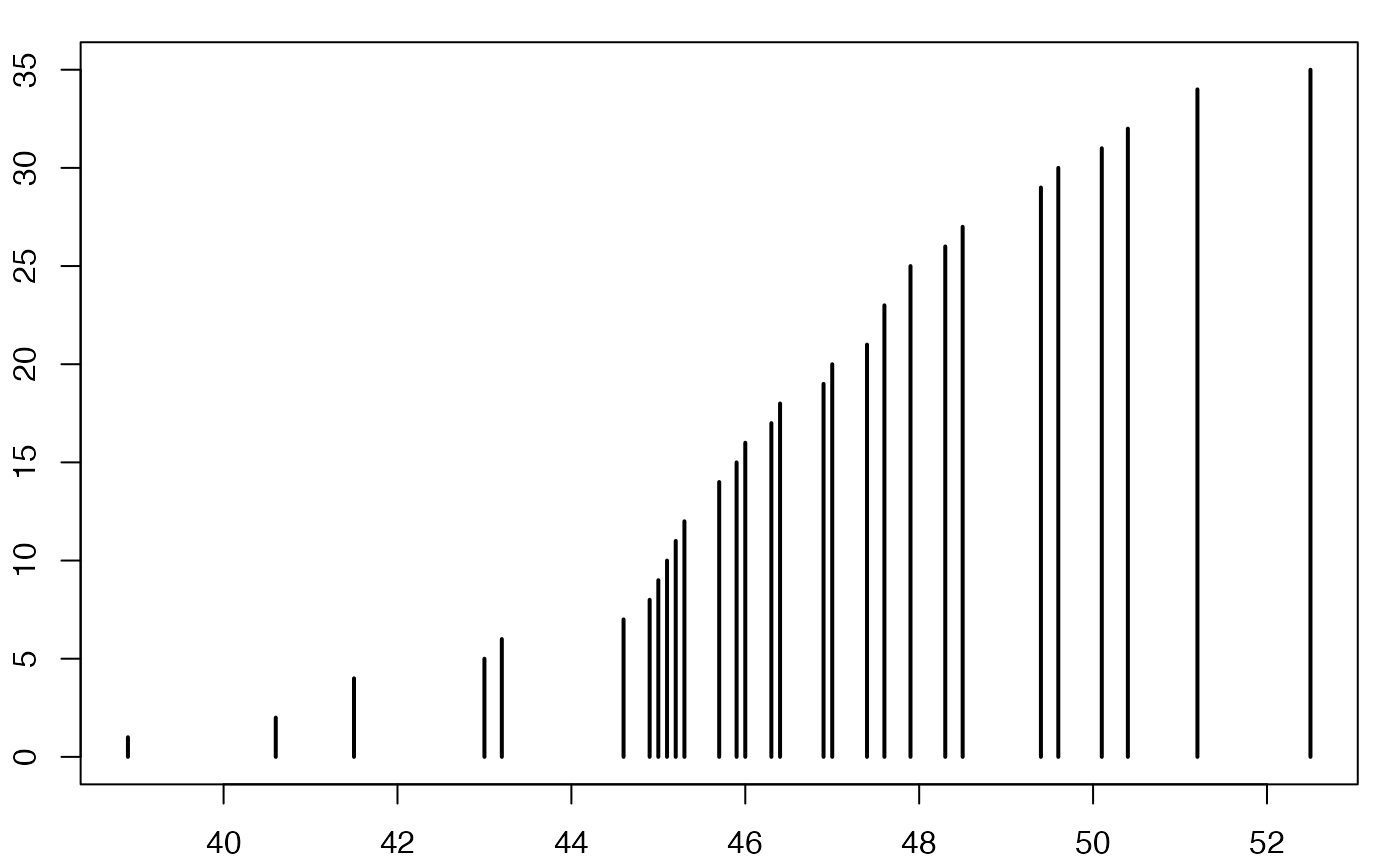
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"effcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Diagramme en bâtons des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
plot(xi,nisum,type='h',xlab="",ylab="",lwd=2,ylim=c(0,max(nisum))) #,xaxt="n"
dev.off()
#> agg_png
#> 2Histogramme des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)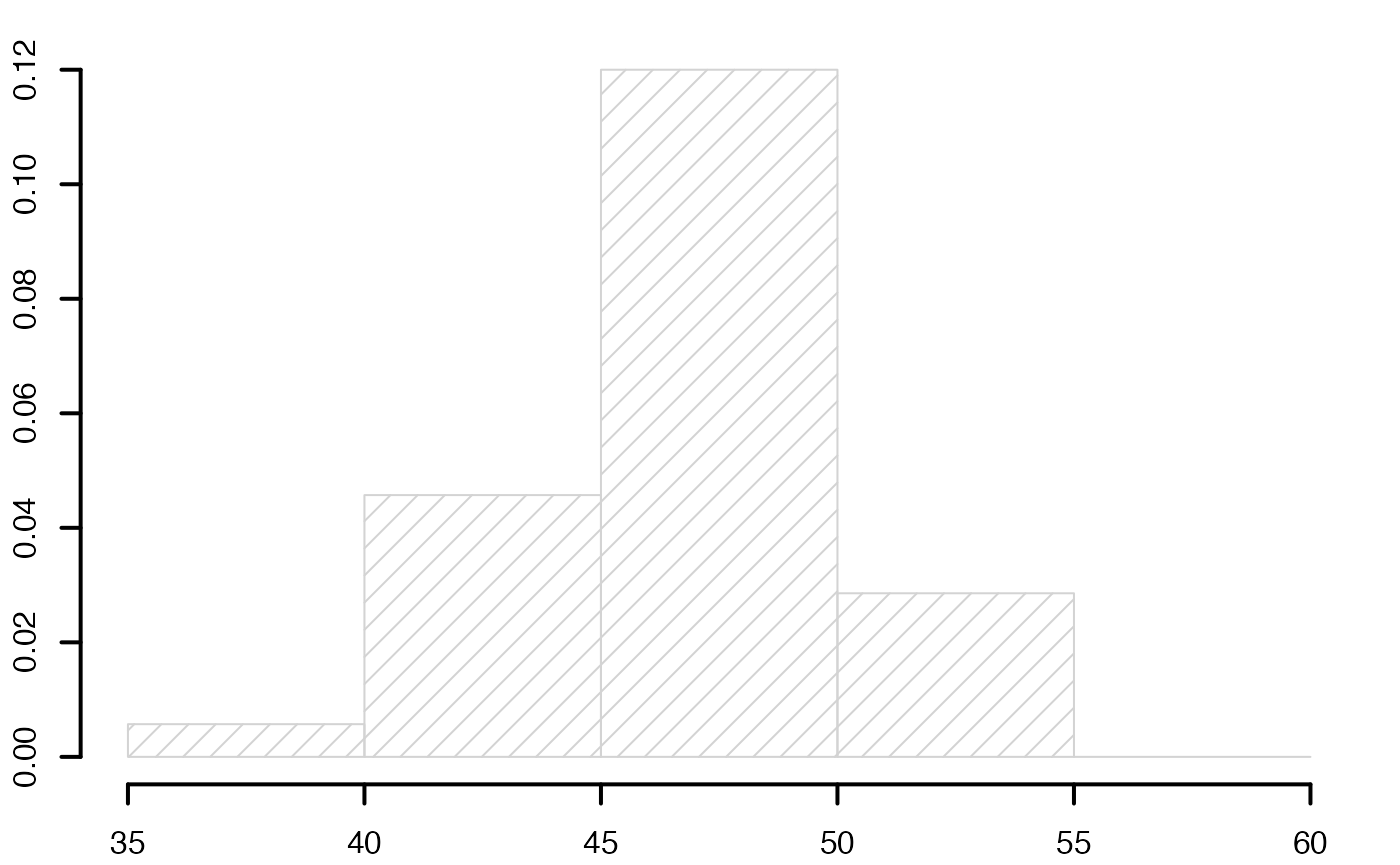
#suppressWarnings(par(oldpar))
Chemin <- "~/Documents/Recherche/DeBoeck/Graphes/Donnees/"
colmodel="cmyk"
pdf(file = paste(Chemin,"histofrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
dev.off()
#> agg_png
#> 2Histogramme des fréquences et polygone des fréquences
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")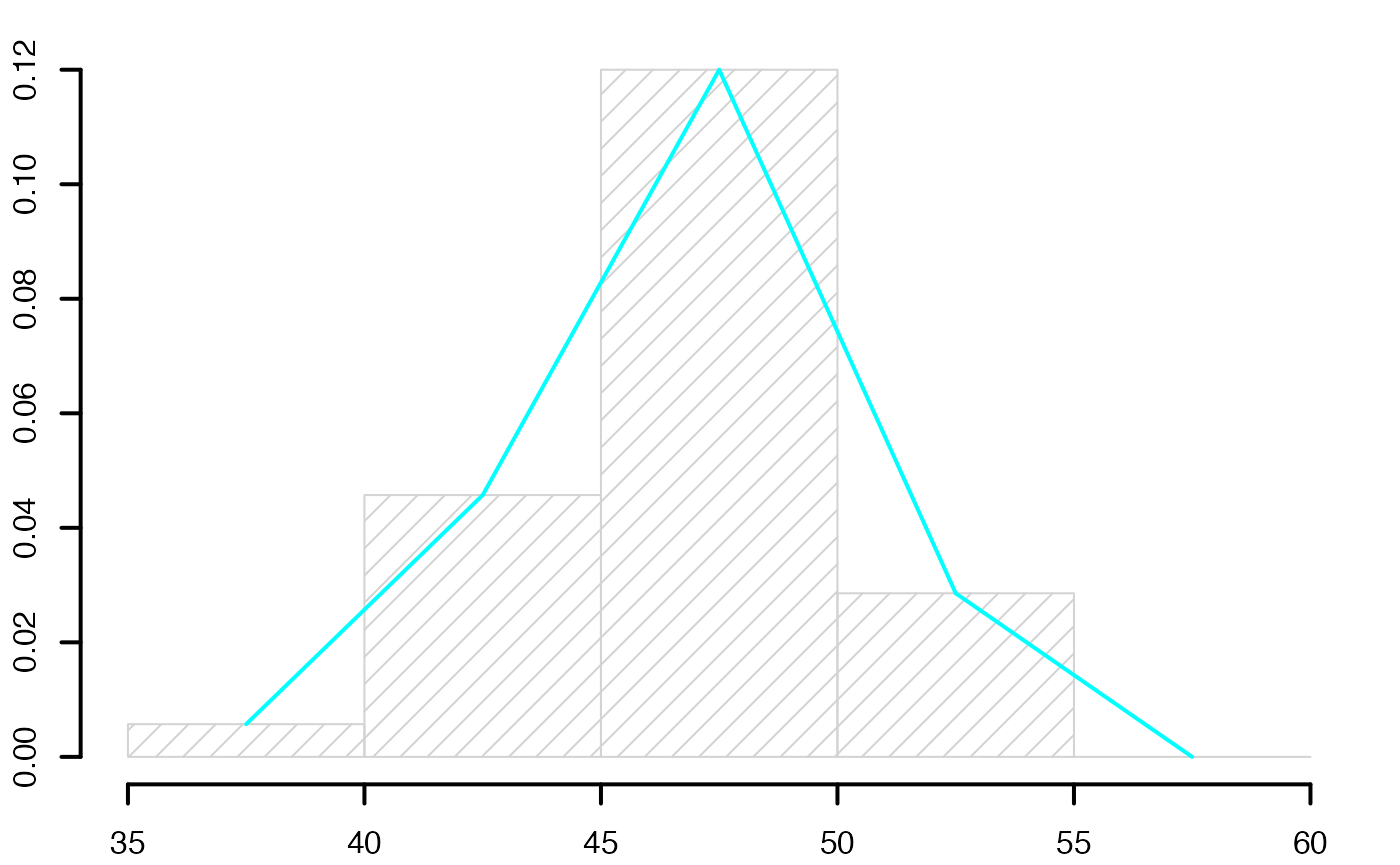
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrenew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences et polygone des fréquences", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofre <- hist(data3,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,density=10)
segments(histofre$mids[-(length(breaks0)-1)],histofre$density[-(length(breaks0)-1)],histofre$mids[-1],histofre$density[-1],lwd=2,col="#00FFFF")
dev.off()
#> agg_png
#> 2Histogramme des effectifs cumulés et polygone des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopoleffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulés
data4 <- rep((breaks0[-length(breaks0)]+diff(breaks0)/2)
,cumsum(table(cut(var1,breaks0))))
table(data4)
#> data4
#> 37.5 42.5 47.5 52.5 57.5
#> 1 9 30 35 35
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histofrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des fréquences cumulées et polygone des fréquences cumulées
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)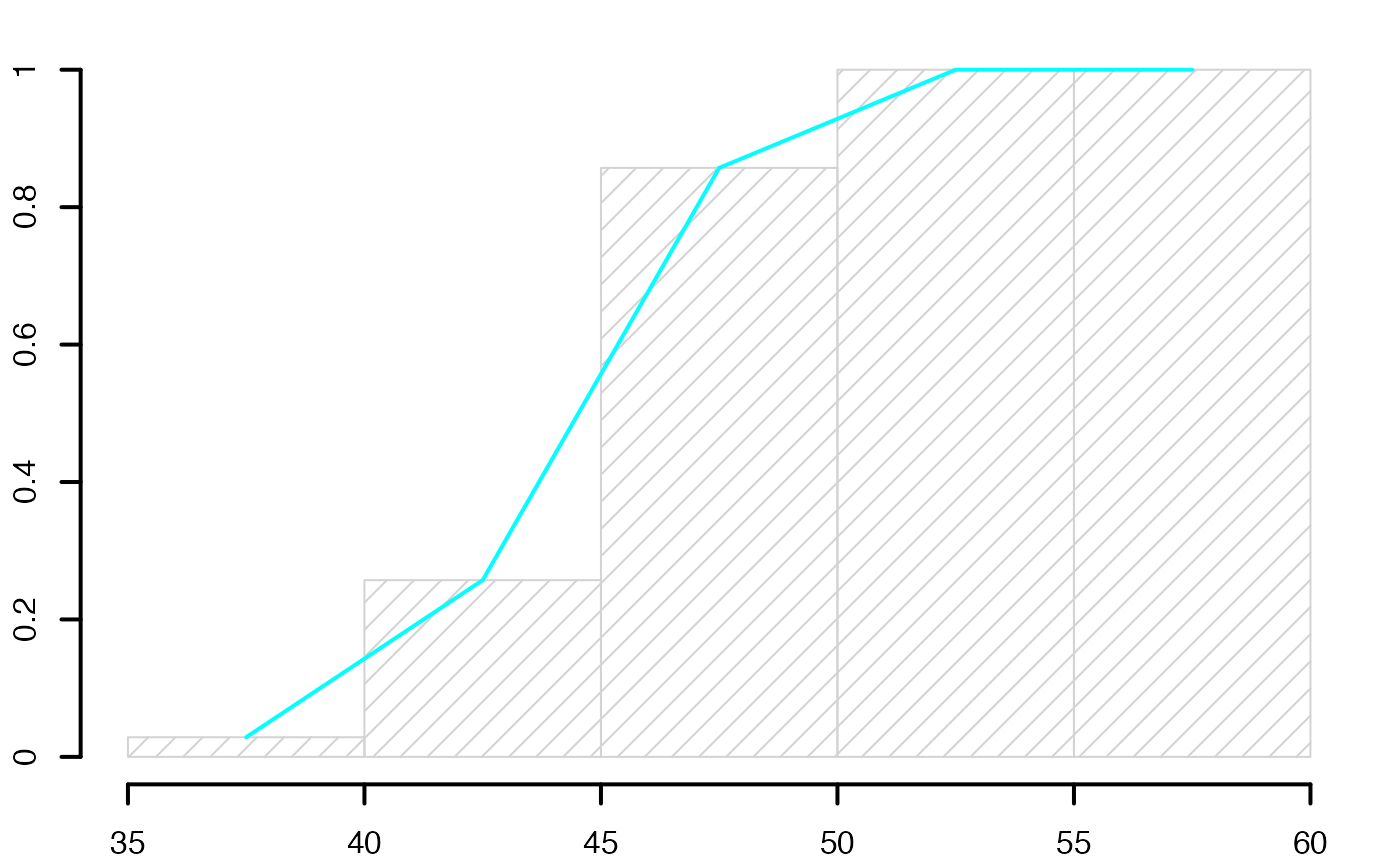
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopolfrecumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des fréquences cumulées et polygone des fréquences cumulées", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histofrecum <- hist(data4,breaks=breaks0,main="",freq=F,xlab="",ylab="",lwd=2,yaxt="n",density=10)
segments(histofrecum$mids[-(length(breaks0)-1)],histofrecum$density[-(length(breaks0)-1)],histofrecum$mids[-1],histofrecum$density[-1],lwd=2,col="#00FFFF")
axis(2,at=c(0,.2,.4,.6,.8,1)*max(histofrecum$dens),labels=c(0,.2,.4,.6,.8,1),lwd=2)
dev.off()
#> agg_png
#> 2Histogramme des effectifs cumulés et polygone des effectifs cumulés
oldpar <- par()
par(mar = c(2, 2, 1, 1) + 0.1, mgp=c(2,1,0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")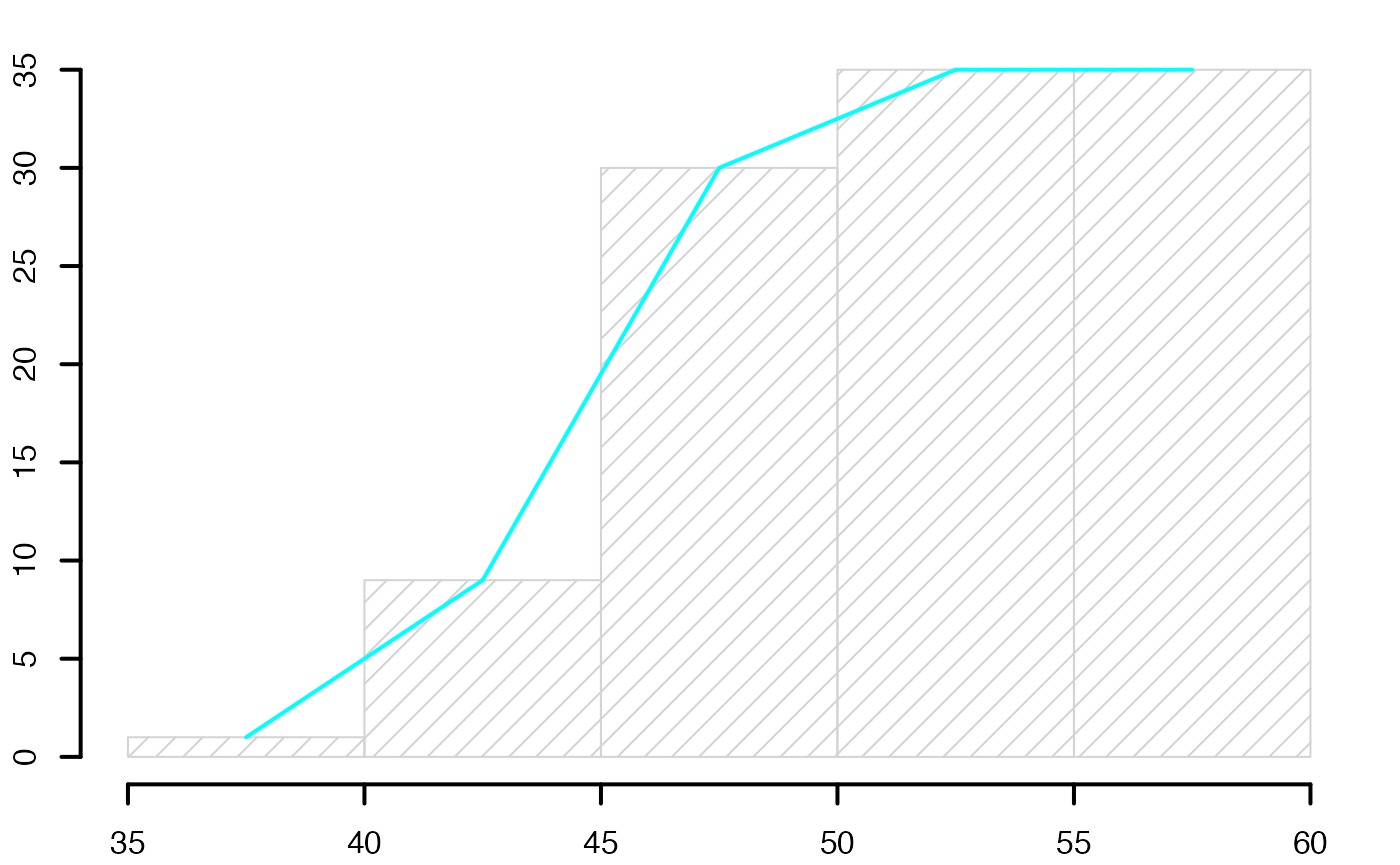
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
#suppressWarnings(par(oldpar))
pdf(file = paste(Chemin,"histopoleffcumnew",varN,".pdf",sep=""),
width = 8, height = 7, onefile = TRUE, family = "Helvetica",
title = "Histogramme des effectifs cumulés et polygone des effectifs cumulés", paper = "special", colormodel = colmodel)
par(mar = c(2, 2, 1, 1) + 0.1, mgp = c(2, 1, 0))
histoeffcum <- hist(data4,breaks=breaks0,main="",freq=T,xlab="",ylab="",lwd=2,density=10) #,yaxt="n"
segments(histoeffcum$mids[-(length(breaks0)-1)],histoeffcum$counts[-(length(breaks0)-1)],histoeffcum$mids[-1],histoeffcum$counts[-1],lwd=2,col="#00FFFF")
#axis(2,at=c(0,.2,.4,.6,.8,1)*length(data3),labels=c(0,.2,.4,.6,.8,1)*length(data3),lwd=2)
dev.off()
#> agg_png
#> 2Analyse du tableau Europe emploi
data(Europe)Paramètres de position
format(cbind(Moy=colMeans(Europe[,c(2,3,4,6,5)]), Q1=
c(quantile(Europe[,2],type=6)[2],quantile(Europe[,3],type=6)[2],quantile(Europe[,4],type=6)[2],quantile(Europe[,6],type=6)[2],quantile(Europe[,5],type=6)[2]), Q2=
c(quantile(Europe[,2],type=6)[3],quantile(Europe[,3],type=6)[3],quantile(Europe[,4],type=6)[3],quantile(Europe[,6],type=6)[3],quantile(Europe[,5],type=6)[3]), Q3=
c(quantile(Europe[,2],type=6)[4],quantile(Europe[,3],type=6)[4],quantile(Europe[,4],type=6)[4],quantile(Europe[,6],type=6)[4],quantile(Europe[,5],type=6)[4])),digits=3)
#> Moy Q1 Q2 Q3
#> Partiel_Ens "15.01" " 6.30" "11.30" "22.50"
#> Partiel_H " 8.03" " 4.70" " 6.60" "10.10"
#> Partiel_F "23.09" " 9.30" "17.00" "33.90"
#> NonSalariés "46.51" "45.00" "46.40" "48.50"
#> Salariés "40.72" "39.60" "40.50" "41.20"Paramètres de dispersion
tab2=cbind(
sd=c(sqrt(1/length(Europe[,2])*sum((Europe[,2]-mean(Europe[,2]))^2)),
sqrt(1/length(Europe[,3])*sum((Europe[,3]-mean(Europe[,3]))^2)),
sqrt(1/length(Europe[,4])*sum((Europe[,4]-mean(Europe[,4]))^2)),
sqrt(1/length(Europe[,6])*sum((Europe[,6]-mean(Europe[,6]))^2)),
sqrt(1/length(Europe[,5])*sum((Europe[,5]-mean(Europe[,5]))^2))
),
mad=c(quantile(abs(Europe[,2]-quantile(Europe[,2],type=6)[3]),type=6)[3],
quantile(abs(Europe[,3]-quantile(Europe[,3],type=6)[3]),type=6)[3],
quantile(abs(Europe[,4]-quantile(Europe[,4],type=6)[3]),type=6)[3],
quantile(abs(Europe[,6]-quantile(Europe[,6],type=6)[3]),type=6)[3],
quantile(abs(Europe[,5]-quantile(Europe[,5],type=6)[3]),type=6)[3]
),
Range=c(diff(range(Europe[,2])),
diff(range(Europe[,3])),
diff(range(Europe[,4])),
diff(range(Europe[,6])),
diff(range(Europe[,5])))
,
EIQ=c(quantile(Europe[,2],type=6)[4],quantile(Europe[,3],type=6)[4],quantile(Europe[,4],type=6)[4],quantile(Europe[,6],type=6)[4],quantile(Europe[,5],type=6)[4])-c(quantile(Europe[,2],type=6)[2],quantile(Europe[,3],type=6)[2],quantile(Europe[,4],type=6)[2],quantile(Europe[,6],type=6)[2],quantile(Europe[,5],type=6)[2])
,
CV=c(sqrt(1/length(Europe[,2])*sum((Europe[,2]-mean(Europe[,2]))^2)),
sqrt(1/length(Europe[,3])*sum((Europe[,3]-mean(Europe[,3]))^2)),
sqrt(1/length(Europe[,4])*sum((Europe[,4]-mean(Europe[,4]))^2)),
sqrt(1/length(Europe[,6])*sum((Europe[,6]-mean(Europe[,6]))^2)),
sqrt(1/length(Europe[,5])*sum((Europe[,5]-mean(Europe[,5]))^2)))/colMeans(Europe[,c(2,3,4,6,5)])*100)
rownames(tab2) <- colnames(Europe)[c(2,3,4,6,5)]
round(tab2,digits=3)
#> sd mad Range EIQ CV
#> Partiel_Ens 10.559 6.5 48.3 16.2 70.364
#> Partiel_H 5.071 2.9 26.2 5.4 63.184
#> Partiel_F 17.188 10.8 73.1 24.6 74.426
#> NonSalariés 3.074 1.5 13.6 3.5 6.610
#> Salariés 1.847 0.7 10.5 1.6 4.536Paramètres de forme
nn=length(Europe[,2])
tab5=
cbind(
AsyFis=
c(
1/nn*sum((Europe[,2]-mean(Europe[,2]))^3)/(1/nn*sum((Europe[,2]-mean(Europe[,2]))^2))^{3/2},
1/nn*sum((Europe[,3]-mean(Europe[,3]))^3)/(1/nn*sum((Europe[,3]-mean(Europe[,3]))^2))^{3/2},
1/nn*sum((Europe[,4]-mean(Europe[,4]))^3)/(1/nn*sum((Europe[,4]-mean(Europe[,4]))^2))^{3/2},
1/nn*sum((Europe[,5]-mean(Europe[,5]))^3)/(1/nn*sum((Europe[,5]-mean(Europe[,5]))^2))^{3/2},
1/nn*sum((Europe[,6]-mean(Europe[,6]))^3)/(1/nn*sum((Europe[,6]-mean(Europe[,6]))^2))^{3/2}
)
,
AsyPea=
c(
1/nn*sum((Europe[,2]-mean(Europe[,2]))^3)/(1/nn*sum((Europe[,2]-mean(Europe[,2]))^2))^{3/2},
1/nn*sum((Europe[,3]-mean(Europe[,3]))^3)/(1/nn*sum((Europe[,3]-mean(Europe[,3]))^2))^{3/2},
1/nn*sum((Europe[,4]-mean(Europe[,4]))^3)/(1/nn*sum((Europe[,4]-mean(Europe[,4]))^2))^{3/2},
1/nn*sum((Europe[,5]-mean(Europe[,5]))^3)/(1/nn*sum((Europe[,5]-mean(Europe[,5]))^2))^{3/2},
1/nn*sum((Europe[,6]-mean(Europe[,6]))^3)/(1/nn*sum((Europe[,6]-mean(Europe[,6]))^2))^{3/2}
)^2
,
AppFis=
c(
1/nn*sum((Europe[,2]-mean(Europe[,2]))^4)/(1/nn*sum((Europe[,2]-mean(Europe[,2]))^2))^{2},
1/nn*sum((Europe[,3]-mean(Europe[,3]))^4)/(1/nn*sum((Europe[,3]-mean(Europe[,3]))^2))^{2},
1/nn*sum((Europe[,4]-mean(Europe[,4]))^4)/(1/nn*sum((Europe[,4]-mean(Europe[,4]))^2))^{2},
1/nn*sum((Europe[,5]-mean(Europe[,5]))^4)/(1/nn*sum((Europe[,5]-mean(Europe[,5]))^2))^{2},
1/nn*sum((Europe[,6]-mean(Europe[,6]))^4)/(1/nn*sum((Europe[,6]-mean(Europe[,6]))^2))^{2}
)-3
,
AppPea=
c(
1/nn*sum((Europe[,2]-mean(Europe[,2]))^4)/(1/nn*sum((Europe[,2]-mean(Europe[,2]))^2))^{2},
1/nn*sum((Europe[,3]-mean(Europe[,3]))^4)/(1/nn*sum((Europe[,3]-mean(Europe[,3]))^2))^{2},
1/nn*sum((Europe[,4]-mean(Europe[,4]))^4)/(1/nn*sum((Europe[,4]-mean(Europe[,4]))^2))^{2},
1/nn*sum((Europe[,5]-mean(Europe[,5]))^4)/(1/nn*sum((Europe[,5]-mean(Europe[,5]))^2))^{2},
1/nn*sum((Europe[,6]-mean(Europe[,6]))^4)/(1/nn*sum((Europe[,6]-mean(Europe[,6]))^2))^{2}
)
)Analyse du tableau Europe emploi après regroupement des données
Création des groupes
Création naïve des classes
distg1<-table(cut(Europe$Partiel_Ens,c(0,10,20,30,40,50,60,70,80)))
distg2<-table(cut(Europe$Partiel_H,c(0,10,20,30,40,50,60,70,80)))
distg3<-table(cut(Europe$Partiel_F,c(0,10,20,30,40,50,60,70,80)))
distg1
#>
#> (0,10] (10,20] (20,30] (30,40] (40,50] (50,60] (60,70] (70,80]
#> 16 9 8 1 0 1 0 0
distg2
#>
#> (0,10] (10,20] (20,30] (30,40] (40,50] (50,60] (60,70] (70,80]
#> 25 9 1 0 0 0 0 0
distg3
#>
#> (0,10] (10,20] (20,30] (30,40] (40,50] (50,60] (60,70] (70,80]
#> 9 9 4 8 3 0 1 1
#Centres des classes
vald123<-(c(0,10,20,30,40,50,60,70,80)[-9]+c(0,10,20,30,40,50,60,70,80)[-1])/2
distg4<-table(cut(Europe$Salariés,c(35,40,45,50,55,60)))
distg5<-table(cut(Europe$NonSalariés,c(35,40,45,50,55,60)))
distg4
#>
#> (35,40] (40,45] (45,50] (50,55] (55,60]
#> 11 23 1 0 0
distg5
#>
#> (35,40] (40,45] (45,50] (50,55] (55,60]
#> 1 8 21 5 0
#Centres des classes
vald45<-(c(35,40,45,50,55,60)[-6]+c(35,40,45,50,55,60)[-1])/2Utilisation du package actuar
if(!("actuar" %in% installed.packages())){install.packages("actuar")}
library(actuar)
x1 <- grouped.data(Partiel_Ens=Europe$Partiel_Ens,breaks=c(0,10,20,30,40,50,60,70,80))
x2 <- grouped.data(Partiel_Ens=Europe$Partiel_H,breaks=c(0,10,20,30,40,50,60,70,80))
x3 <- grouped.data(Partiel_Ens=Europe$Partiel_F,breaks=c(0,10,20,30,40,50,60,70,80))
x4 <- grouped.data(Salariés=Europe$Salariés,breaks=c(0,10,20,30,40,50,60,70,80))
x5 <- grouped.data(NonSalariés=Europe$NonSalariés,breaks=c(0,10,20,30,40,50,60,70,80))
cbind(x1,x2,x3)
#> Var.1 Partiel_Ens Var.3 Partiel_Ens Var.5 Partiel_Ens
#> 1 (0, 10] 16 (0, 10] 25 (0, 10] 9
#> 2 (10, 20] 9 (10, 20] 9 (10, 20] 9
#> 3 (20, 30] 8 (20, 30] 1 (20, 30] 4
#> 4 (30, 40] 1 (30, 40] 0 (30, 40] 8
#> 5 (40, 50] 0 (40, 50] 0 (40, 50] 3
#> 6 (50, 60] 1 (50, 60] 0 (50, 60] 0
#> 7 (60, 70] 0 (60, 70] 0 (60, 70] 1
#> 8 (70, 80] 0 (70, 80] 0 (70, 80] 1
cbind(x4,x5)
#> Var.1 Salariés Var.3 NonSalariés
#> 1 (0, 10] 0 (0, 10] 0
#> 2 (10, 20] 0 (10, 20] 0
#> 3 (20, 30] 0 (20, 30] 0
#> 4 (30, 40] 11 (30, 40] 1
#> 5 (40, 50] 24 (40, 50] 29
#> 6 (50, 60] 0 (50, 60] 5
#> 7 (60, 70] 0 (60, 70] 0
#> 8 (70, 80] 0 (70, 80] 0Statistiques descriptives groupées
c(
weighted.mean(vald123,distg1),
weighted.mean(vald123,distg2),
weighted.mean(vald123,distg2),
weighted.mean(vald45,distg4),
weighted.mean(vald45,distg5)
)
#> [1] 14.428571 8.142857 8.142857 41.071429 46.785714
tab3=cbind(
c(
mean(x1),
mean(x2),
mean(x3),
mean(x4),
mean(x5)
),
rbind(quantile(x1),quantile(x2),quantile(x3),quantile(x4),quantile(x5))[,c(2,3,4)]
)
round(c(
mean(x1),
mean(x2),
mean(x3),
mean(x4),
mean(x5)
),digits=2)
#> Partiel_Ens Partiel_Ens Partiel_Ens Salariés NonSalariés
#> 14.43 8.14 23.86 41.86 46.14
round( rbind(quantile(x1),quantile(x2),quantile(x3),quantile(x4),quantile(x5))[,c(2,3,4)],digits=2)
#> 25% 50% 75%
#> [1,] 5.47 11.67 21.56
#> [2,] 3.50 7.00 11.39
#> [3,] 9.72 19.44 35.31
#> [4,] 37.95 42.71 46.35
#> [5,] 42.67 45.69 48.71
round(tab3,digits=2)
#> 25% 50% 75%
#> Partiel_Ens 14.43 5.47 11.67 21.56
#> Partiel_Ens 8.14 3.50 7.00 11.39
#> Partiel_Ens 23.86 9.72 19.44 35.31
#> Salariés 41.86 37.95 42.71 46.35
#> NonSalariés 46.14 42.67 45.69 48.71
tab4=cbind(
sigma=c(
sqrt(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])-1/12*weighted.mean(diff(x1[,1])^2,x1[,2])),
sqrt(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])-1/12*weighted.mean(diff(x2[,1])^2,x2[,2])),
sqrt(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])-1/12*weighted.mean(diff(x3[,1])^2,x3[,2])),
sqrt(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])-1/12*weighted.mean(diff(x4[,1])^2,x4[,2])),
sqrt(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])-1/12*weighted.mean(diff(x5[,1])^2,x5[,2]))
),
EIQ=t(diff(t(rbind(quantile(x1),quantile(x2),quantile(x3),quantile(x4),quantile(x5))[,c(2,4)]))),
CV=c(
sqrt(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])-1/12*weighted.mean(diff(x1[,1])^2,x1[,2]))/mean(x1)*100,
sqrt(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])-1/12*weighted.mean(diff(x2[,1])^2,x2[,2]))/mean(x2)*100,
sqrt(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])-1/12*weighted.mean(diff(x3[,1])^2,x3[,2]))/mean(x3)*100,
sqrt(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])-1/12*weighted.mean(diff(x4[,1])^2,x4[,2]))/mean(x4)*100,
sqrt(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])-1/12*weighted.mean(diff(x5[,1])^2,x5[,2]))/mean(x5)*100
))
round(tab4[,1],digits=2)
#> Partiel_Ens Partiel_Ens Partiel_Ens Salariés NonSalariés
#> 10.82 4.35 17.12 3.64 2.74
round(tab4[,2],digits=2)
#> Partiel_Ens Partiel_Ens Partiel_Ens Salariés NonSalariés
#> 16.09 7.89 25.59 8.40 6.03
round(tab4[,3],digits=2)
#> Partiel_Ens Partiel_Ens Partiel_Ens Salariés NonSalariés
#> 74.98 53.43 71.78 8.69 5.94
tab6=cbind(
asyFis=
c(
weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^3,x1[,2])/sqrt(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])-1/12*weighted.mean(diff(x1[,1])^2,x1[,2]))^3,
weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^3,x2[,2])/sqrt(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])-1/12*weighted.mean(diff(x2[,1])^2,x2[,2]))^3,
weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^3,x3[,2])/sqrt(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])-1/12*weighted.mean(diff(x3[,1])^2,x3[,2]))^3,
weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^3,x4[,2])/sqrt(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])-1/12*weighted.mean(diff(x4[,1])^2,x4[,2]))^3,
weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^3,x5[,2])/sqrt(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])-1/12*weighted.mean(diff(x5[,1])^2,x5[,2]))^3
)
,
asyPea=
c(
weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^3,x1[,2])/sqrt(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])-1/12*weighted.mean(diff(x1[,1])^2,x1[,2]))^3,
weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^3,x2[,2])/sqrt(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])-1/12*weighted.mean(diff(x2[,1])^2,x2[,2]))^3,
weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^3,x3[,2])/sqrt(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])-1/12*weighted.mean(diff(x3[,1])^2,x3[,2]))^3,
weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^3,x4[,2])/sqrt(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])-1/12*weighted.mean(diff(x4[,1])^2,x4[,2]))^3,
weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^3,x5[,2])/sqrt(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])-1/12*weighted.mean(diff(x5[,1])^2,x5[,2]))^3
)^2
,
appFis=
c(
(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^4,x1[,2])
-10^2/2*weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])
+7/240*10^4)/sqrt(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])-1/12*weighted.mean(diff(x1[,1])^2,x1[,2]))^4,
(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^4,x2[,2])
-10^2/2*weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])
+7/240*10^4)/sqrt(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])-1/12*weighted.mean(diff(x2[,1])^2,x2[,2]))^4,
(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^4,x3[,2])
-10^2/2*weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])
+7/240*10^4)/sqrt(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])-1/12*weighted.mean(diff(x3[,1])^2,x3[,2]))^4,
(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^4,x4[,2])
-10^2/2*weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])
+7/240*10^4)/sqrt(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])-1/12*weighted.mean(diff(x4[,1])^2,x4[,2]))^4,
(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^4,x5[,2])
-10^2/2*weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])
+7/240*10^4)/sqrt(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])-1/12*weighted.mean(diff(x5[,1])^2,x5[,2]))^4
)-3
,
appPea=
c(
(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^4,x1[,2])
-10^2/2*weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])
+7/240*10^4)/sqrt(weighted.mean(((x1[,1][-1]+x1[,1][-length(x1[,1])])/2-mean(x1))^2,x1[,2])-1/12*weighted.mean(diff(x1[,1])^2,x1[,2]))^4,
(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^4,x2[,2])
-10^2/2*weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])
+7/240*10^4)/sqrt(weighted.mean(((x2[,1][-1]+x2[,1][-length(x2[,1])])/2-mean(x2))^2,x2[,2])-1/12*weighted.mean(diff(x2[,1])^2,x2[,2]))^4,
(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^4,x3[,2])
-10^2/2*weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])
+7/240*10^4)/sqrt(weighted.mean(((x3[,1][-1]+x3[,1][-length(x3[,1])])/2-mean(x3))^2,x3[,2])-1/12*weighted.mean(diff(x3[,1])^2,x3[,2]))^4,
(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^4,x4[,2])
-10^2/2*weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])
+7/240*10^4)/sqrt(weighted.mean(((x4[,1][-1]+x4[,1][-length(x4[,1])])/2-mean(x4))^2,x4[,2])-1/12*weighted.mean(diff(x4[,1])^2,x4[,2]))^4,
(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^4,x5[,2])
-10^2/2*weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])
+7/240*10^4)/sqrt(weighted.mean(((x5[,1][-1]+x5[,1][-length(x5[,1])])/2-mean(x5))^2,x5[,2])-1/12*weighted.mean(diff(x5[,1])^2,x5[,2]))^4
)
)
rownames(tab6) <- colnames(Europe[,-1])
round(tab6,digits=2)
#> asyFis asyPea appFis appPea
#> Partiel_Ens 1.61 2.60 3.06 6.06
#> Partiel_H 2.40 5.75 2.23 5.23
#> Partiel_F 1.00 1.01 0.69 3.69
#> Salariés -1.67 2.77 -3.14 -0.14
#> NonSalariés 2.85 8.10 11.58 14.58